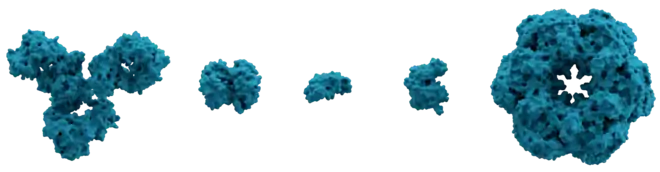
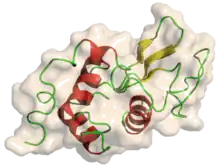
Protéine
Les protéines sont des macromolécules biologiques présentes dans toutes les cellules vivantes. Ce sont des polymères, formées d'une ou de plusieurs chaînes polypeptidiques. Chacune de ces chaînes est constituée de l'enchaînement de résidus d'acides aminés liés entre eux par des liaisons peptidiques.
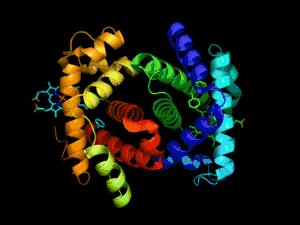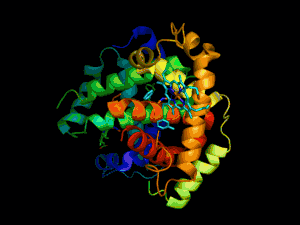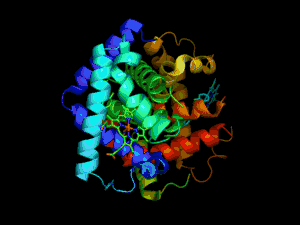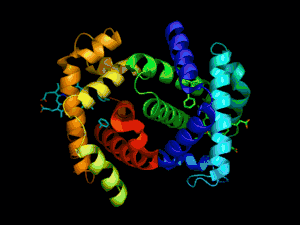
Les protéines assurent une multitude de fonctions au sein de la cellule vivante et dans les tissus. Ce sont des protéines enzymatiques (enzymes) qui catalysent les réactions chimiques de synthèse et de dégradation nécessaires au métabolisme de la cellule. D'autres protéines assurent un rôle structurel au sein du cytosquelette ou des tissus (actine, collagène), certaines sont des moteurs moléculaires qui permettent la mobilité (myosine), d'autres sont impliquées dans le conditionnement de l'ADN (histones), la régulation de l'expression génétique (facteurs de transcription), le métabolisme énergétique (ATP synthase) ou encore la transmission de signaux cellulaires (récepteurs membranaires).
Les chaînes protéiques sont synthétisées dans la cellule au niveau du ribosome, à partir de l'information codée dans les gènes qui détermine l'ordre dans lequel s'enchaînent les 22 acides aminés, dits « protéinogènes », qui sont incorporés directement lors de la biosynthèse des protéines. La succession des acides aminés est appelée « séquence » du polypeptide. Des modifications post-traductionnelles peuvent intervenir ensuite, une fois la protéine synthétisée, ce qui peut avoir pour effet d'en modifier les propriétés physiques ou chimiques. Il est également fréquent que des molécules non protéiques, appelées « groupes prosthétiques », se fixent de manière stable sur des protéines et contribuent de manière déterminante à leurs fonctions biologiques : c'est par exemple le cas de l'hème dans l'hémoglobine, sans lequel cette protéine ne pourrait pas transporter l'oxygène dans le sang.
Les protéines adoptent une structure tridimensionnelle qui leur permet d'assurer leur fonction biologique. Cette structure particulière est déterminée avant tout par leur séquence en acides aminés dont les propriétés physico-chimiques diverses conduit la chaîne protéique à adopter un repliement stable.
Au laboratoire, elles peuvent être séparées des autres constituants cellulaires à l'aide de diverses techniques telles que l'ultracentrifugation, la précipitation, l'électrophorèse et la chromatographie. Le génie génétique a introduit un grand nombre de méthodes permettant de faciliter la purification des protéines. Leur structure peut être étudiée par immunohistochimie, par mutagenèse dirigée, par cristallographie aux rayons X, par résonance magnétique nucléaire et par spectrométrie de masse.
Les protéines sont un composant important de l'alimentation animale, elles sont dégradées dans le tube digestif et les acides aminés libérés sont absorbés au niveau de l'intestin grêle pour ensuite être réutilisés par l'organisme.
Étymologie

Le terme protéine vient du grec ancien prôtos qui signifie premier, essentiel, plus précisément de πρωτει ̃ος / proteios (« qui occupe le premier rang, premier »), auquel vient s'adjoindre le suffixe -ine qui permet de former des substantifs féminin dans le vocabulaire scientifique, et particulièrement en chimie[1] - [2].
Cette étymologie fait probablement référence au fait que les protéines sont indispensables à la vie et qu'elles constituent souvent la part majoritaire (environ 60 %) du poids sec des cellules (animales). Une autre théorie voudrait que protéine fasse référence, comme l'adjectif protéiforme, au dieu grec Protée, qui pouvait changer de forme à volonté. Les protéines adoptent en effet de multiples formes et assurent de multiples fonctions. Toutefois, cette caractéristique ne fut mise en évidence bien plus tard, au cours du XXe siècle.
Histoire de la découverte
Les protéines furent découvertes à partir de 1835 aux Pays-Bas par le chimiste organicien Gerardus Johannes Mulder[3] (1802-1880), sous le nom de wortelstof. C'est son confrère suédois Jöns Jacob Berzelius qui lui suggéra en 1838 le nom de protéine.
Biochimie
Les protéines sont formées d'une ou plusieurs chaînes polypeptidiques, qui sont des biopolymères linéaires pouvant être très longs, composés d'acides L-α-aminés dont il existe une vingtaine de variétés. On parle généralement de protéine au-delà d'une cinquantaine de résidus dans la molécule[4] et de peptide jusqu'à quelques dizaines de résidus.
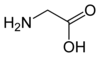
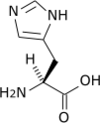
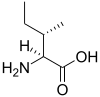
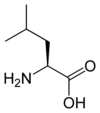
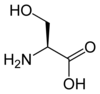
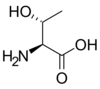
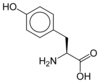
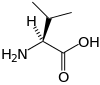
Tous les acides aminés protéinogènes — à l'exception de la proline — partagent une structure commune, constituée d'une fonction acide carboxylique, d'une amine primaire sur le carbone α, et d'une chaîne latérale. Cette dernière présente une très grande variété de structures chimiques, et c'est l'effet combiné de toutes ces chaînes latérales d'une chaîne polypeptidique qui détermine la structure tridimensionnelle ainsi que les propriétés chimiques de cette dernière[5]. La planche ci-dessous présente la structure chimique des 22 acides aminés protéinogènes :
| Structure des 22 acides aminés protéinogènes. La pyrrolysine et la sélénocystéine (ci-dessus grisées) sont spécifiques à certaines protéines : - la pyrrolysine ne se rencontre que chez certaines archées méthanogènes, - la sélénocystéine est présente également chez les eucaryotes mais a priori dans quelques dizaines d'enzymes de la famille des oxydoréductases. Les 20 autres acides aminés, dits standards, sont en revanche universellement distribués chez tous les êtres vivants connus. |
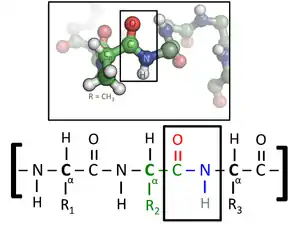
Les acides aminés d'une chaîne polypeptidique sont liés entre eux par des liaisons peptidiques qui s'établissent entre le carboxyle –COOH d'un premier acide aminé et l'amine primaire –NH2 d'un second :

Le squelette de la protéine est ainsi constitué d'un enchaînement linéaire d'acides aminés sur lequel sont branchées les chaînes latérales et reliées par des liaisons peptidiques. La liaison peptidique présente deux formes de résonance qui lui confèrent en partie les propriétés d'une double liaison, ce qui limite les rotations autour de son axe, de sorte que les quatre atomes du groupement amide -(C=O)NH- sont toujours à peu près coplanaires. Les deux autres liaisons constituant le squelette de l'acide aminé peuvent en revanche tourner librement. Les deux angles dièdres correspondant à ces deux liaisons internes déterminent la géométrie locale adoptée par la chaîne protéique.
L'extrémité de la chaîne polypeptidique côté carboxyle est appelée extrémité C-terminale, tandis que celle côté amine est appelée extrémité N-terminale. Les mots protéine, polypeptide et peptide sont assez ambigus et leur sens peut se recouvrir. On parle généralement de protéine en référence à la molécule biologique complète dotée d'une conformation stable, tandis qu'un peptide désigne généralement une molécule plus courte dépourvue de structure tridimensionnelle stable. La limite entre les deux est très imprécise et se situe autour de quelques dizaines de résidus d'acides aminés[6].
Tailles
Les protéines étaient censées toujours être de grande taille (aux échelles biomoléculaires) ; on sait dès le début des années 1990 que ce n'est pas le cas, à la suite de la découverte d'une, puis de quelques autres microprotéines (parfois dénommées MiPs). Depuis, les scientifiques ont mis en évidence l'existence de centaines puis de milliers de microprotéines et de nanoprotéines (n'associant parfois que quelques acides aminés, peut-être auto-assemblés[7]), si petites que les systèmes classiques d'analyse génomique ne les repéraient pas[8]. Elles semblent avoir des rôles-clés au sein des cellules au sein du complexe protéique, en interagissant dans les relations protéines-protéines. Certaines contrôlent ainsi l’activité de protéines plus grosses, jouant un rôle de régulateurs post-traductionnels, sans interagir directement avec l'ADN ou l'ARN[9]. D'autres promeuvent le développement musculaire et régulent la contraction musculaire. D'autres encore contribuent à la gestion des déchets intracellulaires (ARN ancien, dégradé ou défectueux)[8]. Chez les plantes[10] - [11], elles pourraient participer à la détection de la lumière et dans d'autres cas jouer un rôle dans la signalisation phytohormonale. Chez l'animal, elles participeraient au fonctionnement de l'horloge biologique.
On en trouve notamment dans les venins (d'araignées, de scorpions et d'autres animaux venimeux)[8]. Des nanoprotéines complexes peuvent être créées in vitro par autoassemblage d'acides aminés ; elles pourraient peut-être être utilisées pour la reconnaissance et à la catalyse biomoléculaires[7]. On leur a déjà trouvé un intérêt commercial : certains insecticides en utilisent[8]. Elles présentent un intérêt médical : on s'en sert pour marquer des tumeurs cérébrales afin de permettre une chirurgie plus précise[8].
Structure
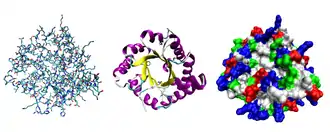
À gauche : représentation de tous les atomes et de leurs liaisons, chaque élément chimique étant représenté par une couleur différente.
Au centre : représentation de la conformation du squelette polypeptidique colorée par structure secondaire.
À droite : surface moléculaire en contact avec le solvant colorée par type de résidu (acide en rouge, basique en bleu, polaire en vert, apolaire en blanc).
La nature des protéines est déterminée avant tout par leur séquence en acides aminés, qui constitue leur structure primaire. Les acides aminés ayant des propriétés chimiques très diverses, leur disposition le long de la chaîne polypeptidique détermine leur arrangement spatial. Celui-ci est décrit localement par leur structure secondaire, stabilisée par des liaisons hydrogène entre résidus d'acides aminés voisins, et globalement par leur structure tertiaire, stabilisée par l'ensemble des interactions entre les résidus — parfois très éloignés sur la séquence peptidique mais mis en contact spatialement par le repliement de la protéine — ainsi qu'entre la protéine elle-même et son environnement. Enfin, l'assemblage de plusieurs sous-unités protéiques pour former un complexe fonctionnel est décrit par la structure quaternaire de cet ensemble.
Il peut aussi se former des liaisons covalentes supplémentaires, soit au sein d'une même chaîne protéique, soit entre différentes chaînes peptidiques au sein d'une protéine, notamment au travers de la formation de ponts disulfure entre résidus de cystéine.
La plupart des protéines adoptent une conformation tridimensionnelle unique. La forme naturelle d'une protéine in vivo est son état natif, qui correspond à la forme qu'elle prend pour être biologiquement active et fonctionnelle. De nombreuses protéines prennent par elles-mêmes leur forme biologiquement active sous l'effet de la distribution spatiale des résidus d'acides aminés qui les constituent, d'autres ont besoin d'être assistées pour ce faire par des protéines chaperonnes pour être repliées selon leur état natif.
Niveaux d'organisation
En biochimie, on peut donc distinguer quatre niveaux d'organisation pour décrire la structure des protéines :
- La structure primaire correspond à la séquence en acides aminés.
- La structure secondaire décrit l'arrangement des résidus d'acides aminés observable à l'échelle atomique. Stabilisés par des liaisons hydrogène, ces arrangements locaux sont par exemple les hélices α, les feuillets β, les tonneaux β, ou les coudes. Il en existe plusieurs variétés, et il est courant qu'une protéine possède globalement plusieurs types de structures secondaires.
- La structure tertiaire correspond à la forme générale de la protéine observable à l'échelle de la molécule tout entière. Elle décrit les interactions entre les différents éléments de la structure secondaire. Elle est stabilisée par tout un ensemble d'interactions conduisant le plus souvent à la formation d'un cœur hydrophobe, avec éventuellement des liaisons salines, des liaisons hydrogène, des ponts disulfure, voire des modifications post-traductionnelles. On désigne souvent par structure tertiaire le repliement d'une protéine.
- La structure quaternaire décrit le complexe résultant de l'assemblage de plusieurs molécules de protéines (plusieurs chaînes polypeptidiques), appelées dans ce cas sous-unités protéiques, pour former un complexe protéique unique. Toutes les protéines ne sont pas nécessairement constituées de plusieurs sous-unités et ne possèdent par conséquent pas toujours de structure quaternaire.
Les protéines ne sont pas des molécules entièrement rigides. Elles sont susceptibles d'adopter plusieurs conformations apparentées en réalisant leurs fonctions biologiques. La transition d'une de ces conformations à une autre est appelée changement conformationnel. Dans le cas d'une enzyme par exemple, de tels changements conformationnels peuvent être induits par l'interaction avec le substrat au niveau du site actif. En solution, les protéines subissent également de nombreux changements conformationnels en raison de la vibration thermique de la collision avec d'autres molécules.
Implications biologiques et détermination des structures tertiaire et quaternaire
On peut distinguer trois grands groupes de protéines en fonction de leur structure tertiaire ou quaternaire : les protéines globulaires, les protéines fibreuses et les protéines membranaires. Presque toutes les protéines globulaires sont solubles et ce sont souvent des enzymes. Les protéines fibreuses jouent souvent un rôle structurel, à l'instar du collagène, constituant principal des tissus conjonctifs, ou de la kératine, constituant protéique des poils et des ongles. Les protéines membranaires sont souvent des récepteurs ou des canaux permettant aux molécules polaires ou électriquement chargées de traverser la membrane.
La connaissance de la structure tertiaire, voire quaternaire, d'une protéine peut fournir des éléments importants pour comprendre comment cette protéine remplit sa fonction biologique. La cristallographie aux rayons X et la spectroscopie RMN sont des méthodes expérimentales courantes pour étudier la structure des protéines, qui peuvent l'une et l'autre fournir des informations avec une résolution à l'échelle atomique. Les données RMN permettent d'obtenir des informations à partir desquelles il est possible d'estimer un sous-ensemble de distances entre certaines paires d'atomes, ce qui permet d'en déduire les conformations possible de cette molécule. L'interférométrie par double polarisation est une méthode analytique quantitative permettant de mesurer la conformation globale de la protéine ainsi que ses changements conformationnels en fonction de son interaction avec d'autres stimulus. Le dichroïsme circulaire fournit une autre technique de laboratoire permettant de résoudre certains éléments de la structure secondaire des protéines (hélices α et feuillets β notamment). La cryo-microscopie électronique permet d'obtenir des informations structurelles à plus faible résolution sur les très grosses protéines, notamment les virus. La cristallographie électronique (en), technique issue de la précédente, permet dans certains cas de produire également des données à haute résolution, notamment pour les cristaux bidimensionnels de protéines membranaires[19]. Les structures protéiques résolues sont généralement déposées dans la Protein Data Bank (PDB), une base de données en accès libre donnant la structure d'un millier de protéines pour laquelle les coordonnées cartésiennes de chaque atome sont disponibles[20].
Le nombre de protéines dont la structure a été résolue est bien plus faible que le nombre de gènes dont la séquence est connue. De plus, le sous-ensemble de protéines dont la structure a été résolue est biaisé en faveur des protéines qui peuvent être aisément préparées en vue d'une analyse par cristallographie aux rayons X, l'une des principales méthodes de détermination des structures protéiques. En particulier, les protéines globulaires sont comparativement les plus faciles à cristalliser en vue d'une cristallographie, tandis que les protéines membranaires sont plus difficiles à cristalliser et sont sous-représentées parmi les protéines disponibles dans la PDB[21]. Pour remédier à cette situation, des démarches de génomique structurale ont été entreprises afin de résoudre les structures représentatives des principales classes de repliement des protéines. Les méthodes de prédiction de la structure des protéines visent à fournir le moyen de générer la structure plausible d'une protéine à partir des structures qui ont pu être déterminées expérimentalement[22].
Synthèse
Les acides α-aminés protéinogènes sont assemblés en polypeptides au sein des cellules par les ribosomes à partir de l'information génétique transmise par les ARN messagers depuis l'ADN constituant les gènes. C'est la séquence nucléotidique de l'ADN, transcrite à l'identique dans l'ARN messager, qui porte l'information lue par les ribosomes pour produire les protéines selon la séquence peptidique spécifiée par les gènes. La correspondance entre la séquence nucléotidique de l'ADN et de l'ARN messager d'une part et la séquence peptidique des protéines synthétisées d'autre part est déterminée par le code génétique, qui est essentiellement le même pour tous les êtres vivants connus hormis un certain nombre de variantes assez limitées.
Code génétique

Le code génétique établit la correspondance entre un triplet de bases nucléiques, appelé codon, sur l'ARN messager et un acide α-aminé protéinogène. Cette correspondance est réalisée in vivo par les ARN de transfert, qui sont des ARN comptant une centaine de nucléotides tout au plus et portant un acide aminé esterifiant leur extrémité 3’-OH. Chacun des acides aminés est lié à des ARN de transfert spécifiques, portant des codons eux aussi spécifiques, de sorte que chacun des 64 codons possibles ne peut coder qu'un seul acide aminé. En revanche, chacun des 22 acides aminés protéinogènes peut être codé par plusieurs codons différents. Ce sont les enzymes réalisant l'estérification des ARN messagers avec les acides aminés — les aminoacyl-ARNt synthétases — qui maintiennent le code génétique : en effet, ces enzymes se lient spécifiquement à la fois à un ARN de transfert donné et à un acide aminé donné, de sorte que chaque type d'ARN de transfert n'est estérifié que par un acide aminé spécifique.
Le cas de la sélénocystéine et de la pyrrolysine est quelque peu différent en ce que ces acides aminés particuliers ne sont pas codés directement par des codons spécifiques mais par recodage traductionnel de codons stop en présence de séquences d'insertions particulières appelées respectivement élément SECIS et élément PYLIS, qui recodent les codons stop UGA (Opale) et UAG (Ambre) respectivement en sélénocystéine et en pyrrolysine. De surcroît, la sélénocystéine n'est pas liée telle quelle à son ARN de transfert, car elle est trop réactive pour exister librement dans la cellule ; c'est la sérine qui est liée à un ARN de transfert de sélénocystéine ARNtSec par la sérine-ARNt ligase. Le séryl-ARNtSec ne peut être utilisé par les ribosomes car il n'est pas reconnu par les facteurs d'élongation intervenant au cours de la biosynthèse des protéines, de sorte que la sérine ne peut être incorporée dans les sélénoprotéines à la place de la sélénocystéine. En revanche, le séryl-ARNtSec est un substrat pour certaines enzymes qui assurent sa conversion en sélénocystéinyl-ARNtSec : conversion directe par la sélénocystéine synthase[23] chez les bactéries, conversion indirecte via l'O-phosphoséryl-ARNtSec successivement par la O-phosphoséryl-ARNtSec kinase[24] et la O-phosphoséryl-ARNt:sélénocystéinyl-ARNt synthase[25] chez les archées et les eucaryotes.
Les gènes codés dans l'ADN sont tout d'abord transcrits en ARN pré-messager par des enzymes telles que les ARN polymérases. La plupart des êtres vivants modifient cet ARN pré-messager à travers un ensemble de processus appelés modifications post-transcriptionnelles conduisant à l'ARN messager mature. Ce dernier est alors utilisable par les ribosomes pour servir de modèle lors de la biosynthèse des protéines. Chez les procaryotes, l'ARN messager peut être utilisé dès qu'il est synthétisé ou être traduit en protéines après avoir quitté le nucléoïde. En revanche, chez les eucaryotes, l'ARN messager est produit dans le noyau de la cellule tandis que les protéines sont synthétisées dans le cytoplasme, de sorte que l'ARN messager doit traverser la membrane nucléaire.
Biosynthèse

La biosynthèse d'une protéine à partir d'un ARN messager est la traduction de cet ARNm. L'ARN messager se lie au ribosome, qui le lit séquentiellement à raison de trois nucléotides à chaque étape de la synthèse. Chaque triplet de nucléotides constitue un codon sur l'ARN messager, auquel peut se lier l'anticodon d'un ARN de transfert apportant l'acide aminé correspondant. L'appariement entre le codon et l'anticodon repose sur la complémentarité de leurs séquences respectives. C'est cette complémentarité qui assure la reconnaissance entre l'ARN de transfert et le codon de l'ARN messager. L'acide aminé apporté par l'ARN de transfert sur le ribosome établit une liaison peptidique avec l'extrémité C-terminale de la chaîne naissante, ce qui permet de l'allonger d'un résidu d'acide aminé. Le ribosome se déplace alors de trois nucléotides sur l'ARN messager pour faire face à un nouveau codon, qui suit exactement le codon précédent. Ce processus se répète jusqu'à ce que le ribosome soit en face d'un codon stop, auquel cas la traduction s'arrête.
La biosynthèse d'une protéine s'effectue ainsi résidu après résidu, de l'extrémité N-terminale vers l'extrémité C-terminale. Une fois synthétisée, la protéine peut subir diverses modifications post-traductionnelles telles que clivage, phosphorylation, acétylation, amidation, méthylation, glycosylation, lipidation, voire la formation de ponts disulfure. La taille des protéines ainsi synthétisées est très variable. Cette taille peut être exprimée en nombre de résidus d'acides aminés constituant ces protéines, ainsi qu'en daltons (symbole Da), qui correspondent en biologie moléculaire à l'unité de masse atomique. Les protéines étant souvent des molécules assez grosses, leur masse est souvent exprimée en kilodaltons (symbole kDa). À titre d'exemple, les protéines de levure ont une longueur moyenne de 466 résidus d'acides aminés, pour une masse de 53 kDa. Les plus grosses protéines connues sont les titines des sarcomères formant les myofibrilles des muscles striés squelettiques[26] : la titine de souris contient quelque 35 213 résidus d'acides aminés formés de 551 739 atomes pour une masse de plus de 3 900 kDa et une longueur de l'ordre de 1 µm[27].
Synthèse chimique
Les petites protéines peuvent également être synthétisées in vitro par un ensemble de méthodes appelées synthèse peptidique, qui reposent sur des techniques de synthèse organique telles que la ligature chimique (en) pour produire efficacement des peptides[28]. La synthèse chimique permet d'introduire des acides aminés non naturels dans la chaîne polypeptidique, en posant par exemple des sondes fluorescentes sur la chaîne latérale de certains d'entre eux[29]. Ces méthodes sont utiles au laboratoire en biochimie et en biologie cellulaire mais ne sont généralement pas employées pour des applications commerciales. La synthèse chimique n'est pas efficace pour synthétiser des peptides de plus de 300 résidus d'acides aminés environ, et les protéines ainsi produites peuvent ne pas adopter facilement leur structure tertiaire native. La plupart des méthodes de synthèse chimique des protéines procèdent de l'extrémité C-terminale vers l'extrémité N-terminale, c'est-à-dire dans le sens inverse de la biosynthèse des protéines par les ribosomes[30].
Fonctions
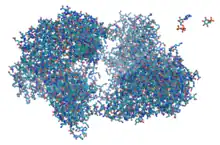
Parmi tous les constituants de la cellule, les protéines sont les éléments les plus actifs. Hormis certains ARN, la plupart des autres molécules biologiques sont chimiquement assez peu réactives et ce sont les protéines qui agissent sur elles. Les protéines constituent environ la moitié de la matière sèche d'une cellule d'E. coli tandis que l'ARN et l'ADN en constituent respectivement un cinquième et 3 %. L'ensemble des protéines exprimées dans une cellule constitue son protéome.
La caractéristique principale des protéines qui leur permet de réaliser leurs fonctions biologiques est leur faculté de se lier à d'autres molécules de façon à la fois très spécifique et très étroite. La région d'une protéine permettant de se lier à une autre molécule est son site de liaison, qui forme souvent une dépression, une cavité, ou « poche », dans la surface de la molécule. C'est la structure tertiaire de la protéine et la nature chimique des chaînes latérales des résidus d'acides aminés du site de liaison qui déterminent la spécificité de cette interaction. Les sites de liaison peuvent conduire à des liaisons particulièrement spécifiques et étroites : ainsi, l'inhibiteur de ribonucléase se lie à l'angiogénine humaine avec une constante de dissociation sub-femtomolaire (< 10−15 mol/L) mais ne se lie pas du tout à la ranpirnase, homologue d'amphibien de cette protéine (constante supérieure à 1 mol/L). Une légère modification chimique peut radicalement modifier la faculté d'une molécule à interagir avec une protéine donnée. Ainsi l'aminoacyl-ARNt synthétase spécifique de la valine se lie à cette dernière sans interagir avec l'isoleucine, qui lui est pourtant structurellement très proche[31].
Les protéines peuvent se lier selon les cas à d'autres protéines ou à de petites molécules comme substrats. Lorsqu'elles se lient spécifiquement à d'autres protéines identiques à elles-mêmes, elles peuvent polymériser pour former des fibrilles. Ceci est fréquent pour les protéines structurelles, formées de monomères globulaires qui s'auto-assemblent pour former des fibres rigides. Des interactions protéine-protéine régulent également leur activité enzymatique, l'avancement du cycle cellulaire et l'assemblage de grands complexes protéiques réalisant des réactions étroitement apparentées partageant une fonction biologique commune. Les protéines peuvent également se lier à la surface des membranes cellulaires et même fréquemment en faire partie intégrante. La capacité de certaines protéines à changer de conformation lorsqu'elles se lient à des molécules spécifiques permet de construire des réseaux de signalisation cellulaire extrêmement complexes. D'une manière générale, l'étude des interactions entre protéines spécifiques est un élément clé de notre compréhension du fonctionnement des cellules et de leur faculté à échanger de l'information[32] - [33].
Enzymes
Le rôle le plus visible des protéines dans la cellule est celui d'enzyme, c'est-à-dire de biomolécule catalysant des réactions chimiques. Les enzymes sont généralement très spécifiques et n'accélèrent qu'une ou quelques réactions chimiques. La très grande majorité des réactions chimiques du métabolisme sont réalisées par des enzymes. Outre le métabolisme, ces dernières interviennent également dans l'expression génétique, la réplication de l'ADN, la réparation de l'ADN, la transcription de l'ADN en ARN, et la traduction de l'ARN messager en protéines. Certaines enzymes agissent sur d'autres protéines pour y lier ou en cliver certains groupes fonctionnels et des résidus d'autres biomolécules, selon un processus appelé modification post-traductionnelle. Les enzymes catalysent plus de 5 000 réactions chimiques différentes[34]. Comme tous les catalyseurs, elles ne modifient pas les équilibres chimiques mais accélèrent les réactions, parfois dans des proportions considérables ; ainsi, l'orotidine-5'-phosphate décarboxylase catalyse en quelques millisecondes une réaction qui prendrait sinon plusieurs millions d'années[35] - [36].
Les molécules qui se lient aux enzymes et sont modifiées chimiquement par elles sont appelées substrats. Bien que les enzymes soient parfois constituées de plusieurs centaines de résidus d'acides aminés, seuls quelques-uns d'entre eux entrent en contact avec le ou les substrats de l'enzyme, et un très petit nombre — généralement trois ou quatre — sont impliqués directement dans la catalyse. On appelle site actif la région d'une enzyme impliquée dans la réaction chimique catalysée par cette protéine : il regroupe les résidus qui se lient au substrat ou contribuent à son positionnement, ainsi que les résidus qui catalysent directement la réaction.
Signalisation cellulaire et liaison de ligands
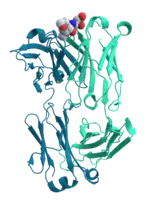
De nombreuses protéines sont impliquées dans les mécanismes de signalisation cellulaire et de transduction de signal. Certaines protéines telles que l'insuline appartiennent au milieu extracellulaire et transmettent un signal de la cellule où elles sont synthétisées vers d'autre cellules parfois situées dans des tissus éloignés. D'autres sont des protéines membranaires qui agissent comme récepteurs dont la fonction principale est de se lier aux molécules porteuses de signaux et d'induire une réponse biochimique dans la cellule cible. De nombreux récepteurs membranaires ont un site de liaison exposé à l'extérieur de la cellule et un domaine effecteur (en) en contact avec le milieu intracellulaire. Ce domaine effecteur peut être porteur d'une activité enzymatique ou peut subir des changements conformationnels agissant sur d'autres protéines intracellulaires.
Les anticorps sont les constituants protéiques du système immunitaire dont la fonction principale est de se lier aux antigènes ou aux xénobiotiques afin de les marquer pour élimination par l'organisme. Les anticorps peuvent être sécrétés dans le milieu extracellulaire ou bien ancrés dans la membrane plasmique de lymphocytes B spécialisés appelés plasmocytes. Là où les enzymes sont très spécifiques de leurs substrats afin d'accélérer des réactions chimiques très précises, les anticorps n'ont pas cette contrainte ; en revanche, leur affinité pour leur cible est extrêmement élevée.
De nombreuses protéines transporteuses de ligands se lient spécifiquement à de petites molécules et les transportent à destination à travers les cellules et les tissus des organismes multicellulaires. Ces protéines doivent posséder une forte affinité pour leur ligand lorsque la concentration de celui-ci est élevée, mais doivent également pouvoir le libérer lorsque sa concentration est faible dans les tissus cibles. L'exemple canonique de la protéine porteuse de ligand est l'hémoglobine, qui transporte l'oxygène des poumons vers les autres organes et tissus chez tous les vertébrés et a des homologues apparentés dans tous les règnes du vivant. Les lectines sont des protéines qui se lient réversiblement à certains glucides avec une très grande spécificité. Elles jouent un rôle dans les phénomènes de reconnaissance biologique impliquant cellules et protéines[37].
Les protéines transmembranaires peuvent également jouer le rôle de protéines transporteuses de ligands susceptibles de modifier la perméabilité de la membrane plasmique aux petites molécules polaires et aux ions. La membrane elle-même possède un cœur hydrophobe à travers lequel les molécules polaires ou électriquement chargées ne peuvent pas diffuser. Les protéines membranaires peuvent ainsi contenir un ou plusieurs canaux à travers la membrane cellulaire et permettant à ces molécules et à ces ions de la traverser. De nombreux canaux ioniques sont très spécifiques de l'ion dont ils permettent la circulation. Ainsi, les canaux potassiques et les canaux sodiques sont souvent spécifiques de l'un des deux ions potassium et sodium à l'exclusion de l'autre.
Protéines structurelles
Les protéines structurelles confèrent raideur et rigidité à des constituants biologiques qui, sans elles, seraient fluides. La plupart des protéines structurelles sont fibreuses. C'est par exemple le cas du collagène et de l'élastine qui sont des constituants essentiels de tissus conjonctifs tels que le cartilage, et de la kératine présente dans les structures dures ou filamenteuses telles que les poils, les ongles, les plumes, les sabots et l'exosquelette de certains animaux. Certaines protéines globulaires peuvent également jouer un rôle structurel, par exemple l'actine et la tubuline dont les monomères sont globulaires et solubles mais polymérisent pour former de longs filaments rigides constituant le cytosquelette, ce qui permet à la cellule de maintenir sa forme et sa taille.
Les protéines motrices sont des protéines structurelles particulières qui sont capables de générer des forces mécaniques. Ce sont par exemple la myosine, la kinésine et la dynéine. Ces protéines sont essentielles à la motilité des organismes unicellulaires ainsi qu'aux spermatozoïdes des organismes multicellulaires. Elles permettent également de générer les forces à l'œuvre dans la contraction musculaire et jouent un rôle essentiel dans le transport intracellulaire.
Les mannoprotéines semblent pourtant avoir des rôles-clé au sein des cellules, notamment en y contrôlant la porosité de la paroi cellulaire[38] - [39] - [40].
Récapitulatif de fonctions assurées par les protéines
Les protéines remplissent ainsi des fonctions très diverses au sein de la cellule et de l'organisme[41] :
- les protéines structurelles, qui permettent à la cellule de maintenir son organisation dans l'espace, et qui sont les constituants du cytosquelette ;
- les protéines de transport, qui assurent le transfert des différentes molécules dans et en dehors des cellules ;
- les protéines régulatrices, qui modulent l'activité d'autres protéines ou qui contrôlent l'expression des gènes ;
- les protéines de signalisation, qui captent les signaux extérieurs, et assurent leur transmission dans la cellule ou l'organisme ; il en existe plusieurs sortes, par exemple les protéines hormonales, qui contribuent à coordonner les activités d'un organisme en agissant comme des signaux entre les cellules ;
- les protéines réceptrices, qui détectent les molécules messagères et les autres signaux pour que la cellule agisse en conséquence :
- les protéines sensorielles détectent les signaux environnementaux (ex. : lumière) et répondent en émettant des signaux dans la cellule,
- les récepteurs d'hormone détectent les hormones et envoient des signaux à la cellule pour qu'elle agisse en conséquence (ex. : l'insuline est une hormone qui, lorsqu'elle est captée, signale à la cellule d'absorber et d'utiliser le glucose) ;
- les protéines motrices, permettant aux cellules ou organismes ou à certains éléments (cils) de se mouvoir ou se déformer (ex. : l'actine et la myosine permettent au muscle de se contracter) ;
- les protéines de défense, qui protègent la cellule contre les agents infectieux (ex. : les anticorps) ;
- les protéines de stockage, qui permettent la mise en réserve d'acides aminés pour pouvoir biosynthétiser d'autres protéines (ex. : l'ovalbumine, la principale protéine du blanc d'œuf permet leur stockage pour le développement des embryons de poulet) ;
- les enzymes, qui modifient la vitesse de presque toutes les réactions chimiques dans la cellule sans être transformées dans la réaction.
Méthodes d'étude
La structure et les fonctions des protéines peuvent être étudiées in vivo, in vitro et in silico. Les études in vivo permettent d'explorer le rôle physiologique d'une protéine au sein d'une cellule vivante ou même au sein d'un organisme dans son ensemble. Les études in vitro de protéines purifiées dans des environnements contrôlés sont utiles pour comprendre la façon dont une protéine fonctionne in vivo : par exemple, l'étude de la cinétique d'une enzyme permet d'analyser le mécanisme chimique de son activité catalytique et de son affinité relative vis-à-vis de différents substrats. Les études in silico utilisent des algorithmes informatiques pour modéliser des protéines.
Purification des protéines
Pour pouvoir être analysée in vitro, une protéine doit préalablement avoir été purifiée des autres constituants chimiques de la cellule. Ceci commence généralement par la lyse de la cellule, au cours de laquelle la membrane plasmique est rompue afin d'en libérer le contenu dans une solution pour donner un lysat. Ce mélange peut être purifié par ultracentrifugation, ce qui permet d'en séparer les constituants en fractions contenant respectivement les protéines solubles, les lipides et protéines membranaires, les organites cellulaires, et les acides nucléiques. La précipitation des protéines par relargage permet de les concentrer à partir de ce lysat. Il est alors possible d'utiliser plusieurs types de chromatographie pour isoler les protéines que l'on souhaite étudier en fonction de leurs propriétés physico-chimiques telles que leur masse molaire, leur charge électrique, ou encore leur affinité de liaison. Le degré de purification peut être suivi à l'aide de plusieurs types d'électrophorèse sur gel si la masse moléculaire et le point isoélectrique des protéines étudiées sont connus, par spectroscopie si la protéine présente des caractéristiques spectroscopiques identifiables, ou par dosage enzymatique (en) si la protéine est porteuse d'une activité enzymatique. Par ailleurs, les protéines peuvent être isolées en fonction de leur charge électrique par focalisation isoélectrique[42].
Les protéines naturelles requièrent éventuellement une série d'étapes de purification avant de pouvoir être étudiées en laboratoire. Afin de simplifier ce procédé, le génie génétique est souvent utilisé pour modifier les protéines en les dotant de caractéristiques qui les rendent plus faciles à purifier sans pour autant altérer leur structure ni leur activité. On ajoute ainsi des « étiquettes » reconnaissables sur les protéines sous forme de séquences d'acides aminés identifiées, souvent une série de résidus d'histidine — étiquette poly-histidine, ou His-tag — à l'extrémité C-terminale ou à l'extrémité N-terminale de la chaîne polypeptidique. De ce fait, lorsque le lysat est placé dans une colonne chromatographique contenant du nickel, les résidus d'histidine se complexent au nickel et restent liées à la colonne tandis que les constituants dépourvus d'étiquette la traversent sans être arrêtés. Plusieurs types d'étiquettes ont été développés afin de permettre aux chercheurs de purifier des protéines particulières à partir de mélanges complexes[43].
Localisation cellulaire
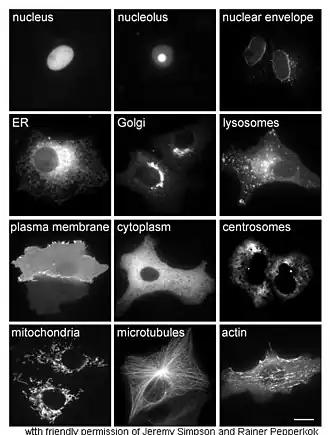
L'étude in vivo des protéines implique souvent de savoir précisément où elles sont synthétisées et où elles se trouvent dans les cellules. Bien que la plupart des protéines intracellulaires soient produites dans le cytoplasme et que la plupart des protéines membranaires ou sécrétées dans le milieu extracellulaire sont produites dans le réticulum endoplasmique, il est rare qu'on comprenne précisément comment les protéines ciblent spécifiquement certaines structures cellulaires ou certains organites. Le génie génétique offre des outils utiles pour se faire une idée de la localisation de certaines protéines, par exemple en liant la protéine étudiée à une protéine permettant de la repérer, c'est-à-dire en réalisant une protéine de fusion entre la protéine étudiée et une protéine utilisée comme marqueur, telle que la protéine fluorescente verte[44]. La localisation intracellulaire de la protéine de fusion résultante peut être facilement et efficacement visualisée par microscopie[45].
D'autres méthodes de localisation intracellulaire des protéines impliquent l'utilisation de marqueurs connus pour certains compartiments cellulaires tels que le réticulum endoplasmique, l'appareil de Golgi, les lysosomes, les mitochondries, les chloroplastes, la membrane plasmique, etc. Il est par exemple possible de localiser des protéines marquées avec une étiquette fluorescente ou ciblées avec des anticorps contre ces marqueurs. Les techniques d'immunofluorescence permettent ainsi de localiser des protéines spécifiques. Des pigments fluorescents sont également utilisés pour marquer des compartiments cellulaires dans un but similaire[46].
L'immunohistochimie utilise généralement un anticorps ciblant une ou plusieurs protéines étudiées qui sont conjugués à des enzymes émettant des signaux luminescents ou chromogènes pouvant être comparés à divers échantillons, ce qui permet d'en déduire des informations sur la localisation des protéines étudiées. Il est également possible d'utiliser des techniques de cofractionnement dans un gradient de saccharose (ou d'une autre substance) à l'aide d'une centrifugation isopycnique.
La microscopie immunoélectronique combine l'utilisation d'une microscopie électronique classique à l'utilisation d'un anticorps dirigé contre la protéine étudiée, cet anticorps étant préalablement conjugué à un matériau à forte densité électronique telle que l'or. Ceci permet de localiser des détails ultrastructurels ainsi que la protéine étudiée[47].
Protéomique
L'ensemble des protéines d'une cellule ou d'un type de cellule constitue son protéome, et la discipline scientifique qui l'étudie est la protéomique. Ces deux termes ont été forgés par analogie avec le génome et la génomique. Si le protéome dérive du génome, il n'est cependant pas possible de prédire exactement quel sera le protéome d'une cellule à partir de la simple connaissance de son génome. En effet, l'expression d'un gène varie d'une cellule à l'autre au sein d'un même organisme en fonction de la différenciation cellulaire, voire dans la même cellule en fonction du cycle cellulaire. Par ailleurs, un même gène peut donner plusieurs protéines (par exemple les polyprotéines virales), et des modifications post-traductionnelles sont souvent nécessaires pour rendre une protéine active.
Parmi les techniques expérimentales utilisées en protéomique, on relève l'électrophorèse bidimensionnelle[48], qui permet la séparation d'un grand nombre de protéines, la spectrométrie de masse[49], qui permet l'identification de protéines rapide et à haut débit ainsi que le séquençage de peptides (le plus souvent après digestion en gel (en)), les puces à protéines (en)[50], qui permettent la détection de concentrations relatives d'un grand nombre de protéines présentes dans une cellule, et l'approche double hybride qui permet également l'exploration des interactions protéine-protéine[51]. L'ensemble des interactions protéine-protéine d'une cellule est appelé interactome[52]. L'approche visant à déterminer la structure des protéines parmi toutes leurs conformations possibles est la génomique structurale[53].
Bio-informatique
Il existe à présent tout un ensemble de méthodes informatiques permettant d'analyser la structure, la fonction et l'évolution des protéines. Le développement de tels outils a été rendu nécessaire par la grande quantité de données génomiques et protéomiques disponibles pour un très grand nombre d'êtres vivants, à commencer par le génome humain. Il est impossible d'étudier toutes les protéines expérimentalement, de sorte que seules un petit nombre d'entre elles font l'objet d'études au laboratoire tandis que les outils de calcul permettent d'extrapoler les résultats ainsi obtenus à d'autres protéines qui leur sont semblables. De telles protéines homologues sont efficacement identifiées par les techniques d'alignement de séquences. Des outils de profilage des séquences peptidiques permettent de localiser les sites clivés par les enzymes de restriction, les cadres de lecture dans les séquences nucléotidiques, et de prédire les structures secondaires. Il est également possible de construire des arbres phylogénétiques et d'élaborer des hypothèses relatives à l'évolution à l'aide de logiciels tels que ClustalW (en) permettant de remonter aux ancêtres des organismes modernes et à leurs gènes. Les outils bio-informatiques sont devenus indispensables à l'étude des gènes et des protéines exprimées par ces gènes.
Prédiction de structure et simulation
En plus de la génomique structurelle, la prédiction de la structure des protéines vise à développer des moyens permettant d'élaborer efficacement des modèles plausibles décrivant la structure de protéines qui n'ont pu être résolues expérimentalement[54]. Le mode de prédiction de structure le plus efficace, appelé modélisation par homologie, se fonde sur l'existence de structures modèles connues dont la séquence présente des similitudes avec celle de la protéine étudiée. Le but de la génomique structurelle est de fournir suffisamment de données sur les structures résolues afin de permettre l'élucidation de celles qui restent à résoudre[55]. Bien qu'il demeure malaisé de modéliser précisément des structures lorsqu'il n'existe que des modèles structurels éloignés auxquels se référer, on pense que le nœud du problème se trouve au niveau de l'alignement des séquences car des modèles très exacts peuvent être établis dès lors qu'un alignement de séquences très exact est connu[56]. De nombreuses prédictions de structures ont été utiles au domaine émergent du génie protéique (en), qui a notamment élaboré de nouveaux modes de repliement[57]. Un problème plus complexe à résoudre par le calcul est la prédiction des interactions intermoléculaires, comme la prédiction de l'ancrage des molécules et des interactions protéine-protéine[58].
Le repliement et la liaison des protéines peuvent être simulés à l'aide de techniques telles que la mécanique moléculaire, la dynamique moléculaire et la méthode de Monte Carlo, qui bénéficient de plus en plus des architectures informatiques parallèles et du calcul distribué, comme le projet Folding@home ou la modélisation moléculaire sur processeur graphique. Le repliement de petits domaines protéiques en hélice α, comme la coiffe de la villine[59] et la protéine accessoire du VIH[60] ont été simulées in silico avec succès, et les méthodes hybrides qui combinent la dynamique moléculaire standard avec des éléments de mécanique quantique ont permis l'exploration des états électroniques des rhodopsines[61].
Phénotype
Le plan de fabrication des protéines dépend donc en premier lieu du gène. Or les séquences des gènes ne sont pas strictement identiques d'un individu à l'autre. De plus, dans le cas des êtres vivants diploïdes, il existe deux exemplaires de chaque gène. Et ces deux exemplaires ne sont pas nécessairement identiques. Un gène existe donc en plusieurs versions d'un individu à l'autre et parfois chez un même individu. Ces différentes versions sont appelées allèles. L'ensemble des allèles d'un individu forme le génotype.
Puisque les gènes existent en plusieurs versions, les protéines vont également exister en différentes versions. Ces différentes versions de protéines vont provoquer des différences d'un individu à l'autre : tel individu aura les yeux bleus mais tel autre aura les yeux noirs, etc. Ces caractéristiques, visibles ou non, propres à chaque individu sont appelées le phénotype. Chez un même individu, un groupe de protéines à séquence similaire et fonction identique est dit isoforme. Les isoformes peuvent être le résultat de l'épissage alternatif d'un même gène, l'expression de plusieurs allèles d'un gène, ou encore la présence de plusieurs gènes homologues dans le génome.
Évolution
Au cours de l'évolution, les accumulations de mutations ont fait diverger les gènes au sein des espèces et entre espèces. De là provient la diversité des protéines qui leur sont associées. On peut toutefois définir des familles de protéines, elles-mêmes correspondant à des familles de gènes. Ainsi, dans une espèce peuvent coexister des gènes, et par conséquent des protéines, très similaires formant une famille. Deux espèces proches ont de fortes chances d'avoir des représentants de même famille de protéines.
On parle d'homologie entre protéines lorsque différentes protéines ont une origine commune, un gène ancestral commun.
La comparaison des séquences de protéines permet de mettre en évidence le degré de « parenté » entre différentes protéines, on parle ici de similarité de séquence. La fonction des protéines peut diverger au fur et à mesure que la similarité diminue, donnant ainsi naissance à des familles de protéines ayant une origine commune mais ayant des fonctions différentes.
L'analyse des séquences et des structures de protéine a permis de constater que beaucoup s'organisaient en domaines, c'est-à-dire en parties acquérant une structure et remplissant une fonction spécifique. L'existence de protéines à plusieurs domaines peut être le résultat de la recombinaison en un gène unique de plusieurs gènes originellement individuels, et réciproquement des protéines composés d'un unique domaine peuvent être le fruit de la séparation en plusieurs gènes d'un gène originellement codant une protéine à plusieurs domaines.
Alimentation humaine
Lors de la digestion, à partir de l'estomac les protéines d'origine végétale, bactérienne, fongique ou animale sont désagrégées (hydrolysées) par des protéases ; découpées en polypeptides et ensuite en acides aminés utiles pour l'organisme, y compris en acides aminés essentiels (que l'organisme ne peut pas synthétiser). Le pepsinogène est converti en pepsine au contact de l'acide chlorhydrique stomacal. La pepsine est la seule enzyme protéolytique qui digère le collagène, la principale protéine du tissu conjonctif.
La digestion des protéines a surtout lieu dans le duodénum. Elles sont principalement absorbées quand elles arrivent dans le jéjunum et seules 1 % des protéines ingérées se retrouvent dans les fèces. Certains acides aminés restent dans les cellules épithéliales de l'intestin, utilisés pour la biosynthèse de nouvelles protéines, y compris des protéines intestinales constamment digérées, recyclées et absorbées par l'intestin grêle.
La digestibilité des protéines varie considérablement selon leur nature et la préparation de l'aliment.
Quantités recommandées
L'ANSES recommande un apport nutritionnel conseillé (ANC) de 0,83 g/kg par jour, pour un maximum de 2,2 g/kg par jour chez l’adulte en bonne santé[62], soit 62 g par jour pour un homme de 75 kg. À noter que les ANC sont supérieurs aux besoins moyens qui sont de 0,66 g/kg par jour selon ce même rapport, ce qui donnerait 49,5 g par jour pour le cas précédent.
Les besoins moyens en protéines ont été définis par la FAO qui recommande 49 g de protéines pour les hommes adultes et 41 g pour les femmes (47 si enceintes, 58,5 si allaitantes)[63].
Protéines animales, fongiques, végétales
Selon l'American Heart Association, il n'est pas nécessaire de consommer des protéines animales pour avoir suffisamment de protéines dans son alimentation : les protéines végétales peuvent fournir suffisamment d'acides aminés essentiels et non essentiels, pourvu que les sources de protéines alimentaires soient variées et que l'apport calorique suffise à répondre aux besoins énergétiques. Il n'est pas nécessaire de les combiner dans un même repas[64]. L'Association américaine de diététique rappelle elle aussi que les protéines végétales peuvent répondre aux exigences en matière de protéines si l'alimentation végétale est variée et répond aux besoins en énergie. De plus, « un assortiment d'aliments végétaux consommés au cours d'une journée peut fournir tous les acides aminés essentiels et assurer une rétention et une utilisation suffisantes de l'azote chez les adultes en bonne santé, de sorte que la combinaison de protéines au cours d'un même repas n'est pas nécessaire[65]. »
Protéines animales
Les protéines animales sont toujours accompagnées de lipides saturés dont la consommation est souvent excessive, et parfois d'additifs alimentaires (tels les nitrites des charcuteries, soupçonnés d'être cancérigènes par le Centre international de recherche sur le cancer (CIRC), et qui provoquent la formation de deux produits cancérogènes avérés en milieu acide, comme c'est le cas dans l'estomac). Les protéines animales, ou des produits associés comme les amines hétérocycliques seraient également un facteur de risque pour certains cancers (côlon, vessie)[66]. Depuis 2015, l'OMS et le CIRC ont classé la viande rouge (porc, boeuf, mouton, cheval et chèvre) cancérigène probable et la viande transformée comme cancérigène avéré (34 000 décès/an dans le monde, selon une étude du Global Burden of Disease Project ; selon l'OMS, manger cinquante grammes de viande transformée par jour augmente le risque de cancer colorectal de 18 % (une viande est dite « transformée » si elle a subi une salaison, maturation, fermentation, fumaison ou d'autres processus visant à améliorer sa saveur ou sa conservation)[67]. Faute de données, le Groupe de travail du CIRC n'a pas pu classer la viande crue vis à vis du risque de cancer, mais il rappelle qu'elle présente un risque infectieux[67]. Les animaux risquent plus d'avoir bioconcentré des polluants via la chaine alimentaire.
Protéines végétales
Les protéines végétales ont des effets positifs associés aux végétaux riches en protéines. Les légumes secs sont riches en protéines, mais aussi en fibres, en minéraux et apportent un sentiment de satiété pour un indice glycémique faible. Consommer des haricots contribue à diminuer le taux de cholestérol[68] - [69] et le risque d'accident cardio-vasculaire[70] et de certains cancers (colorectal, de la prostate, et du pancréas)[66]. Elles sont bien évidemment une alternative pour les végans ou végétariens. Les noix, les légumes, les haricots, le quinoa et les céréales contiennent de grandes quantités de protéines mais aussi d'énergie.
Protéines fongiques
Protéines fongiques : les champignons comestibles sont souvent riches en protéines et, comme les plantes, ils sont sources de fibres alimentaires et de minéraux. Par contre, récoltés dans la nature ou cultivés sur des substrats pollués, ils tendent à fortement accumuler de nombreux métaux lourds, métalloïdes voire des radionucléides.
Qualité des protéines
La totalité des acides aminés nécessaires doit être apportée par la nourriture, sous peine d'être carencé, ce qui implique des sources diversifiés de protéines.
La recommandation de combiner les protéines animale et végétales dans chaque repas est invalidée depuis 1994 à la suite d'un article de Vernon Young et Peter Pellett devenu une référence sur le métabolisme des protéines chez l'homme, confirmant que la combinaison de protéines dans les repas est totalement inutile. Les personnes ne souhaitant pas manger de protéines animales ne risquent pas de déséquilibre d'amino-acides des protéines végétales de leur régime alimentaire[71]. De nombreuses protéines végétales contiennent un peu moins d'un ou de plusieurs des acides aminés essentiels (lysine surtout et moindrement méthionine et thréonine), sans pour autant que la consommation exclusive de sources de protéines végétales empêche d'avoir une alimentation équilibrée en acides aminés essentiels[71].
Les conclusions de l'article de Young et Pellet sont seulement à considérer dans le cas très général où les céréales ne sont pas la source exclusive de l'alimentation, ce qu'ils prennent soin de préciser d'ailleurs, où qu'ils explicitent dans d'autres articles[72]. Ainsi dans certaines régions défavorisées, les rations alimentaires peuvent ne comporter que des céréales, ce qui entraîne de graves problèmes de santé pour les jeunes enfants, par exemple dans les foyers pauvres de l'État du Madhya Pradesh en Inde (blé et riz)[73].
Par ailleurs, des firmes semencières cherchent à obtenir ou ont déjà obtenu des variétés de céréales à contenu en acides aminés modifié (OGM), par exemple des maïs enrichis en lysine[74] - [75].
Les autorités françaises de santé (AFSSA/ANSES) refusent encore de trancher cette question[76].
Compléments alimentaires
Les compléments alimentaires protéinés existent, pour les sportifs souhaitant développer leur volume musculaire, et pour les personnes en carences de protéines. Les protéines utilisées sont souvent des protéines issues de la luzerne (Luzerne sous forme d'extrait foliaire (EFL)) de la féverolle, du pois ou du lactosérum (sous le nom de « whey »), et des acides aminés ramifiés désignés sous le nom de « BCAA ».
Champignons
Les champignons ont des teneurs en protéines difficiles à mesurer avec précision (teneurs autrefois surestimées de 70 à 200 % parfois[77]) mais souvent relativement élevées (15 à 35 % du poids sec du champignon[78] - [79]), beaucoup plus élevées que des céréales telles que le blé et le maïs, d'intérêt alimentaire[80]. Ces teneurs sont comparables à celle de légumineuses telles que pois et lentilles.
Acides aminés essentiels
Les acides aminés essentiels comptent souvent pour une part importante de ces protéines (ex. : 61,8 et 63,3 % des teneurs totales en acides aminés respectivement chez Tricholoma portentosum et Tricholoma terreum (chez lesquels la leucine, l'isoleucine et le tryptophane sont les acides aminés limitants)[81]. Les scores d'acides aminés corrigés (PDCAAS) des protéines de ces deux champignons sont faibles par rapport à ceux de la caséine, du blanc d'œuf et du soja, mais supérieurs à ceux de nombreuses protéines végétales. La teneur en matières grasses était faible (5,7 % pour Tricholoma portentosum et 6,6 % pour Tricholoma terreum) chez les deux espèces, les acides oléique et linoléique représentant plus de 75 % du total des acides gras[81].
Certains comme le champignon de Paris (3,09 g de protéine pour 100 g) sont depuis longtemps cultivés[82] et séchés, mais individuellement (comme d'autres aliments) ils peuvent être déficients en certains acides aminés (ex. : acides aminés soufrés, méthionine et cystine dans le cas des pleurotes par exemple)[83] mais ils sont riches en lysine et leucine qui manquent par exemple dans les céréales[79]. On leur découvre encore des vertus (ex. : l'une de ces protéines semble chez la souris inhiber les allergies alimentaires[84]) et des défauts (ex. : une autre protéine fongique s'est montrée cardiotoxique)[85].
Aliments d'origine animale
Les aliments d'origine animale sont généralement plus protéinés que ceux d'origine végétale, et notamment les œufs (riche en albumine) ou le fromage blanc, fromage (caséine…), comme le parmesan par exemple qui en contient 39,4 g/100 g, plus que la viande et le poisson. Outre certaines viandes (ex. : blanc de poulet cuit avec une teneur moyenne de 29,2 g/100 g[86]) comme le bœuf qui en contient 26 g/100 g, des poissons tels que le thon blanc et rouge, le saumon, le cabillaud, le maquereau ou la sardine en contiennent aussi environ 30 g/100 g. Les œufs sont aussi une source de protéines (24 g/100 g pour quatre œufs[87]).
Aliments végétaux
Certains végétaux ou graines sont très riches en protéines : graines oléagineuses (amande, pistache, lin, etc.) et de légumineuses (pois chiche, haricot, lentille, etc.). Ainsi 100 g de produit brut contiennent une part en protéines de : 58 g pour la spiruline, 38 g pour le soja, 30 g pour les graines de citrouille, 25 g pour le haricot noir, 24 g pour les lentilles, 21 g pour le seitan (gluten) et les noix, 20 g pour les amandes et la semoule, 15 g pour les flocons d'avoine, 15 g pour le riz sauvage, 14 g pour le quinoa[87].
Levures, bactéries et cyanobactéries
Les levures, bactéries et cyanobactéries sont rarement cultivées pour être directement mangées, mais la levure de bière ou la spiruline (58 g de protéines pour 100 g pour la spiruline) sont très riches en protéines.
Notes et références
- « PROTÉINE, subst. fém. », sur cnrtl.fr (consulté le ).
- « -INE, élément formant (v. III) », sur cnrtl.fr/ (consulté le ).
- Découvertes des protéines « http://www.ulysse.u-bordeaux.fr/atelier/ikramer/biocell_diffusion/gbb.cel.fa.106.b3/content/exc01.htm »(Archive.org • Wikiwix • Archive.is • Google • Que faire ?)
- Gregory A. Petsko et Dagmar Ringe (trad. de l'anglais), Structure et fonction des protéines, Bruxelles, De Boek Université, , 190 p. (ISBN 978-2-8041-5888-0, lire en ligne).
- (en) Alex Gutteridge et Janet M. Thornton, « Understanding nature's catalytic toolkit », Trends in Biochemical Sciences, vol. 30, no 11, , p. 622-629 (PMID 16214343, DOI 10.1016/j.tibs.2005.09.006, lire en ligne)
- (en) Harvey Lodish, Arnold Berk, Paul Matsudaira, Chris A. Kaiser, Monty Krieger, Matthew P. Scott, Lawrence Zipursky et James Darnell, Molecular Cell Biology, New York, W. H. Freeman & Company 5e édition, , 973 p. (ISBN 978-0-7167-4366-8)
- Garcia Martin S. (2015), Dynamic Nanoproteins: Self-Assembly of Peptides on Monolayer Protected Gold Nanoparticles.
- Mitch Leslie (2019), Outsize impact, Science, 18 octobre 2019, vol. 366, no 6463, p. 296-299, DOI 10.1126/science.366.6463.296 (résumé).
- Staudt, A. C. et Wenkel, S. (2011), Regulation of protein function by ‘microProteins’, EMBO reports, 12 (1), 35-42.
- (Wang et al (2009)
- zhang et al, 2009)
- (en) Zhaohui Xu, Arthur L. Horwich et Paul B. Sigler, « The crystal structure of the asymmetric GroEL–GroES–(ADP)7 chaperonin complex », Nature, vol. 388, no 6644, , p. 741-750 (PMID 9285585, DOI 10.1038/41944, lire en ligne)
- (en) Lisa J Harris, Eileen Skaletsky et Alexander McPherson, « Crystallographic structure of an intact IgG1 monoclonal antibody », Journal of Molecular Biology, vol. 275, no 5, , p. 861-872 (PMID 9480774, DOI 10.1006/jmbi.1997.1508, lire en ligne)
- (en) W. Bolton et M. F. Perutz, « Three Dimensional Fourier Synthesis of Horse Deoxyhaemoglobin at 2.8 Å Resolution », Nature, vol. 228, no 5271, , p. 551-552 (PMID 5472471, DOI 10.1038/228551a0, Bibcode 1970Natur.228..551B, lire en ligne)
- (en) Edward N. Baker, Thomas L. Blundell, John F. Cutfield, Susan M. Cutfield, Eleanor J. Dodson, Guy G. Dodson, Dorothy M. Crowfoot Hodgkin, Roderick E. Hubbard, Neil W. Isaacs, Colin D. Reynolds, Kiwako Sakabe, Norioshi Sakabe et Numminate M. Vijayan, « The structure of 2Zn pig insulin crystals at 1.5 Å resolution », Philosophical Transactions B, vol. 319, no 1195, , p. 369-456 (PMID 2905485, DOI 10.1098/rstb.1988.0058, Bibcode 1988RSPTB.319..369B, lire en ligne)
- (en) Michael B. Berry et George N. Phillips Jr., « Crystal structures of Bacillus stearothermophilus adenylate kinase with bound Ap5A, Mg2+ Ap5A, and Mn2+ Ap5A reveal an intermediate lid position and six coordinate octahedral geometry for bound Mg2+ and Mn2+ », Proteins: Structure, Function, and Bioinformatics, vol. 32, no 3, , p. 276-288 (PMID 9715904, lire en ligne)
- (en) Harindarpal S. Gill et David Eisenberg, « The Crystal Structure of Phosphinothricin in the Active Site of Glutamine Synthetase Illuminates the Mechanism of Enzymatic Inhibition », Biochemistry, vol. 40, no 7, , p. 1903-1912 (PMID 11329256, DOI 10.1021/bi002438h, lire en ligne)
- (en) Wojciech R. Rypniewski, Hazel M. Holden et Ivan Rayment, « Structural consequences of reductive methylation of lysine residues in hen egg white lysozyme: An x-ray analysis at 1.8-Å resolution », Biochemistry, vol. 32, no 37, , p. 9851-9858 (PMID 8373783, DOI 10.1021/bi00088a041, lire en ligne)
- (en) Tamir Gonen, Yifan Cheng, Piotr Sliz, Yoko Hiroaki, Yoshinori Fujiyoshi, Stephen C. Harrison et Thomas Walz, « Lipid–protein interactions in double-layered two-dimensional AQP0 crystals », Nature, vol. 438, no 7068, , p. 633-638 (PMID 16319884, PMCID 1350984, DOI 10.1038/nature04321, lire en ligne)
- (en) Daron M. Standley, Akira R. Kinjo, Kengo Kinoshita et Haruki Nakamura, « Protein structure databases with new web services for structural biology and biomedical research », Briefings in Bioinformatics, vol. 9, no 4, , p. 276-285 (PMID 18430752, DOI 10.1093/bib/bbn015, lire en ligne)
- (en) Peter Walian, Timothy A Cross et Bing K Jap, « Structural genomics of membrane proteins », Genome Biology, vol. 5, no 4, , p. 215 (PMID 15059248, PMCID 395774, DOI 10.1186/gb-2004-5-4-215, lire en ligne)
- (en) Roy D. Sleator, « Prediction of Protein Functions », Methods in Molecular Biology, vol. 815, , p. 15-24 (PMID 22130980, DOI 10.1007/978-1-61779-424-7_2, lire en ligne)
- K. Forchhammer et A. Bock, « Selenocysteine synthase from Escherichia coli. Analysis of the reaction sequence », J. Biol. Chem., vol. 266, no 10, , p. 6324–8 (PMID 2007585)
- (en) Yuhei Araiso, Sotiria Palioura, Ryuichiro Ishitani, R. Lynn Sherrer, Patrick O’Donoghue, Jing Yuan, Hiroyuki Oshikane, Naoshi Domae, Julian DeFranco, Dieter Söll et Osamu Nureki, « Structural insights into RNA-dependent eukaryal and archaeal selenocysteine formation », Nucleic Acids Research, vol. 36, no 4, , p. 1187-1199 (PMID 18158303, PMCID 2275076, DOI 10.1093/nar/gkm1122, lire en ligne)
- (en) Sotiria Palioura, R. Lynn Sherrer, Thomas A. Steitz, Dieter Söll et Miljan Simonović, « The Human SepSecS-tRNASec Complex Reveals the Mechanism of Selenocysteine Formation », Science, vol. 325, no 5938, , p. 321-325 (PMID 19608919, PMCID 2857584, DOI 10.1126/science.1173755, Bibcode 2009Sci...325..321P, lire en ligne)
- (en) Alice B. Fulton et William B. Isaacs, « Titin, a huge, elastic sarcomeric protein with a probable role in morphogenesis », BioEssays, vol. 13, no 4, , p. 157-161 (PMID 1859393, DOI 10.1002/bies.950130403, lire en ligne)
- (en) « Titin (EC 2.7.11.1) (Connectin) Mus musculus (Mouse) », sur ProtParam sur ExPASy (consulté le )
- (en) Thomas Bruckdorfer, Oleg Marder et Fernando Albericio, « From Production of Peptides in Milligram Amounts for Research to Multi-Tons Quantities for Drugs of the Future », Current Pharmaceutical Biotechnology, vol. 5, no 1, , p. 29-43 (PMID 14965208, DOI 10.2174/1389201043489620, lire en ligne)
- (en) Dirk Schwarzer et Philip A Cole, « Protein semisynthesis and expressed protein ligation: chasing a protein's tail », Current Opinion in Chemical Biology, vol. 9, no 6, , p. 561-569 (PMID 16226484, DOI 10.1016/j.cbpa.2005.09.018, lire en ligne)
- (en) Stephen B. H. Kent, « Total chemical synthesis of proteins », Chemical Society Reviews, vol. 38, no 2, , p. 338-351 (PMID 19169452, DOI 10.1039/B700141J, lire en ligne)
- (en) R. Sankaranarayanan et D. Moras, « The fidelity of the translation of the genetic code », Acta Biochimica Polonica, vol. 48, no 2, , p. 323-335 (PMID 11732604, lire en ligne)
- (en) John A. Copland, Melinda Sheffield-Moore, Nina Koldzic-Zivanovic, Sean Gentry, George Lamprou, Fotini Tzortzatou-Stathopoulou, Vassilis Zoumpourlis, Randall J. Urban et Spiros A. Vlahopoulos, « Sex steroid receptors in skeletal differentiation and epithelial neoplasia: is tissue-specific intervention possible? », BioEssays, vol. 31, no 6, , p. 629-641 (PMID 19382224, DOI 10.1002/bies.200800138, lire en ligne)
- (en) Stanislav Samarin et Asma Nusrat, « Regulation of epithelial apical junctional complex by Rho family GTPases », Frontiers in Bioscience, vol. 14, , p. 1129-1142 (PMID 19273120, DOI 10.2741/3298, lire en ligne)
- (en) Ida Schomburg, Antje Chang, Sandra Placzek, Carola Söhngen, Michael Rother, Maren Lang, Cornelia Munaretto, Susanne Ulas, Michael Stelzer, Andreas Grote, Maurice Scheer et Dietmar Schomburg, « BRENDA in 2013: integrated reactions, kinetic data, enzyme function data, improved disease classification: new options and contents in BRENDA », Nucleic Acids Research, vol. 41, no D1, , D764-D772 (PMID 23203881, DOI 10.1093/nar/gks1049, lire en ligne)
- (en) A. Radzicka et R. Wolfenden, « A proficient enzyme », Science, vol. 267, no 5194, , p. 90-93 (PMID 7809611, DOI 10.1126/science.7809611, lire en ligne)
- (en) Brian P. Callahan et Brian G. Miller, « OMP decarboxylase—An enigma persists », Bioorganic Chemistry, vol. 35, no 6, , p. 465-469 (PMID 17889251, DOI 10.1016/j.bioorg.2007.07.004, lire en ligne)
- (en) Harold Rudiger, Hans-Christian Siebert, Dolores Solis, Jesus initial Jimenez-Barbero, Antonio Romero, Claus-Wilhelm von der Lieth, Teresa Diaz-Maurino et Hans-Joachim Gabius, « Medicinal Chemistry Based on the Sugar Code: Fundamentals of Lectinology and Experimental Strategies with Lectins as Targets », Current Medicinal Chemistry, vol. 7, no 4, , p. 389-416 (PMID 10702616, DOI 10.2174/0929867003375164, lire en ligne)
- De Nobel, J. G., Klis, F. M., Priem, J., Munnik, T. et Van Den Ende, H. (1990), The glucanase‐soluble mannoproteins limit cell wall porosity in Saccharomyces cerevisiae, Yeast, 6 (6), 491-499.
- Zlotnik, H. I. N. D. A., Fernandez, M. P., Bowers, B. L. A. I. R. et Cabib, E. N. R. I. C. O. (1984), Saccharomyces cerevisiae mannoproteins form an external cell wall layer that determines wall porosity, Journal of Bacteriology, 159(3), 1018-1026.
- Caridi A. (2006), Enological functions of parietal yeast mannoproteins, Antonie Van Leeuwenhoek, 89(3-4), 417-422 (résumé).
- Harvey Lodish, Arnold Berk, Paul Matsudaira, Chris A. Kaiser, Monty Krieger, Matthew P. Scott, S. Laurence Zipursky et James Darnell (trad. Pierre L. Masson et Chrystelle Sanlaville), Biologie moléculaire de la cellule [« Molecular Cell Biology »], Bruxelles, De Boeck Université, , 3e éd., 1096 p. [détail de l’édition] (ISBN 2-8041-4802-5)
- (en) Julie Hey, Anton Posch, Andrew Cohen, Ning Liu et Adrianna Harbers, « Fractionation of Complex Protein Mixtures by Liquid-Phase Isoelectric Focusing », Methods in Molecular Biology, vol. 424, , p. 225-239 (PMID 18369866, DOI 10.1007/978-1-60327-064-9_19, lire en ligne)
- (en) K. Terpe, « Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems », Applied Microbiology and Biotechnology, vol. 60, no 5, , p. 523-533 (PMID 12536251, DOI 10.1007/s00253-002-1158-6, lire en ligne)
- (en) Olesya V. Stepanenko, Vladislav V. Verkhusha, Irina M. Kuznetsova, Vladimir N. Uversky and K. K. Turoverov, « Fluorescent Proteins as Biomarkers and Biosensors: Throwing Color Lights on Molecular and Cellular Processes », Current Protein & Peptide Science, vol. 9, no 4, , p. 338-369 (PMID 18691124, PMCID 2904242, DOI 10.2174/138920308785132668, lire en ligne)
- (en) Rafael Yuste, « Fluorescence microscopy today », Nature Methods, vol. 2, no 12, , p. 902-904 (PMID 16299474, DOI 10.1038/nmeth1205-902, lire en ligne)
- (en) William Margolin, « Green Fluorescent Protein as a Reporter for Macromolecular Localization in Bacterial Cells », Methods, vol. 20, no 1, , p. 62-72 (PMID 10610805, DOI 10.1006/meth.1999.0906, lire en ligne)
- (en) Terry M. Mayhew et John M. Lucocq, « Developments in cell biology for quantitative immunoelectron microscopy based on thin sections: a review », Histochemistry and Cell Biology, vol. 130, no 2, , p. 299-313 (PMID 18553098, PMCID 2491712, DOI 10.1007/s00418-008-0451-6, lire en ligne)
- (en) Angelika Görg, Walter Weiss et Michael J Dunn, « Current two-dimensional electrophoresis technology for proteomics », Proteomics, vol. 4, no 12, , p. 3665-3685 (PMID 15543535, DOI 10.1002/pmic.200401031, lire en ligne)
- (en) P. Conrotto et S. Souchelnytskyi, « Proteomic approaches in biological and medical sciences: principles and applications », Experimental Oncology, vol. 30, no 3, , p. 171-180 (PMID 18806738)
- (en) T. Joos et J. Bachmann, « Protein microarrays: potentials and limitations », Frontiers in Bioscience, vol. 14, , p. 4376-4385 (PMID 19273356, DOI 10.2741/3534)
- (en) Manfred Koegl et Peter Uetz, « Improving yeast two-hybrid screening systems », Briefings in Functional Genomics, vol. 6, no 4, (PMID 18218650, DOI 10.1093/bfgp/elm035, bfg.oxfordjournals.org/content/6/4/302.full.pdf+html)
- (en) Dariusz Plewczyński et Krzysztof Ginalski, « The interactome: Predicting the protein-protein interactions in cells », Cellular and Molecular Biology Letters, vol. 14, no 1, , p. 1-22 (PMID 18839074, DOI 10.2478/s11658-008-0024-7, lire en ligne)
- (en) Chao Zhang et Sung-Hou Kim, « Overview of structural genomics: from structure to function », Current Opinion in Chemical Biology, vol. 7, no 1, , p. 28-32 (PMID 12547423, DOI 10.1016/S1367-5931(02)00015-7, lire en ligne)
- (en) Yang Zhang, « Progress and challenges in protein structure prediction », Current Opinion in Structural Biology, vol. 18, no 3, , p. 342-348 (PMID 18436442, PMCID 2680823, DOI 10.1016/j.sbi.2008.02.004, lire en ligne)
- (en) Zhexin Xiang, « Advances in Homology Protein Structure Modeling », Current Protein & Peptide Science, vol. 7, no 3, , p. 217-227 (PMID 16787261, PMCID 1839925, DOI 10.2174/138920306777452312#sthash.hup2vFsH.dpuf, lire en ligne)
- (en) Yang Zhang et Jeffrey Skolnick, « The protein structure prediction problem could be solved using the current PDB library », Proceedings of the National Academy of Sciences of the United States of America, vol. 102, no 4, , p. 1029-1034 (PMID 15653774, PMCID 545829, DOI 10.1073/pnas.0407152101, lire en ligne)
- (en) Brian Kuhlman, Gautam Dantas, Gregory C. Ireton, Gabriele Varani, Barry L. Stoddard et David Baker, « Design of a Novel Globular Protein Fold with Atomic-Level Accuracy », Science, vol. 302, no 5649, , p. 1364-1368 (PMID 14631033, DOI 10.1126/science.1089427, lire en ligne)
- (en) David W. Ritchie, « Recent Progress and Future Directions in Protein-Protein Docking », Current Protein & Peptide Science, vol. 9, no 1, , p. 1-15 (PMID 18336319, DOI 10.2174/138920308783565741, lire en ligne)
- (en) Bojan Zagrovic, Christopher D. Snow, Michael R. Shirts et Vijay S. Pande, « Simulation of Folding of a Small Alpha-helical Protein in Atomistic Detail using Worldwide-distributed Computing », Journal of Molecular Biology, vol. 323, no 5, , p. 927-937 (PMID 12417204, DOI 10.1016/S0022-2836(02)00997-X, lire en ligne)
- (en) T. Herges et W. Wenzel, « In Silico Folding of a Three Helix Protein and Characterization of Its Free-Energy Landscape in an All-Atom Force Field », Physical Reviez Letters, vol. 94, no 1, , p. 018101 (PMID 15698135, DOI 10.1103/PhysRevLett.94.018101, Bibcode 2005PhRvL..94a8101H, lire en ligne)
- (en) Michael Hoffmann, Marius Wanko, Paul Strodel, Peter H. König, Thomas Frauenheim, Klaus Schulten, Walter Thiel, Emad Tajkhorshid et Marcus Elstner, « Color Tuning in Rhodopsins: The Mechanism for the Spectral Shift between Bacteriorhodopsin and Sensory Rhodopsin II », Journal of the American Chemical Society, vol. 128, no 33, , p. 10808-10818 (PMID 16910676, DOI 10.1021/ja062082i, lire en ligne)
- Les protéines, sur anses.fr.
- « Guide de nutrition familiale » (consulté le ).
- (en-US) American Heart Association, « Vegetarian, Vegan Diet & Heart Health », Go Red For Women, (lire en ligne, consulté le ).
- (en) « Position of the American Dietetic Association: Vegetarian Diets », Journal of the American Dietetic Association, , p. 1267–1268 (lire en ligne)
- Adventist Health Study-1 - Cancer Findings: Some Highlights.
- Communiqué OMS Cancérogénicité de la consommation de viande rouge et de viande transformée, octobre 2015.
- Fiche Haricot Sec sur le site Passeport Santé
- Non-soy legume consumption lowers cholesterol levels: a meta-analysis of randomized controlled trials
- Legume consumption and risk of coronary heart disease in US men and women: NHANES I Epidemiologic Follow-up Study
- V. R. Young et P. L. Pellett, « Plant proteins in relation to human protein and amino acid nutrition », The American Journal of Clinical Nutrition, vol. 59, no 5 Suppl, , p. 1203S–1212S (ISSN 0002-9165, PMID 8172124, lire en ligne, consulté le )
- (en) V. R. Young et P. L. Pellett, « Wheat proteins in relation to protein requirements and availability of amino acids », The American Journal of Clinical Nutrition, vol. 41, no 5, , p. 1077–1090 (ISSN 0002-9165 et 1938-3207, DOI 10.1093/ajcn/41.5.1077, lire en ligne, consulté le )
- Sophie Landrin, « En Inde, la religion s’invite dans l’assiette des écoliers », sur Le Monde, (consulté le ).
- « Les domaines d'application de la transgénèse », sur gnis-pedagogie.org (consulté le ).
- « États-Unis - Un maïs enrichi en lysine », sur inf'OGM, (consulté le ).
- Rapport AFSSA/ANSES, pages 232 et 233
- Il est difficile de mesurer précisément la teneur en protéines des champignons car leur chitine et d'autres composés azotés interfèrent avec l'analyse de l'azote total (par exemple la méthode de Kjeldahl) autrefois utilisée. Source : Danell E. et Eaker D. (1992), Amino acid and total protein content of the edible mushroom Cantharellus cibarius (Fries), Journal of the Science of Food and Agriculture, 60(3), 333-337.
- Dıez V.A. et Alvarez A. (2001), Compositional and nutritional studies on two wild edible mushrooms from northwest Spain, Food Chemistry, 75(4), 417-422 (résumé)
- Chang S.T. et Buswell J.A. (1996), Mushroom nutriceuticals, World Journal of Microbiology and biotechnology, 12(5), 473-476 (résumé).
- Peter C.K. Cheung (2009), Nutritional Value and Health Benefits of Mushrooms, Mushrooms as Functional Foods, (71-109).
- (en) V. A. Dı́ez et A. Alvarez, Compositional and nutritional studies on two wild edible mushrooms from northwest Spain. Dans Food Chemistry. Band 75, no 4, décembre 2001, S. 417–422, DOI 10.1016/S0308-8146(01)00229-1
- Chang S.T. et Miles P.G. (1991), Recent trends in world production of edible mushrooms, The Mushroom Journal, 503, 15–18.
- Shah H., Khalil I.A. et Jabeen S. (1997), Nutritional composition and protein quality of Pleurotus mushroom, Sarhad Journal of Agriculture (Pakistan) (résumé Agris/FAO).
- Hsieh, K. Y., Hsu, C. I., Lin, J. Y., Tsai, C. C. et Lin, R. H. (2003), Oral administration of an edible‐mushroom‐derived protein inhibits the development of food‐allergic reactions in mice, Clinical & Experimental Allergy, 33(11), 1595-1602 (résumé)
- Lin, J. Y., Lin, Y. J., Chen, C. C., Wu, H. L., Shi, G. Y. et Jeng, T. W. (1974), Cardiotoxic protein from edible mushrooms, Nature, 252(5480), 235 (résumé).
- « Ciqual Table de composition nutritionnelle des aliments », sur ciqual.anses.fr (consulté le ).
- Albert-François Creff et Daniel Layani, Manuel de diététique en pratique médicale courante, Paris, Masson, , 301 p. (ISBN 978-2-294-01346-1, lire en ligne), p. 4
Voir aussi
Bibliographie
- Gregory Petsko, Dagmar Ringe (trad. Chrystelle Sanlaville, Dominique Charmot-Bensimon), Structure et fonction des protéines, De Boeck, Bruxelles, 2009 (ISBN 978-2-8041-5888-0).
- Lubert Stryer, Jeremy Mark Berg, John L. Tymoczko (trad. Serge Weinman), Biochimie, Flammarion, « Médecine-Sciences », Paris, 2008, 6e éd. (ISBN 978-2-257-00003-3).
- Carl-Ivar Brändén, John Tooze (trad. Bernard Lubochinsky, préf. Joël Janin), Introduction à la structure des protéines, De Boeck Université, Bruxelles, 1996 (ISBN 978-2-8041-2109-9).
Articles connexes
- Combinaison de protéines (alimentation - exemple : riz/lentilles)
- Protéines recombinantes
- Sous-famille de protéines
- Agrégation des protéines
Disciplines et méthodologies
Classes et familles de protéine
Liens externes
- (en) Projet Predictor, un logiciel de calcul partagé qui utilise la plateforme BOINC, pour étudier le repliement des protéines.
- (en) Serveur MRS, un serveur de banques de données biologiques, où l'identification d'une entrée dans la banque PDB permet de visualiser la structure à l'écran, en mode dynamique (voir par exemple ce que produit une recherche dans la banque PDB des entrées correspondant à la trypsine).
- (en) Proteins@home, un projet à grande échelle pour étudier le repliement des protéines, auquel on peut participer avec un ordinateur.

.png.webp)