Modification post-transcriptionnelle
Les modifications post-transcriptionnelles représentent l'ensemble des modifications qu'un ARN subit après avoir été transcrit. On parle également de maturation de l’ARN. Les modifications les plus connues sont l'ajout d'une coiffe, la polyadénylation ou encore l’épissage, mais il en existe bien d'autres.
Définition et rôle
La modification post-transcriptionnelle des ARN est une étape importante du contrôle de l'expression des gènes, on considère généralement que les différentes modifications sont les éléments de régulation post-transcriptionnelle de l'expression des gènes. Ces différentes modifications peuvent influer sur différentes caractéristiques de l'ARN, telle que sa stabilité, sa capacité à être traduit ou bien même modifier la séquence à traduire.
La plupart des ARN sont modifiés post-transcriptionnellement, mais la nature des ARN modifiés et des modifications varient entre procaryote et eucaryote, et entre le noyau et les organites chez les eucaryotes.
Les modifications post-transcriptionnelles
Ajout de la coiffe en 5'
Chez les eucaryotes, certains ARN, et en particulier les ARN messagers, subissent une modification de leur extrémité 5', qui la protège contre la dégradation par des exonucléases. Cette modification, appelée coiffe, consiste en l'addition d'une 7-méthylguanosine sur le premier nucléotide de l'ARN, par une liaison 5'-5' triphosphate[1]. Les deux premiers riboses de l'ARN transcrit peuvent aussi subir une méthylation de leur position 2'.

Dans le cas des ARN messager, cette coiffe sera ensuite reconnue par un complexe protéique spécifique, comprenant notamment le facteur d'initiation eIF4E, qui permet en particulier le recrutement du ribosome et la traduction en protéine.
Polyadénylation en 3'
Le pré ARNm est d'abord clivé dans le noyau par un complexe protéique, au niveau d'un site consensus de polyadénylation (AAUAAA). Ensuite, on a la réaction de polyadénylation qui consiste en l'addition d'environ 200 résidus adénosine (A) sur le produit de clivage en amont de la coupure, alors que le fragment en aval est rapidement dégradé.
On a, comme pour la coiffe, un complexe protéique de reconnaissance qui comprend la PAB (PolyA Binding protein).
Épissage
L'épissage consiste soit en l'excision d'une ou plusieurs séquence (intron) d'un ARN (Cis-épissage), soit en la suture de deux ARN différents reformant au point de jonction un intron qui sera excisé par la suite.
Édition
L'édition des ARN est un terme qui recouvre différents processus aboutissant à la modification de la séquence de l'ARN.
Méthylation
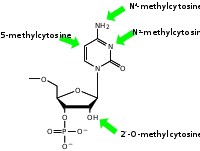
Les méthylations sont parmi les modifications post-transcriptionnelles des nucléotides les plus fréquentes. On distingue deux grandes classes de méthylation :
- La méthylation du ribose sur la position 2'-OH. Les ribonucléotides 2'-O-méthyles sont résistants à l'hydrolyse par des ribonucléases et par des bases. Ils sont plus contraints et perdent la capacité à former des liaisons hydrogène. Les méthylations des riboses sont fréquentes dans les ARNt, les ARN ribosomiques et au niveau de la coiffe des ARN messagers.
- La méthylation des bases. On observe principalement des N-méthylations, comme la N-7-méthylguanine (m7G), présente dans la coiffe des ARN messagers et dans la boucle variable des ARNt. On trouve aussi des C-méthylations, comme dans la ribothymidine (m5U ou rT) ou la 5-méthylcytosine (m5C) présentes dans les ARNt. Ces modifications affectent les capacités d'appariement des bases, en bloquant certaines positions impliquées dans des liaisons hydrogène. Elles peuvent aussi renforcer les interactions hydrophobes d'empilement des bases dans un duplex.
Ces méthylations sont ajoutées de manière post-transcriptionnelle par des méthyltransférases, des enzymes qui utilisent la S-adénosylméthionine comme cofacteur donneur de méthyle.
Pseudouridylation

La pseudouridylation est une isomérisation des uridines qui transforme celles-ci en pseudouridines[2] - [3], par inversion du cycle pyrimidine. C'est la modification chimique la plus fréquente dans l'ARN, avec la 2'-O-méthylation. Par rapport à l'uridine, les pseudouridines ont un groupement NH imino additionnel qui peut former des liaisons hydrogène supplémentaires et stabiliser ainsi la structure de l'ARN.
Chez les eucaryotes, la pseudouridylation des ARN ribosomiques s'effectue dans le nucléole et est guidée par les petits ARN nucléolaires. Ceux-ci contiennent une région complémentaire de la zone entourant la pseudouridine à modifier et s'hybrident à l'ARNr cible, au sein d'un complexe de modification comprenant plusieurs protéines.
Thiolation
La thiolation correspond au remplacement de l'atome d'oxygène d'une fonction oxo d'une base par un atome de soufre. Par exemple, les positions 2 ou 4 de l'uridine peuvent subir des thiolations pour donner la 2-thiouridine (s2U)[4] ou la 4-thiouridine (s4U)[5]. On observe en particulier une base thiolée s4U en position 8 de la majorité des ARN de transfert où elle joue un rôle dans la structure tertiaire de ces ARN[6].
La thiolation des uridines a plusieurs conséquences physico-chimiques. Le remplacement de l'oxygène par un soufre abaisse le pK de l'azote N1 de l'uracile vers 8.0-8.5[5], ce qui rend sa protonation plus facile dans des conditions physiologiques. D'autre part, les bases thiolées absorbent dans l'UV proche (340 nm). Cette seconde propriété est responsable de la sensibilité des cellules à l'irradiation dans l'UV proche, qui provoque des photopontages au niveau des thiouridines dans les ARNt[7].
Notes et références
- (en) Banerjee A.K., « 5'-terminal cap structure in eucaryotic messenger ribonucleic acids. », Microbiol. Rev, vol. 44, no 2, , p. 175-205 (PMID 6247631)
- (en) Cohn W.E., « Pseudouridine, a carbon-carbon linked ribonucleoside in ribonucleic acids: isolation, structure, and chemical characteristics. », J. Biol. Chem., vol. 235, , p. 1488-1498 (PMID 13811056)
- (en) Cortese R., Kammen H.O., Spengler S.J., Ames B.N., « Biosynthesis of pseudouridine in transfer ribonucleic acid. », J. Biol. Chem., vol. 249, , p. 1103-1108 (PMID 4592259)
- (en) Carbon J.A., Hung L., Jones D.S., « A reversible oxidative in activation of specific transfer RNA species. », Proc. Natl. Acad. Sci. USA, vol. 53, no 5, , p. 979-986 (PMID 5330358)
- (en) Lipsett M.N., « The isolation of 4-thiouridylic acid from the soluble ribonucleic acid of Escherichia coli. », J. Biol. Chem., vol. 240, no 10, , p. 3975-3978 (PMID 5320644)
- (en) Saenger W., « Structure and function of nucleosides and nucleotides. », Angew. Chem. Int. Ed. Engl., vol. 12, no 8, , p. 591-601 (PMID 4202101)
- (en) Favre A., Hajnsdorf E., Thiam K., Caldeira de Araujo A., « Mutagenesis and growth delay induced in Escherichia coli by near-ultraviolet radiations. », Biochimie, vol. 67, nos 3-4, , p. 335-342 (PMID 2412594)
Voir aussi
Articles connexes
Bibliographie
- (en) Henri Grosjean et Rob Benne (dir.) Modification and editing of RNA, ASM Press, Washington, 1998, (ISBN 1-55581-133-7)
Liens externes
- The RNA modification database, University of Utah
- Modomics Base de données des mécanismes de modification de l'ARN (Varsovie)