Triose-phosphate isomérase
La triose-phosphate isomérase (TPI) est une isomérase qui catalyse la réaction :
| Triose-phosphate isomérase | ||
 Dimère de triose-phosphate isomérase humaine (PDB 1WYI[1]). | ||
| Caractéristiques générales | ||
|---|---|---|
| Symbole | TPI1 | |
| N° EC | 5.3.1.1 | |
| Classification | ||
| Pfam | PF00121 | |
| Clan Pfam | CL0036 | |
| InterPro | IPR000652 | |
| PROSITE | PDOC00155 | |
| SCOP | 1tph | |
| SUPERFAMILY | 1tph | |
| Homo sapiens | ||
| Locus | 12p13.31 | |
| Masse moléculaire | 30 791 Da[2] | |
| Nombre de résidus | 286 acides aminés[2] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
Cette enzyme intervient à la 5e étape de la glycolyse pour catalyser l'isomérisation réversible de la dihydroxyacétone phosphate (DHAP) en D-glycéraldéhyde-3-phosphate (G3P). Ainsi, chaque molécule de β-D-fructose-1,6-bisphosphate métabolisée par la glycolyse donne en fin de compte deux molécules de D-glycéraldéhyde-3-phosphate.
Elle est présente chez pratiquement tous les êtres vivants où on l'a recherchée, des animaux aux insectes en passant par les mycètes, les plantes et les bactéries. Seules quelques bactéries qui ne possèdent pas le matériel enzymatique nécessaire à la glycolyse ne disposent pas non plus de triose-phosphate isomérase, telles que celles du genre Ureaplasma.
La triose-phosphate isomérase est une enzyme particulièrement efficace, qui réalise cette réaction des milliards de fois plus rapidement que naturellement en solution. Cette réaction est tellement efficace qu'il s'agit d'une enzyme parfaite : elle n'est limitée que par la vitesse de diffusion des molécules entrant et sortant du site actif[3] - [4].
Chez l'homme, un déficit en triose-phosphate isomérase (en) est une maladie neurologique grave, caractérisée par une anémie hémolytique chronique. Cette maladie est induite par diverses mutations, dont la plupart impliquent le changement du résidu de glutamate en aspartate en position 140[5].
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
Structure

La triose-phosphate isomérase est une enzyme homodimérique, c'est-à-dire qu'elle est formée de deux sous-unités identiques, contenant chacune environ 250 résidus d'acides aminés. La structure tertiaire de chaque sous-unité contient huit hélices α à l'extérieur et huit feuillets β parallèles à l'intérieur, l'ensemble formant un tonneau TIM. Le site actif de l'enzyme se trouve au centre de ce tonneau. La réaction catalysée fait intervenir des résidus de glutamate et d'histidine et la séquence entourant le site actif est conservée dans toutes les triose-phosphate isomérases connues.
La structure de cette enzyme est adaptée à sa fonction. Outre les résidus de glutamate et d'histidine judicieusement situés, une chaîne de dix ou onze résidus agit comme une boucle stabilisant l'intermédiaire réactionnel. Cette boucle, constituée des résidus 166 à 176, enferme le substrat et forme une liaison hydrogène avec son groupe phosphate, ce qui stabilise l'intermédiaire ènediol et les autres états intermédiaires de la réaction[6]. En particulier, la liaison hydrogène entre l'enzyme et le groupe phosphate a pour effet de prévenir la décomposition de ces intermédiaires en méthylglyoxal et phosphate inorganique. Le méthylglyoxal est toxique et, s'il se forme, est éliminé par le système glyoxalase[7]. De plus, la formation de glyoxalate conduit à la perte d'un groupe phosphate à haut potentiel de transfert pour le reste de la glycolyse, ce qui est énergétiquement défavorable pour la cellule.
Certaines études suggèrent qu'un résidu de lysine en position 12, proche du site actif, joue également un rôle déterminant dans le fonctionnement de l'enzyme. Ce résidu est protoné à pH physiologique et pourrait ainsi contribuer à neutraliser la charge électrique négative du groupe phosphate. L'enzyme perd toute activité lorsque ce résidu est remplacé par celui d'un acide aminé neutre au cours d'une mutation génétique, tandis qu'elle conserve une certaine activité si ce résidu est remplacé par celui d'un autre acide aminé à chaîne latérale basique[8].
Mécanisme
Le mécanisme réactionnel de la triose-phosphate isomérase implique la formation d'un intermédiaire ènediol. L'enthalpie libre de l'état fondamental et de l'état de transition de chaque intermédiaire a pu être déterminé expérimentalement et son évolution est tracée sur la figure ci-dessous[3] :
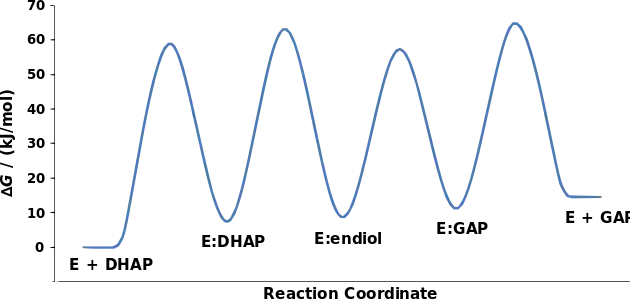 (en) Variations d'enthalpie libre ΔG au cours de la conversion de la dihydroxyacétone phosphate (DHAP) en glycéraldéhyde-3-phosphate (GAP) par la triose-phosphate isomérase (E).
(en) Variations d'enthalpie libre ΔG au cours de la conversion de la dihydroxyacétone phosphate (DHAP) en glycéraldéhyde-3-phosphate (GAP) par la triose-phosphate isomérase (E).
La structure de la triose-phosphate isomérase facilite l'interconversion entre la dihydroxyacétone phosphate et le glycéraldéhyde-3-phosphate. Le résidu nucléophile de Glu-165 de l'enzyme agit en déprotonant le substrat[9], tandis que le résidu électrophile d'His-95 cède un proton pour former l'intermédiaire ènediol[10] - [11]. L'intermédiaire ènediolate déprotoné absorbe un proton du résidu de Glu-165 protoné pour donner le glycéraldéhyde-3-phosphate. La réaction inverse est catalysée de façon analogue, avec le même intermédiaire ènediol, mais avec une réaction sur l'atome d'oxygène en C2[6].
La triose-phosphate isomérase est une enzyme limitée par la diffusion des substrats — c'est-à-dire une enzyme parfaite. D'un point de vue thermodynamique, la formation de dihydroxyacétone phosphate est favorisée à 20:1 par rapport à la formation de glycéraldéhyde-3-phosphate[12]. Cependant, ce dernier est consommé par la glycéraldéhyde-3-phosphate déshydrogénase ou cours de la glycolyse, ce qui déplace l'équilibre vers la formation de ce composé au détriment du dihydroxyacétone phosphate.
La triose-phosphate isomérase est inhibée par les ions sulfate SO42−, phosphate PO43− et arséniate AsO43−, qui se lient au site actif[13]. Parmi les autres inhibiteurs de cette enzyme, on note le 2-phosphoglycolate, un analogue de l'état de transition (en), et le D-glycérol-1-phosphate (en), analogue structurel du substrat[14].
Notes et références
- (en) T. Kinoshita, R. Maruki, M. Warizaya, H. Nakajima et S. Nishimura, « Structure of a high-resolution crystal form of human triosephosphate isomerase: improvement of crystals using the gel-tube method », Acta Crystallographica Section F, vol. 61, no Pt 4, , p. 346-349 (PMID 16511037, PMCID 1952429, DOI 10.1107/S1744309105008341, lire en ligne)
- Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
- (en) W. John Albery et Jeremy R. Knowles, « Free-energy profile for the reaction catalyzed by triosephosphate isomerase », Biochemistry, vol. 15, no 25, , p. 5627-5631 (PMID 999838, DOI 10.1021/bi00670a031, lire en ligne)
- (en) Irwin A. Rose, Wen Jian Fung et Jessie V. B. Warms, « Proton diffusion in the active site of triosephosphate isomerase », Biochemistry, vol. 29, no 18, , p. 4312-4317 (PMID 2161683, DOI 10.1021/bi00470a008, lire en ligne)
- (en) Ferenc Orosz, Judit Oláh et Judit Ovádi, « Triosephosphate isomerase deficiency: Facts and doubts », IUBMB Life, vol. 58, no 12, , p. 703-715 (PMID 17424909, DOI 10.1080/15216540601115960, lire en ligne)
- (en) Jeremy R. Knowles, « Enzyme catalysis: not different, just better », Nature, vol. 350, no 6314, , p. 121-124 (PMID 2005961, DOI 10.1038/350121a0, lire en ligne)
- (en) Donald J. Creighton et Diana S. Hamilton, « Brief History of Glyoxalase I and What We Have Learned about Metal Ion-Dependent, Enzyme-Catalyzed Isomerizations », Archives of Biochemistry and Biophysics, vol. 387, no 1, , p. 1-10 (PMID 11368170, DOI 10.1006/abbi.2000.2253, lire en ligne)
- (en) Patricia J. Lodi, Louise C. Chang, Jeremy R. Knowles et Elizabeth A. Komives, « Triosephosphate Isomerase Requires a Positively Charged Active Site: The Role of Lysine-12 », Biochemistry, vol. 33, no 10, , p. 2809-2814 (PMID 8130193, DOI 10.1021/bi00176a009, lire en ligne)
- (en) T. Alber, D. W. Banner, A. C. Bloomer, G. A. Petsko, David Phillips, P. S. Rivers et I. A. Wilson, « On the Three-Dimensional Structure and Catalytic Mechanism of Triose Phosphate Isomerase », Philosophical Transactions B, vol. 293, no 1063, , p. 159-171 (PMID 6115415, DOI 10.1098/rstb.1981.0069, lire en ligne)
- (en) Elliott B. Nickbarg, Robert C. Davenport, Gregory A. Petsko et Jeremy R. Knowles, « Triosephosphate isomerase: removal of a putatively electrophilic histidine residue results in a subtle change in catalytic mechanism », Biochemistry, vol. 27, no 16, , p. 5948-5960 (PMID 2847777, DOI 10.1021/bi00416a019, lire en ligne)
- (en) Elizabeth A. Komives, Louise C. Chang, Elias Lolis, Robert F. Tilton, Gregory A. Petsko et Jeremy R. Knowles, « Electrophilic catalysis in triosephosphate isomerase: the role of histidine-95 », Biochemistry, vol. 30, no 12, , p. 3011-3019 (PMID 2007138, DOI 10.1021/bi00226a005, lire en ligne)
- (en) Thomas K. Harris, Robert N. Cole, Frank I. Comer et Albert S. Mildvan, « Proton Transfer in the Mechanism of Triosephosphate Isomerase », Biochemistry, vol. 37, no 47, , p. 16828-16838 (PMID 9843453, DOI 10.1021/bi982089f, lire en ligne)
- (en) Anne-Marie Lambeir, Fred R. Opperdoes et Rik K. Wierenga, « Kinetic properties of triose-phosphate isomerase from Trypanosoma brucei brucei », The FEBS Journal, vol. 168, no 1, , p. 69-74 (PMID 3311744, DOI 10.1111/j.1432-1033.1987.tb13388.x, lire en ligne)
- (en) Elias Lolis et Gregory A. Petsko, « Crystallographic analysis of the complex between triosephosphate isomerase and 2-phosphoglycolate at 2.5-Å resolution: implications for catalysis », Biochemistry, vol. 29, no 28, , p. 6619-6625 (PMID 2204418, DOI 10.1021/bi00480a010, lire en ligne)


