Mécanique moléculaire
La mécanique moléculaire correspond à l'utilisation de la mécanique newtonienne pour modéliser la structure des systèmes moléculaires. L'approche de la mécanique moléculaire est souvent appliquée pour améliorer des structures moléculaires ou des simulations utilisant soit la dynamique moléculaire, soit la méthode de Monte-Carlo.
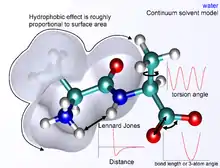
Typiquement, la mécanique moléculaire considère l'ensemble des interactions entre une collection d'atomes sphériques reliés entre eux par des ressorts fictifs qui représentent les liaisons chimiques. La représentation de tels interactions interatomiques engendre des forces internes et sont généralement décrites par de simples fonctions mathématiques qui peuvent dépendre soit de la distance entre paires atomiques ou de leurs angles. Parfois, la loi de Hooke peut-être employée pour représenter très simplement les interactions de liaison. Les interactions entre atomes peuvent être traités soit en les assimilant à des sphères dures non-élastiques ou en interaction avec une énergie potentielle, par exemple sous la forme d'un potentiel de Lennard-Jones.
Voir aussi
Logiciels
Cette liste n'est pas exhaustive :
Références
- U. Burkert and N.L. Allinger, Molecular Mechanics, 1982, (ISBN 0841208859)
- O. Becker, A.D. MacKerell, Jr., B. Roux and M. Watanabe, Editors, Computational Biochemistry and Biophysics, Marcel Dekker Inc., New York, 2001, (ISBN 082470455X)
- MacKerell, A.D., Jr., Empirical Force Fields for Biological Macromolecules: Overview and Issues, Journal of Computational Chemistry, 25: 1584-1604, 2004