Sélénocystéine
La sélénocystéine (abréviations IUPAC-IUBMB : Sec et U) est un acide α-aminé non standard dont l'énantiomère L est l'un des 22 acides aminés protéinogènes, encodé sur les ARN messagers par le codon-stop opale UGA en présence d'une séquence d'insertion dite élément SECIS. Il s'agit d'un analogue sélénié de la cystéine qui entre dans la constitution de certaines enzymes de la classe des oxydoréductases[3] telles que la glutathion peroxydase, la thiorédoxine réductase, les iodothyronine désiodases (thyroxine 5'-désiodase et thyroxine 5-désiodase), la glycine réductase ou encore la formiate déshydrogénase, qu'on nomme sélénoprotéines. On dénombre actuellement trois gènes codant des sélénoprotéines chez E. coli. La tellurocystéine est un autre analogue de la cystéine, avec un atome de tellure à la place du sélénium.
| Sélénocystéine | |
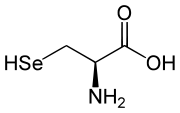 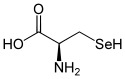 L ou R(+)-sélénocystéine et D ou S(–)-sélénocystéine |
|
| Identification | |
|---|---|
| Nom UICPA | acide 2-amino- 3-selanyl-propanoïque |
| Synonymes |
U, Sec |
| No CAS | L ou R (racémique) |
| No ECHA | 100.236.386 |
| PubChem | 6326983 |
| ChEBI | 16633 |
| SMILES | |
| InChI | |
| Propriétés chimiques | |
| Formule | C3H7NO2Se [Isomères] |
| Masse molaire[1] | 168,05 ± 0,03 g/mol C 21,44 %, H 4,2 %, N 8,33 %, O 19,04 %, Se 46,99 %, |
| Propriétés biochimiques | |
| Codons | Codon-stop UGA opale avec élément SECIS |
| pH isoélectrique | 5,47[2] |
| Acide aminé essentiel | non |
| Unités du SI et CNTP, sauf indication contraire. | |
Contrairement à la plupart des acides aminés non standards, qui n'appartiennent pas à la série des 20 acides aminés codés directement par le code génétique, la sélénocystéine n'est pas formée sur la protéine par modification post-traductionnelle d'acides aminés standards après la traduction de l'ARNm, mais est incorporée directement lors de la synthèse de la chaîne polypeptidique par le ribosome.
Historique
Un acide aminé comportant du sélénium a été isolé en 1941[4], identifié plus tard comme une méthylsélénocystéine[5]. En 1976, on démontre que cette dernière est le composant sélénié des sélénoprotéines[6].
Synthèse
Malgré son nom, la sélénocystéine dérive métaboliquement de la sérine, convertie en sélénocystéine alors que le résidu d'acide aminé est déjà lié à un ARN de transfert de sélénocystéine, noté ARNtSec. Structurellement, en revanche, elle est analogue à une molécule de cystéine dont on aurait remplacé l'atome de soufre par du sélénium, le groupe thiol étant remplacé par un groupe sélénol.
La formation du sélénocystéinyl-ARNtSec commence par l'estérification d'une molécule d'ARNtSec par une molécule de sérine sous l'action de la séryl-ARNt synthétase pour former le séryl-ARNtSec. Ce dernier est ensuite converti en sélénocystéinyl-ARNtSec par plusieurs enzymes.
- Chez les bactéries, la réaction est directe et catalysée par une seule enzyme, la sélénocystéine synthase[7] :
- Chez les archées et les eucaryotes, en revanche, la réaction se déroule en deux étapes[8] - [9] ; tout d'abord, la O-phosphoséryl-ARNtSec kinase convertit le séryl-ARNtSec en O-phosphoséryl-ARNtSec, puis la O-phosphoséryl-ARNt:sélénocystéinyl-ARNt synthase convertit ce dernier en sélénocystéinyl-ARNtSec :
- ATP + L-séryl-ARNtSec ADP + O-phospho-L-séryl-ARNtSec ;
- O-phospho-L-séryl-ARNtSec + SePO33− + H2O L-sélénocystéinyl-ARNtSec + 2 phosphate.
Ces enzymes utilisent le sélénophosphate SePO33− comme donneur activé de sélénium, produit par la sélénophosphate synthase[10] à partir de séléniure Se2−.
Incorporation dans les protéines
L'incorporation de la sélénocystéine dans les protéines est un processus complexe, car il n'existe pas à proprement parler de codon spécifique pour cet acide aminé. Dans l'ARN messager, à la position correspondant à la sélénocystéine dans la protéine, on trouve un codon UGA, qui est normalement un codon-STOP (appelé « opale »), signal d'arrêt de la traduction. Il y a cependant un ARNt spécifique permettant l'incorporation de cet acide aminé dans la chaîne polypeptidique en formation, dont l'anticodon est complémentaire de UGA[11]. La discrimination entre le codon UGA codant la sélénocystéine du codon-STOP UGA nécessite un mécanisme spécifique :
- Il existe une machinerie protéique particulière qui s'associe à l'ARNt sélénocystéine pour diriger son incorporation au site A du ribosome pendant l'étape d'élongation. Il s'agit de l'isopenténylation du complexe Sélénocystéinyl-ARNtSec, qui correspond à l'ajout d'une courte chaîne hydrocarbonée sur une Adénine de l'ARNt.
- l'ARN messager porte un élément de structure secondaire spécifique appelé élément SECIS (pour Selenocystein Insertion Sequence) qui est impliqué dans le recrutement du complexe protéine/ARNt chargé. Le sélénocystéinyl-ARNt est alors incorporé au site A du ribosome.
Chez les bactéries, le système est composé du facteur protéique SelB, homologue au facteur d'élongation « classique » EF-Tu capable de reconnaître les 20 autres aminoacyl-ARNt. SelB est homologue à EF-Tu sur sa partie N-terminale et de ce fait est capable de lier le GTP, l'ARNt chargé et d'interagir avec le ribosome. Il possède de plus une extension C-terminale composée de quatre domaines en "winged-helix" se liant avec une très forte affinité (nM) à l'élément SECIS, une épingle-à-cheveux, ou « tige-boucle », dans l'ARNm située peu après le codon UGA. L'élément SECIS est composé d'une quarantaine de nucléotides de séquence variable mais dont certains éléments invariants et nécessaires à l'interaction avec SelB ont pu être mis en évidence.
Chez les eucaryotes et les archées, le mécanisme est légèrement différent. Chez les eucaryotes, deux protéines en complexe, mSelB et SBP2, jouent respectivement le rôle de facteur d'élongation et de liaison aux éléments SECIS. Ces derniers sont également différents en séquence et en taille des SECIS procaryotes, et se situent le plus souvent en 3' hors de la partie codante du gène.
Notes et références
- Masse molaire calculée d’après « Atomic weights of the elements 2007 », sur www.chem.qmul.ac.uk.
- (en) Francis A. Carey, « Table of pKa and pI values », sur Département de chimie de l'université de Calgary, (consulté le )
- Voir Carence alimentaire en sélénium
- Horn MJ, Jones DB, Isolation from Astragalus pectinatus of a crystalline amino acid complex containing selenium and sulfur, J Biol Chem, 1941;139:649-660
- Trelease SF, Di Somma AA, Jacobs AL, Seleno-amino acid found in Astragalus bisulcatus, Science, 1960;132:618-618
- Cone JE, Del Rio RM, Davis JN, Stadtman TC, Chemical characterization of the selenoprotein component of clostridial glycine reductase: identification of selenocysteine as the organoselenium moiety, Proc Natl Acad Sci U S A, 1976;73:2659-2663
- Forchhammer K, Bock A, « Selenocysteine synthase from Escherichia coli. Analysis of the reaction sequence », J. Biol. Chem., vol. 266, no 10, , p. 6324–8 (PMID 2007585)
- (en) Sotiria Palioura, R. Lynn Sherrer, Thomas A. Steitz, Dieter Söll et Miljan Simonović, « The Human SepSecS-tRNASec Complex Reveals the Mechanism of Selenocysteine Formation », Science, vol. 325, no 5938, , p. 321-325 (PMID 19608919, PMCID 2857584, DOI 10.1126/science.1173755, Bibcode 2009Sci...325..321P, lire en ligne)
- (en) Yuhei Araiso, Sotiria Palioura, Ryuichiro Ishitani, R. Lynn Sherrer, Patrick O’Donoghue, Jing Yuan, Hiroyuki Oshikane, Naoshi Domae, Julian DeFranco, Dieter Söll et Osamu Nureki, « Structural insights into RNA-dependent eukaryal and archaeal selenocysteine formation », Nucleic Acids Research, vol. 36, no 4, , p. 1187-1199 (PMID 18158303, PMCID 2275076, DOI 10.1093/nar/gkm1122, lire en ligne)
- (en) Z. Veres, L. Tsai, T. D. Scholz, M. Politino, R. S. Balaban et T. C. Stadtman, « Synthesis of 5-methylaminomethyl-2-selenouridine in tRNAs: 31P NMR studies show the labile selenium donor synthesized by the selD gene product contains selenium bonded to phosphorus », Proceedings of the National Academy of Sciences of the United States of America, vol. 89, no 7, , p. 2975–2979 (PMID 1557403, PMCID 48786, DOI 10.1073/pnas.89.7.2975)
- Chambers I, Frampton J, Goldfarb P, Affara N, McBain W, Harrison PR, The structure of the mouse glutathione peroxidase gene: the selenocysteine in the active site is encoded by the `termination' codon, TGA, EMBO J, 1986;5:1221-1227
