Archaea
Les archées (/aʁ.ke/) ou Archaea (du grec ancien ἀρχαῖος / arkhaîos, « originel, primitif »), anciennement appelés archéobactéries, sont des microorganismes unicellulaires procaryotes, c'est-à-dire des êtres vivants constitués d'une cellule unique qui ne comprend ni noyau ni organites, à l'instar des bactéries. D'apparence souvent semblable à ces dernières, les archées ont longtemps été considérées comme des bactéries extrêmophiles particulières, jusqu'à ce que les recherches phylogénétiques sur les procaryotes, commencées en 1965[2], aboutissent, avec les travaux de Carl Woese et George E. Fox[3], à la publication en 1977 d'un arbre phylogénétique fondé sur les séquences des gènes d'ARN ribosomique des organismes étudiés, arbre dans lequel les procaryotes étaient scindés en deux domaines distincts, celui des bactéries et celui des archées[4] - [5]. On sait aujourd'hui que l'arbre des eucaryotes prend naissance parmi des archées d'Asgård. Ainsi les archées forment un clade avec les Eukaryota mais constituent un groupe paraphylétique situé à la base de ces derniers[6].
- Archaebacteria
- Mendosicutes
- Metabacteria
- Domaine : Bacteria
- Domaine : Archaea
- Règne : Archaea
- Domaine : Eukaryota
.jpg.webp)
Du point de vue de leur génétique, leur biochimie et leur biologie moléculaire, les archées sont des organismes aussi différents des bactéries que des eucaryotes. Les enzymes réalisant la réplication de l'ADN, la transcription de l'ADN en ARN ainsi que la traduction de l'ARN messager en protéines chez les archées sont apparentées à celles des eucaryotes et non à celles des bactéries, de même que la présence d'histones dans le matériel génétique des archées rapproche ces dernières des eucaryotes et les distingue des bactéries. Par ailleurs, les gènes des archées possèdent des introns et leur ARN messager subit des modifications post-transcriptionnelles, ce qui est le cas également chez les eucaryotes mais pas chez les bactéries. D'autre part, certaines archées possèdent des voies métaboliques qui n'existent ni chez les bactéries ni chez les eucaryotes, comme la méthanogenèse chez les archées méthanogènes, tandis que les archées dans leur ensemble sont dépourvues d'acide gras synthase, contrairement à la fois aux bactéries et aux eucaryotes : elles font un usage très limité des acides gras, et leur membrane plasmique est constituée essentiellement d'étherlipides, à la différence des bactéries et des eucaryotes. Un autre trait propre aux archées est la présence chez certaines d'entre elles d'une paroi cellulaire constituée de pseudopeptidoglycane, ou pseudomuréine.
Les archées ont longtemps été vues comme des organismes essentiellement extrêmophiles présents notamment dans les sources hydrothermales océaniques, les sources chaudes volcaniques ou encore les lacs salés, mais on en a découvert depuis dans toute une variété de biotopes qui ne sont pas nécessairement extrêmes, tels que le sol, l'eau de mer, des marécages, la flore intestinale et orale[7] et même le nombril humain[8]. Les archées seraient particulièrement nombreuses dans les océans, et celles faisant partie du plancton constitueraient l'un des groupes d'organismes les plus abondants de la Terre. Les archées interviennent par ailleurs de façon non négligeable dans le cycle du carbone et le cycle de l'azote. On ne connaît pas vraiment d'exemple d'archée pathogène ou parasite, mais elles sont souvent mutualistes ou commensales. Les archées méthanogènes de l'intestin humain et des ruminants participent ainsi favorablement à la digestion.
Descriptif
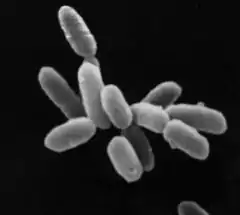
La taille et la forme des archées sont généralement semblables à celles des bactéries, bien que certaines espèces d’archées présentent une forme inhabituelle, comme Haloquadratum walsbyi dont la cellule est plate et carrée. En dépit de ces similitudes visuelles avec les bactéries, les archées s’en distinguent par certains caractères biochimiques, comme la constitution de la membrane cellulaire. De plus, elles présentent des gènes et des voies métaboliques semblables à ceux rencontrés chez les eucaryotes, notamment les enzymes impliquées dans le mécanisme de réplication de l'ADN, la transcription et la traduction. Les archées utilisent une plus grande variété de sources d’énergie que les eucaryotes : composé organique comme les sucres, l’ammoniac, les ions métalliques et même l’hydrogène gazeux comme nutriments. Les Halobacteria utilisent la lumière solaire comme source d’énergie, et certaines espèces d’archées peuvent fixer le carbone. Cependant, il n’y a pas d’espèces d’archées connues capables de réaliser ces deux phénomènes, comme le font les plantes chez les eucaryotes et les cyanobactéries. Les archées se reproduisent de manière asexuée et se divisent par fission binaire, fragmentation ou bourgeonnement. Par opposition aux bactéries et aux eucaryotes, aucune espèce d’archée identifiée à ce jour n’est capable de former des spores.
Les archées sont extrêmement diversifiées. Certaines sont connues pour leur capacité à vivre dans des conditions extrêmes et occupent des niches écologiques qu'elles sont souvent seules à occuper (pH proche de 0, température supérieure à 100 °C, salinité élevée par exemple), mais il existe beaucoup d’archées vivant dans des biotopes plus courants et très variés comme le sol, les lacs, la mer ou l’intestin des animaux. Ces procaryotes sont maintenant ainsi reconnus comme une part majeure du vivant sur Terre, ils peuvent jouer un rôle dans le cycle du carbone et le cycle de l'azote. Il n’y a pas d’exemple clairement reconnu d’archées pathogènes ou parasites, mais il existe des espèces mutualistes ou commensales. Par exemple, les archées méthanogènes du tractus intestinal de l’homme et des ruminants participent à la digestion des aliments. Les archées ont également une importance en technologie, avec par exemple l’utilisation des méthanogènes pour produire des biogaz ou leur participation au traitement des eaux usées. Par ailleurs, les enzymes des archées extrêmophiles, résistantes aux températures élevées et aux solvants organiques, sont exploitées en biotechnologie[9].
Classification
Nouveau domaine
Au début du XXe siècle, les procaryotes étaient considérés comme un seul groupe d'organismes et classés en fonction de leur biochimie, de leur morphologie et du métabolisme. Par exemple, les microbiologistes essayaient de classer les micro-organismes sur la base des structures de leurs parois cellulaires, leurs formes, et les substances qu'ils consomment. Cependant, une nouvelle approche a été proposée en 1965 qui permet d’étudier les liens de parenté entre les procaryotes en utilisant les séquences des gènes de ces organismes. Cette approche, connue sous le nom de la phylogénétique, est la méthode utilisée aujourd'hui.
Les archées ont d'abord été classées comme un groupe distinct des procaryotes en 1977 par Carl Woese (professeur à l'université de l'Illinois à Urbana aux États-Unis) et George E. Fox dans les arbres phylogénétiques fondés sur les séquences de l’ARN ribosomique 16S (ARNr) des gènes[3]. Ces deux groupes ont été initialement nommés les eubactéries et archaeobactéries et traités comme sous-règne ou règne. Woese fait valoir que ce groupe de procaryotes est fondamentalement différent des bactéries. Pour souligner cette différence, et pour insister sur le fait qu’ils composent, avec les eucaryotes, trois domaines bien distincts du vivant, ces deux domaines ont plus tard été renommés Archaea et Bacteria[10]. Le mot archées vient du grec ancien ἀρχαῖα, qui signifie « choses anciennes ». Pour leur part, d'autres comme Thomas Cavalier-Smith considéraient que les archées, alors appelées Archaebacteria, ne sont qu'un embranchement (phylum) des Unibacteria dans le règne des bactéries[11].
Dans un premier temps, seules les bactéries méthanogènes, isolées initialement par Carl Woese, ont été placées dans ce nouveau domaine et les archées ont été considérées comme des extrêmophiles qui n'existent que dans les habitats tels que les sources chaudes et les lacs salés : Woese découvre en 1979 les hyperhalophiles (genre Halobacterium) puis les thermoacidophiles (Thermoplasma acidophilum, Sulfolobus acidocaldarius), Karl Stetter isole en 1981 une archée anaérobie hyperthermophile, Pyrococcus furiosus. À la fin du XXe siècle, les microbiologistes se sont rendu compte que les archées sont en fait un grand groupe diversifié d'organismes qui sont très répandus dans la nature et qui sont communs dans une diversité d’habitats, tels que les sols et les océans[12]. Cette nouvelle appréciation de l’importance et de l'ubiquité des archées a été rendu possible grâce à la réaction en chaîne par polymérase pour détecter les procaryotes dans des échantillons d'eau ou de sol à partir de leurs acides nucléiques. Cela permet la détection et l'identification d'organismes qui ne peuvent pas être cultivés en laboratoire, ou dont la culture est difficile[13] - [14].
Classification actuelle
Ces organismes ont longtemps été regroupés sous le terme générique de « procaryotes » avec les bactéries par opposition aux eucaryotes. Pour les différencier, les microbiologistes avaient élaboré un système de comparaison et de classification fondé sur de petites différences visibles au microscope, ainsi que sur des différences physiologiques (capacité à se développer sur un certain milieu par exemple).
Dès qu'il a été question d'élucider les relations généalogiques entre les différents procaryotes, les biologistes ont dû se rendre à l'évidence : les différences nutritionnelles et phénotypiques ne permettraient pas de classer correctement les différents organismes. Au cours des années 1970, les biologistes ont pris conscience de l'existence irremplaçable d'information, au cœur même des cellules des êtres vivants, permettant de déterminer la phylogénie, l'ADN. Le gène identifié dans une cellule est le variant d'un gène qui a existé il y a de très nombreuses années. La comparaison gène à gène entre deux organismes permet donc de mesurer le temps écoulé depuis la divergence à partir de l'ancêtre commun.
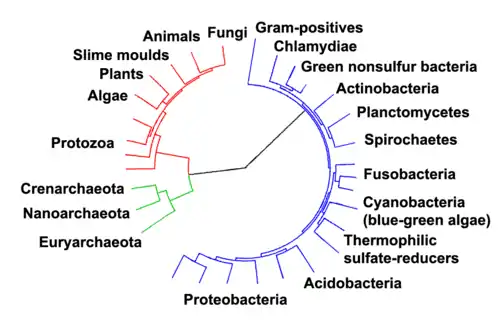
Carl Woese s'est rendu compte que l'ARN ribosomique (ou ARNr, une des molécules contenues dans la cellule) des organismes qu'il étudiait permettait de mettre en évidence l'existence de deux groupes clairement séparés : les bactéries et les archéobactéries. Plus précisément, les ARNr des archées sont en fait aussi différents des ARNr des bactéries que de ceux des eucaryotes. Woese en a conclu qu'il ne fallait plus uniquement séparer en deux grands groupes le monde du vivant, en fonction de la présence ou de l'absence d'un noyau, mais plutôt en trois domaines primitifs : les bactéries, les archées et les eucaryotes.
De nombreuses études confirment le caractère monophylétique de l'ensemble constitué des archées et des eucaryotes (mais pas des archées seules). Ces microorganismes ressemblent par leur forme aux bactéries, mais d'un point de vue moléculaire, si certains de leurs traits les rapprochent des bactéries, d'autres les rapprochent plutôt des eucaryotes. Il n'est donc pas possible de présenter les archées comme des ancêtres des bactéries.
Le classement des archées, et des procaryotes en général, est à la fois en évolution rapide et un domaine litigieux. Sur la base de critères uniquement métaboliques, les archées ont été divisées en quatre grands groupes selon qu'elles sont méthanogènes, halophiles, thermophiles ou sulfo-dépendantes.
Les systèmes de classifications actuels visent à organiser les archées en groupes d'organismes qui partagent des caractéristiques structurelles et des ancêtres communs[15]. Ces classifications s'appuient fortement sur l'usage de la séquence des gènes de l'ARN ribosomique pour révéler les relations entre les organismes (phylogénétique moléculaire)[16]. La plupart des archées cultivables sont membres de deux principaux embranchements : Euryarchaeota et Crenarchaeota. D'autres groupes ont été provisoirement créés. Par exemple, les espèces propres Nanoarchaeum equitans, qui ont été découvertes en 2003, ont été classées dans un nouveau phylum : Nanoarchaeota[17]. Un nouveau phylum, Korarchaeota, a également été proposé ; il contient un petit groupe d'espèces thermophiles inhabituelles qui partagent les caractéristiques des deux principaux embranchements, mais qui sont plus étroitement liées aux Crenarchaeota[18] - [19]. Récemment mises en évidence, d'autres espèces d'archées, tels que les archaeal Richmond Mine acidophilic nanoorganisms (ARMAN), qui ont été découvertes en 2006, sont liées seulement de loin aux autres groupes antérieurement connus[20]. Le superphylum TACK a été proposé en 2011, pour regrouper les Thaumarchaeota, Aigarchaeota, Crenarchaeota et Korarchaeota [21].
L'archée Loki, identifiée en 2015 par son génome qualifié de Candidatus en nomenclature bactérienne, appartiendrait, du point de vue phylogénétique, à l'embranchement le plus proche des eucaryotes[22] - [23]. De nouvelles lignées d'archées, apparentées à Loki, ont été identifiées dans les sédiments aquatiques par analyse métagénomique. Les archées Odin, Thor et Heimdall, formeraient, avec Loki, le super-embranchement Candidatus "Asgard", proposé en 2017 d'après le nom du royaume des dieux de la mythologie nordique[24]. Une autre étude du NCBI[25] indique que les archées d'Asgård et les Eukaryotes formeraient un clade monophylétique nommé Eukaryomorpha.
Liste des embranchements
Selon LPSN[26]
et proposés[27]
- Aenigmarchaeota (es)
- Aigarchaeota
- Altiarchaeota
- Bathyarchaeota (es)
- Diapherotrites (es)
- Geoarchaeota (es)
- Geothermarchaeota
- Heimdallarchaeota
- Helarchaeota
- Hydrothermarchaeota
- Lokiarchaeota
- Mamarchaeota
- Marsarchaeota
- Micrarchaeota (es)
- Nanohaloarchaeota
- Nezhaarchaeota
- Odinarchaeota
- Pacearchaeota (es)
- Parvarchaeota (es)
- Thorarchaeota
- Verstraetearchaeota
- Woesearchaeota (es)
D'après Tom A. Williams et al., 2017[28] et Castelle & Banfield, 2018[29] (DPANN) :
| Neomura |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Origine et évolution
Bien que les fossiles connus de cellules procaryotes aient été datés de près de 3,5 milliards d'années, la plupart des procaryotes n'ont pas de morphologies distinctives et les formes des fossiles ne peuvent pas être utilisées pour les identifier comme étant des archées[32]. Par contre, les fossiles chimiques, sous la forme des lipides caractéristiques des archées, donnent plus d'informations, car ces composés n’existent pas dans d'autres groupes d'organismes[33]. Certaines publications ont suggéré que des lipides fossiles provenant de procaryotes ou d’eucaryotes étaient présents dans les schistes datant de 2,7 milliards d'années[34]. Depuis, ces données ont toutefois été sujettes à question[35]. Ces lipides ont également été détectés dans des roches datant du Précambrien. Les plus anciennes traces connues de ces lipides isopréniques proviennent des roches de la formation d'Isua à l'ouest du Groenland, qui comprennent des sédiments formés il y a 3,8 milliards d'années et qui sont les plus anciens sur Terre[36].
Une fossilisation expérimentale est partie du principe que les premiers fossiles (> 3 Ga) se sont formés par silicification, c'est-à-dire via la précipitation de silice sur des structures cellulaires)[37]. On a fossilisé en laboratoire des souches différentes d'Archées (Methanocaldococcus jannaschii (en) et Pyrococcus abyssi) et de Bactéries (Chloroflexus aurantiacus (en) et Geobacillus (en) sp.) jugées proches des micro-organismes (thermophiles, anaérobies et autotrophes) qui ont colonisé la Terre primitive (et proches d'organismes qui auraient éventuellement pu avoir vécu sur la Planète Mars)[37]. Leur observation en microscopie électronique (MEB, MET, Cryo-MEB) a donné des indications morphologiques utiles pour le repérage de vrais fossiles anciens (à ne pas confondre avec des structures prébiotiques de type sphères submicrométriques, tubules et éléments filamenteux ou d'apparence fibreuses possiblement issus d'une simple chimie organique[38]) ; de même pour des analyses chimiques (GC, GC-MS, HPLC) ont apporté des données sur la dégradation/préservation de la matière organique durant ce processus de fossilisation par silicification[37]. Ce travail a confirmé que certains micro-organismes qui ne se silicifient pas : ainsi l'archée M. jannaschii s'est rapidement lysée alors que P. abyssi, Geobacillus sp. et C. aurantiacus se silicifiaient mais avec une intensité propre à chaque espèce. Certains de ces micro-organismes lors de la silicification tentent d'y survivre en produisant des EPS (Exopolysaccharides) ou via un mécanisme de répulsion de la silice[37]. Les fossiles déjà découverts ne sont donc pas nécessairement représentatifs des espèces réellement présentes à l'époque (ni de leur nombre ou dominance)[37].
Woese propose que les bactéries, les archées et les eucaryotes représentent trois lignées séparées qui aurait divergé à partir d’une colonie d'organismes ancestrale[39] - [40]. D’autres biologistes, comme Gupta ou Cavalier-Smith, cependant, ont proposé que les archées et les eucaryotes proviennent d'un groupe de bactéries[41] - [42]. Il est possible que le dernier ancêtre commun des bactéries et des archées soit un organisme thermophile, ce qui soulève la possibilité que la vie soit apparue dans des conditions de températures élevées[43]. Cette hypothèse n’est toutefois pas approuvée par l’ensemble de la communauté scientifique[44] - [45]. Par ailleurs, le fait que les archées sont plus proches des eucaryotes que des bactéries, conduit à penser le terme procaryote n'a pas de pertinence phylogénétique et devrait être rejeté[46].
La relation entre les archées et les eucaryotes reste un sujet d'importance[47]. En plus des similitudes dans la structure cellulaire et les mécanismes biochimiques qui sont discutées ci-après, de nombreux arbres phylogénétiques groupent les archées et les eucaryotes ensemble. Toutefois, la découverte de gènes provenant d’archées dans le génome de certaines bactéries, telles que Thermotoga maritima, complique l'analyse des relations entre organismes, étant donné l’importance des transferts horizontaux de gènes[48].
Une théorie totalement différente, non basée sur les axiomes courants d'ancêtre commun d'une lignée et de différenciation arborescente, est la théorie endosymbiotique[49]. Selon celle-ci, les eucaryotes se sont développés à partir d’une fusion entre des bactéries et des archées, fusion elle-même découlant de l'évolution d'une relation symbiotique. Cette théorie est aujourd'hui largement acceptée en raison de la variété de faits connus qui la soutiennent. Au sein des archées, le groupe le plus proche des eucaryotes est le superphylum des archées d'Asgård (Asgardarchaeota)[50]. Leur génome code une série de protéines identiques ou similaires à des protéines qu'on pensait spécifiques des eucaryotes, et notamment l'actine qui forme le cytosquelette[51]. Les mitochondries proviendraient quant à elles de l'endosymbiose d'une rhodobactérie (une alpha-protéobactérie)[52].
Génome et génétique
Les archées ont généralement un seul chromosome circulaire. Le plus grand génome archéen séquencé à ce jour est celui de Methanosarcina acetivorans[53] avec 5 751 492 paires de bases alors que le génome de Nanoarchaeum equitans, le plus petit séquencé à ce jour fait un dixième de cette taille avec seulement 490 885 paires de base. Il est estimé que le génome de Nanoarchaeum equitans comporte 537 gènes codant des protéines[54]. Les éléments extrachromosomiques, appelés plasmides sont également présents chez les archées. Ces plasmides peuvent être transférés entre les cellules par contact physique, dans un processus qui pourrait être similaire à la conjugaison bactérienne[55] - [56].
Reproduction
La reproduction des archées a lieu de manière asexuée par division binaire, par fission multiple ou par fragmentation. La méiose ne se produit pas, tous les descendants ont le même matériel génétique. Après la réplication de l’ADN, les chromosomes sont séparés et la cellule se divise[57]. Les détails du cycle cellulaire des archées ont fait l'objet de quelques études dans le genre Sulfolobus. Ce cycle a des caractères qui sont similaires à la fois des systèmes eucaryotes et bactériens. Selon les espèces d’archées, les chromosomes sont répliqués à partir de un ou plusieurs points de départ (origines de réplication) à l'aide d'ADN polymérases qui ressemblent aux enzymes équivalentes des eucaryotes[58]. Toutefois, les protéines de la division cellulaire, tels que la protéine FtsZ (Filamenting temperature-sensitive mutant Z), qui forme un anneau contractant autour de la cellule, et les composants de la cloison naissante dans le cœur de la cellule, sont similaires à leurs équivalents bactériens[57].
S’il existe des spores chez les bactéries et les eucaryotes, elles n’ont jamais été mises en évidence dans toutes les archées connues. Certaines espèces de Haloarchaea peuvent subir des modifications phénotypiques et croître avec différents types de cellules, incluant des parois épaisses. Ces structures qui sont résistantes aux chocs osmotiques (en) permettent aux archées de survivre dans l'eau à de faibles concentrations en sel, mais ce ne sont pas des structures de reproduction et elles ne peuvent aider à la dispersion dans de nouveaux habitats[59].
Diversité des archées, habitat
Caractéristiques cellulaires

Les archées sont très diverses, aussi bien d'un point de vue morphologique que physiologique. Ce sont des êtres unicellulaires avec une taille variant entre 0,1 et 15 µm, mais certains se développent pour former des filaments ou des agrégats (filaments jusqu'à 200 µm). Elles peuvent être sphériques (coques), spirales, en forme de bâtonnet (bacilles), rectangulaires…
Elles font preuve d'une grande diversité de modes de reproduction, par fission binaire, bourgeonnement ou fragmentation.
Métabolisme
D'un point de vue nutritionnel, elles se répartissent en de très nombreux groupes, depuis les chimiolithoautotrophes (tirant leur énergie de gradients chimiques d'origine non biologique) aux organotrophes.
D'un point de vue physiologique, elles peuvent être aérobies, anaérobies facultatives ou strictement anaérobies.
Habitat
Les archées existent dans une large diversité d'habitats et sont une composante importante des écosystèmes de la planète[12]. Elles peuvent contribuer jusqu'à 20 % des cellules microbiennes dans les océans[60]. De nombreuses archées sont extrêmophiles, et les milieux extrêmes étaient initialement considérés comme leur niche écologique[61]. En effet, certaines archées vivent à des températures élevées, souvent supérieures à 100 °C, que l'on rencontre dans les geysers, les fumeurs noirs et des puits de pétrole. D'autres se trouvent dans des habitats très froids et d'autres en milieu très salé, acide ou dans l'eau alcaline. Toutefois, d'autres espèces d’archées sont mésophiles et poussent dans des conditions beaucoup plus douces, dans les marais, les eaux usées, les océans et les sols[12].
Les archées extrêmophiles sont membres des quatre principaux groupes physiologiques. Ce sont les halophiles, thermophiles, alcalophiles et acidophiles[62]. Ces groupes n’ont pas de lien avec leur embranchement dans la classification phylogénétique. Néanmoins, ils sont un point de départ utile pour la classification.
Les halophiles, par exemple le genre Halobacterium, vivent dans des environnements salins, tels que les lacs salés (Grand Lac Salé de l’Utah), le littoral marin, les marais salants, la mer Morte, avec des concentrations en sel jusqu'à 25 %. Les membres de l'ordre des Halobacteriales (Haloferax, Halobacterium, Halococcus, Halorubrum, Natrinema, Natronococcus…) sont des exemples d’archées halophiles. Elles ont souvent une pigmentation rouge à jaune à cause des caroténoïdes et sont responsables de la coloration de certains lacs (Lac Magadi au Kenya par exemple). Les thermophiles se développent mieux à des températures supérieures à 45 °C, dans des lieux tels que les sources d'eau chaude ; les archées hyperthermophiles sont définies comme celles qui se développent au mieux à une température supérieure à 80 °C[63]. Pyrococcus, Methanopyrus, Thermococcus, Sulfolobus, Pyrodictium sont des exemples d’archées hyperthermophiles. Pyrobaculum provient de réservoirs profonds de pétrole chaud. Pyrolobus fumarii est capable de se multiplier jusqu'à 113 °C. Une étude récente a montré que la souche 116 de Methanopyrus kandleri pousse à 122 °C, ce qui est la température la plus élevée enregistrée à laquelle un organisme est encore capable de se développer[64]. D’autres archées peuvent croître dans des conditions très acides ou alcalines[62]. Par exemple, l'une des archées acidophiles les plus extrêmes est Picrophilus torridus, qui croît à un pH de 0, ce qui équivaut à 1,2 mole d'acide sulfurique[65].
Des études plus récentes ont montré que les archées existent non seulement dans les environnements mésophile et thermophile, mais également à basse température, parfois en grand nombre. Ainsi, les archées sont communes dans les environnements océaniques froids tels que les mers polaires[66]. Les archées sont en fait présentes en grand nombre dans tous les océans du monde dans la communauté planctonique (dans le cadre du picoplancton)[67]. Bien que ces archées puissent représenter jusqu'à 40 % de la biomasse microbienne, presque aucune de ces espèces n’a été isolée et étudiée en culture pure[68]. Par conséquent, notre compréhension du rôle des archées dans l'écologie des océans est rudimentaire, de sorte que leur influence sur les cycles biogéochimiques mondiaux reste largement inexplorée[69]. Certaines Crenarchaeota marines sont capables de nitrification, suggérant que ces organismes jouent un rôle important dans le cycle de l'azote océanique[70], bien qu’elles puissent également utiliser d'autres sources énergétiques[71]. Un grand nombre d’archées sont également présentes dans les sédiments qui recouvrent le fond de la mer et constitueraient la majorité des cellules vivantes à des profondeurs de plus d'un mètre dans ces sédiments[72] - [73]. Les archées méthanogènes (productrices de méthane) des marais sont responsables des gaz des marais (Poitevin par exemple). Beaucoup d’archées méthanogènes sont présentes dans le tube digestif des ruminants (Methanomicrobium, Methanosarcina), des termites ou des humains. Des études portant sur la faune nombrilienne (les micro-organismes vivant dans le nombril humain) ont démontré la présence d'archées à cet endroit[8].
Archées et santé humaine
Jusqu'à aujourd'hui, il n'y a pas de démonstration claire qu'il existe des archées pathogènes[74] - [75], bien que des relations aient été proposées entre la présence d'archées méthanogènes et de maladies parodontales[76].
Bien qu’un grand nombre d’archées ne soient aujourd’hui pas cultivables en laboratoire, de nombreuses espèces peuvent être cultivées en utilisant des milieux de culture adaptés et en reproduisant au mieux les conditions environnementales de leurs habitats naturels. Les effets des archées présentes dans le nombril humain n'ont pas encore été étudiés[8].
Comparaison entre archées, eubactéries et eucaryotes
Les archées sont semblables aux bactéries par beaucoup d’aspects de leur structure cellulaire et de leur métabolisme. Cependant, les mécanismes et les protéines impliqués dans les processus de réplication, de transcription et de traduction présentent des traits similaires à ceux rencontrés chez les eucaryotes. Les particularités des archées par rapport aux deux autres domaines du vivant (bactéries et eucaryotes) sont les suivantes :
- la structure et la chimie des parois cellulaires, atypiques (absence de peptidoglycane, classique chez les bactéries) ;
- la structure lipidique de leur membrane : les lipides des archées consistent en de longues chaînes d'alcool isopréniques attachées au glycérol par des liaisons éther, alors que les autres organismes fabriquent les lipides de leurs membranes en assemblant deux chaînes d'acides gras avec une molécule de glycérol par l'intermédiaire d'une liaison ester[77] ;
- les lipides de la membrane peuvent former des monocouches (formé de tétraéthers de glycérol) ou des bicouches (constitué d'ester de glycérols). Les lipides des eucaryotes et des eubactéries forment toujours des bicouches[77] ;
- la présence d'ARN polymérases inhabituelles, beaucoup plus complexes que les ARN-polymérases des bactéries, et étonnamment proches de celles des eucaryotes ;
- un chromosome circulaire de type bactérien mais comportant un génome mosaïque bactéries/eucaryotes ;
- les protéines intervenant dans les processus de réplication et de réparation de l'ADN ressemblent à celles rencontrées chez les eucaryotes[78] ;
- leur métabolisme (méthanogènes…).
Notes et références
- (en) Référence NCBI : Archaea (taxons inclus).
- (en) Emile Zuckerkand et Linus Pauling, « Molecules as documents of evolutionary history », Journal of Theoretical Biology, vol. 8, no 2, , p. 357-366 (lire en ligne) DOI 10.1016/0022-5193(65)90083-4.
- (en) Carl R. Woese et George E. Fox, « Phylogenetic structure of the prokaryotic domain: The primary kingdoms », PNAS, vol. 74, no 11, , p. 5088–5090 (DOI 10.1073/pnas.74.11.5088, lire en ligne).
- (en) William E. Balch, Linda J. Magrum, George E. Fox, Ralph S. Wolfe et Carl R. Woese, « An ancient divergence among the bacteria », Journal of Molecular Evolution, vol. 9, no 4, , p. 305-311 (DOI 10.1007/BF01796092, lire en ligne).
- (en) Norman R. Pace, « Concept Time for a change », Nature, vol. 441, , p. 289 (DOI 10.1038/441289a, lire en ligne).
- (en) Zhichao Zhou, Yang Liu et al., Two or three domains: a new view of tree of life in the genomics era, 2018 DOI 10.1007/s00253-018-8831-x
- Thi Thuy Hong Huynh, « Archaea et cavité orale », Thèse de doctorat en Pathologie humaine - Maladies infectieuses. Université d'Aix-Marseille. Sous la direction de Michel Drancourt et de Gérard Aboudharam., , p. 109 (lire en ligne).
- (en) J. Hulcr, A.M. Latimer, J.B. Henley et al., « A Jungle in There: Bacteria in Belly Buttons are Highly Diverse, but Predictable », PLOS One, vol. 7, , e47712 (lire en ligne).
- Querellou, J. (2010). Biotechnologie des archées. Biofutur, 29(310), 45-48.
- (en) Carl R. Woese, Otto Kandler et Mark L. Wheelis, « Towards a Natural System of Organisms: Proposal for the Domains Archaea, Bacteria, and Eucarya », PNAS, vol. 87, no 12, , p. 4576–4579 (lire en ligne).
- (en) Thomas Cavalier-Smith, « Only six kingdoms of life », Proc. R. Soc. Lond. B, vol. 271, , p. 1251-1262 (lire en ligne [PDF]).
- (en) DeLong EF, « Everything in Moderation : Archaea as 'Non-Extremophiles' », Curr. Opin. Genet. Dev., vol. 8, no 6, , p. 649–654 (PMID 9914204, DOI 10.1016/S0959-437X(98)80032-4).
- (en) Theron J, Cloete TE, « Molecular Techniques for Determining Microbial Diversity and Community Structure in Natural Environments », Crit. Rev. Microbiol., vol. 26, no 1, , p. 37–57 (PMID 10782339, DOI 10.1080/10408410091154174).
- (en) Schmidt TM, « The Maturing of Microbial Ecology », Int. Microbiol., vol. 9, no 3, , p. 217–223 (PMID 17061212, lire en ligne [PDF]).
- (en) D. Gevers, P. Dawyndt, P. Vandamme et al., « Stepping Stones Towards a New Prokaryotic Taxonomy », Philos. Trans. R. Soc. Lond., B, Biol. Sci., vol. 361, no 1475, , p. 1911–1916 (PMID 17062410, DOI 10.1098/rstb.2006.1915, lire en ligne).
- (en) Robertson CE, Harris JK, Spear JR, Pace NR, « Phylogenetic Diversity and Ecology of Environmental Archaea », Curr. Opin. Microbiol., vol. 8, no 6, , p. 638–642 (PMID 16236543).
- (en) Huber H, Hohn MJ, Rachel R, Fuchs T, Wimmer VC, Stetter KO., « A New Phylum of Archaea Represented by a Nanosized Hyperthermophilic Symbiont », Nature, vol. 417, no 6884, , p. 27–28 (PMID 11986665, DOI 10.1038/417063a).
- (en) Barns SM, Delwiche CF, Palmer JD, Pace NR, « Perspectives on Archaeal Diversity, Thermophily and Monophyly from Environmental rRNA Sequences », PNAS, vol. 93, no 17, , p. 9188–9193 (PMID 8799176, DOI 10.1073/pnas.93.17.9188, lire en ligne).
- (en) Elkins JG, Podar M, Graham DE et al., « A Korarchaeal Genome Reveals Insights Into the Evolution of the Archaea », PNAS, vol. 105, no 23, , p. 8102–8107 (PMID 18535141, DOI 10.1073/pnas.0801980105, lire en ligne).
- (en) Baker, B.J., Tyson, G.W., Webb, R.I., Flanagan, J., Hugenholtz, P. et Banfield, J.F., « Lineages of Acidophilic Archaea Revealed by Community Genomic Analysis. Science », Science, vol. 314, no 6884, , p. 1933–1935 (PMID 17185602, DOI 10.1126/science.1132690).
- (en) Lionel Guy et Thijs J.G. Ettema, « The archaeal ‘TACK’ superphylum and the origin of eukaryotes », Trends in Microbiology, vol. 19, no 10, , p. 580-587 (ISSN 0966-842X, DOI 10.1016/j.tim.2011.09.002, lire en ligne).
- Fabien Goubet, « Loki, vieille cousine venue du fond des mers », sur Le Temps, (consulté le ).
- « Une nouvelle organisation du vivant », sur Le Temps, (consulté le ).
- (en) Katarzyna Zaremba-Niedzwiedzka, Eva F. Caceres, Jimmy H. Saw, Disa Bäckström, Lina Juzokaite, Emmelien Vancaester, Kiley W. Seitz, Karthik Anantharaman, Piotr Starnawski, Kasper U. Kjeldsen, Matthew B. Stott, Takuro Nunoura, Jillian F. Banfield, Andreas Schramm, Brett J. Baker, Anja Spang et Thijs J. G. Ettema, « Asgard archaea illuminate the origin of eukaryotic cellular complexity », Nature, vol. 541, no 7637, , p. 353-358 (ISSN 0028-0836, DOI 10.1038/nature21031, lire en ligne).
- (en) Gregory P. Fournier et Anthony M. Poole, « A Briefly Argued Case That Asgard Archaea Are Part of the Eukaryote Tree », Frontiers in Microbiology, vol. 9, (ISSN 1664-302X, PMID 30158917, PMCID PMC6104171, DOI 10.3389/fmicb.2018.01896, lire en ligne, consulté le ).
- (en) bacterio.net.
- (en) Castelle & Banfield, 2018 : Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life. Cell, vol. 172, no 6, p. 1181-1197.
- (en) Tom A. Williams et al., Integrative modeling of gene and genome evolution roots the archaeal tree of life, publié en 2017 DOI 10.1073/pnas.1618463114
- (en) Castelle, C.J., Banfield, J.F., « Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life », Cell, vol. 172, no 6, , p. 1181-1197 (PMID 29522741, DOI 10.1016/j.cell.2018.02.016)
- (en) Kiley W. Seitz, Nina Dombrowski, Laura Eme, Anja Spang, Jonathan Lombard et al., « Asgard archaea capable of anaerobic hydrocarbon cycling », Nature Communications, vol. 10, , article no 1822 (DOI 10.1038/s41467-019-09364-x).
- (en) Ciccarelli FD, Doerks T, von Mering C, Creevey CJ, Snel B, Bork P, « Toward automatic reconstruction of a highly resolved tree of life », Science, vol. 311, no 5765, , p. 1283–1287 (PMID 16513982, DOI 10.1126/science.1123061).
- (en) Schopf J, « Fossil Evidence of Archaean Life »(Archive.org • Wikiwix • Archive.is • Google • Que faire ?) [PDF], (PMID 16754604, DOI 10.1098/rstb.2006.1834), p. 869–885.
- (en) Chappe B, Albrecht P, Michaelis W, « Polar Lipids of Archaebacteria in Sediments and Petroleums », Science, vol. 217, no 4554, , p. 65–66 (PMID 17739984, DOI 10.1126/science.217.4554.65).
- (en) Brocks JJ, Logan GA, Buick R, Summons RE, « Archean Molecular Fossils and the Early Rise of Eukaryotes », Science, vol. 285, no 5430, , p. 1033–1036 (PMID 10446042, DOI 10.1126/science.285.5430.1033).
- (en) Rasmussen B, Fletcher IR, Brocks JJ, Kilburn MR, « Reassessing the First Appearance of Eukaryotes and Cyanobacteria », Nature, vol. 455, no 7216, , p. 1101–1104 (PMID 18948954, DOI 10.1038/nature07381).
- (en) Jürgen Hahn et Pat Haug, « Traces of Archaebacteria in Ancient Sediments », System Applied Microbiology, vol. 7, no Archaebacteria '85 Proceedings, , p. 178–183.
- Orange F (2008) Fossilisation expérimentale de bactéries : appui à l'identification de signatures microbiologiques terrestres et extraterrestres (Doctoral dissertation, Université d'Orléans) résumé.
- (en) M.P. Bassez, Y. Takano et K. Kobayashi, « Prebiotic organic microstructures », Origins of Life and Evolution of Biosphères, vol. 42, no 4, , p. 307-316 (lire en ligne).
- (en) Woese CR, Gupta R, « Are Archaebacteria Merely Derived 'Prokaryotes'? », Nature, vol. 289, no 5793, , p. 95–96 (PMID 6161309, DOI 10.1038/289095a0).
- (en) Carl Woese, « The universal ancestor », PNAS, vol. 95, no 12, , p. 6854–6859 (PMID 9618502, DOI 10.1073/pnas.95.12.6854, lire en ligne).
- (en) Gupta RS, « The Natural Evolutionary Relationships Among Prokaryotes », Crit. Rev. Microbiol., vol. 26, no 2, , p. 111–131 (PMID 10890353, DOI 10.1080/10408410091154219).
- (en) Thomas Cavalier-Smith, « The neomuran origin of archaebacteria, the negibacterial root of the universal tree and bacterial megaclassification », International Journal of Systematic and Evolutionary Microbiology 52: 7–76 (2002).
- (en)Di Giulio, M. (2003). The Universal Ancestor Was a Thermophile or a Hyperthermophile: Tests and Further Evidence. J Theor Biol 221: 425-436.
- (en) Forterre, P., de la Tour, C. B., Philippe, H. et Duguet, M. (2000). Reverse Gyrase from Hyperthermophiles - Probable Transfer of a Thermoadaptation Trait from Archaea to Bacteria. Trends in Genetics 16: 152-154.
- (en) Gribaldo S, Brochier-Armanet C, « The Origin and Evolution of Archaea: a State of the Art »(Archive.org • Wikiwix • Archive.is • Google • Que faire ?), (PMID 16754611, DOI 10.1098/rstb.2006.1841), p. 1007–1022.
- (en) Woese CR, « There Must Be a Prokaryote Somewhere: Microbiology's Search for Itself », Microbiol. Rev., vol. 58, no 1, , p. 1–9 (PMID 8177167, PMCID 372949, lire en ligne).
- Pierre Vauclare, Les Archaea et l’origine de la vie [Rapport de recherche] CNRS-UMR 5075. 2013.
- (en) Nelson KE, Clayton RA, Gill SR et al., « Evidence for Lateral Gene Transfer Between Archaea and Bacteria from Genome Sequence of Thermotoga Maritima », Nature, vol. 399, no 6734, , p. 323–329 (PMID 10360571, DOI 10.1038/20601).
- (en) James A. Lake, « Origin of the Eukaryotic Nucleus Determined by Rate-Invariant Analysis of rRNA Sequences », Nature, vol. 331, no 6152, , p. 184–186 (PMID 3340165, DOI 10.1038/331184a0).
- (en) Laura Eme et Thijs J. G. Ettema, « The eukaryotic ancestor shapes up », Nature, (DOI 10.1038/d41586-018-06868-2).
- (en) Caner Akıl et Robert C. Robinson, « Genomes of Asgard archaea encode profilins that regulate actin », Nature, vol. 562, , p. 439-443 (DOI 10.1038/s41586-018-0548-6).
- (en) D. Yang, Y. Oyaizu, H. Oyaizu, G.J. Olsen et C.R. Woese, « Mitochondrial origins », PNAS, vol. 82, no 13, , p. 4443-4447 (ISSN 0027-8424, PMCID 391117, résumé, lire en ligne [PDF]).
- (en) Galagan JE et al., « The Genome of M. Acetivorans Reveals Extensive Metabolic and Physiological Diversity », Genome Research, vol. 12, no 4, , p. 532–542 (ISSN 1088-9051, DOI 10.1101/gr.223902, lire en ligne).
- (en) Waters E et al., « The Genome of Nanoarchaeum Equitans: Insights Into Early Archaeal Evolution and Derived Parasitism », Proc. Natl. Acad. Sci. U.S.A., vol. 100, no 22, , p. 12984–12988 (PMID 14566062, DOI 10.1073/pnas.1735403100, lire en ligne).
- (en) Schleper C, Holz I, Janekovic D, Murphy J, Zillig W, « A Multicopy Plasmid of the Extremely Thermophilic Archaeon Sulfolobus Effects Its Transfer to Recipients by Mating », J. Bacteriol., vol. 177, no 15, , p. 4417–4426 (PMID 7635827, lire en ligne).
- (en) Sota M; Top EM, Plasmids: Current Research and Future Trends, Norfolk, Caister Academic Press, (ISBN 978-1-904455-35-6, LCCN 2008411779, lire en ligne), « Horizontal Gene Transfer Mediated by Plasmids ».
- (en) Bernander R, « Archaea and the cell cycle », Mol. Microbiol., vol. 29, no 4, , p. 955–961 (PMID 9767564, DOI 10.1046/j.1365-2958.1998.00956.x).
- (en) Kelman LM, Kelman Z, « Multiple origins of replication in archaea », Trends Microbiol., vol. 12, no 9, , p. 399–401 (PMID 153371581, DOI 10.1016/j.tim.2004.07.001).
- (en) Kostrikina NA, Zvyagintseva IS, Duda VI., « Cytological Peculiarities of Some Extremely Halophilic Soil Archaeobacteria », Arch. Microbiol., vol. 156, no 5, , p. 344–349 (DOI 10.1007/BF00248708).
- (en) E.F. DeLong et N.R. Pace, « Environmental diversity of bacteria and archaea », Syst. Biol., vol. 50, no 4, , p. 470–478 (PMID 12116647, DOI 10.1080/106351501750435040).
- Simonetta Gribaldo, Patrick Forterre et Céline Brochier-Armanet : « Les Archaea : évolution et diversité du troisième domaine du vivant[PDF] », Bulletin de la Société Française de Microbiologie, vol. 23, no 3, 2008, p. 137–145.
- (en) Pikuta EV, Hoover RB, Tang J, « Microbial Extremophiles at the Limits of Life », Crit. Rev. Microbiol., vol. 33, no 3, , p. 183–209 (PMID 17653987, DOI 10.1080/10408410701451948).
- (en) Madigan MT, Martino JM, Brock Biology of Microorganisms, Upper Saddle River, Pearson, , 11th ed. éd., poche (ISBN 978-0-13-196893-6, LCCN 2004026995), p. 136.
- (en) Takai K, Nakamura K, Toki T, Tsunogai U, Miyazaki M, Miyazaki J, Hirayama H, Nakagawa S, Nunoura T, Horikoshi K, « Cell Proliferation at 122 °C and Isotopically Heavy CH4 Production by a Hyperthermophilic Methanogen Under High-Pressure Cultivation », Proc Natl Acad Sci U S A, vol. 105, , p. 10949–10954 (DOI 10.1073/pnas.0712334105).
- (en) Ciaramella M, Napoli A, Rossi M, « Another extreme genome: how to live at pH 0 », Trends Microbiol., vol. 13, no 2, , p. 49–51 (PMID 15680761, DOI 10.1016/j.tim.2004.12.001).
- (en) López-García P, López-López A, Moreira D, Rodríguez-Valera F, « Diversity of Free-Living Prokaryotes from a Deep-Sea Site at the Antarctic Polar Front », FEMS Microbiol. Ecol., vol. 36, nos 2–3, , p. 193–202 (PMID 11451524).
- (en) Karner MB, DeLong EF, Karl DM, « Archaeal Dominance in the Mesopelagic Zone of the Pacific Ocean », Nature, vol. 409, no 6819, , p. 507–510 (PMID 11206545, DOI 10.1038/35054051).
- (en) Giovannoni SJ, Stingl U., « Molecular Diversity and Ecology of Microbial Plankton », Nature, vol. 427, no 7057, , p. 343–348 (PMID 16163344, DOI 10.1038/nature04158).
- (en) DeLong EF, Karl DM, « Genomic perspectives in microbial oceanography », Nature, vol. 437, no 7057, , p. 336–342 (PMID 16163343, DOI 10.1038/nature04157).
- (en) Konneke M, Bernhard AE, de la Torre JR, Walker CB, Waterbury JB, Stahl DA., « Isolation of an Autotrophic Ammonia-Oxidizing Marine Archaeon », Nature, vol. 437, no 7057, , p. 543–546 (PMID 16177789, DOI 10.1038/nature03911).
- (en) Agogué H, Maaike B, Dinasquet J, Herndl GJ., « Major Gradients in Putatively Nitrifying and Non-Nitrifying Archaea in the Deep North Atlantic », Nature, vol. 456, no 7223, , p. 788-791 (DOI 10.1038/nature07535).
- (en) Teske A, Sørensen KB, « Uncultured Archaea in Deep Marine Subsurface Sediments: Have We Caught Them All? », ISME J, vol. 2, no 1, , p. 3–18 (PMID 18180743, DOI 10.1038/ismej.2007.90).
- (en) Lipp JS, Morono Y, Inagaki F, Hinrichs KU, « Significant Contribution of Archaea to Extant Biomass in Marine Subsurface Sediments », Nature, (PMID 18641632, DOI 10.1038/nature07174).
- (en)Eckburg P, Lepp P, Relman D (2003). "Archaea and Their Potential Role in Human Disease". Infect Immun 71 (2): 591-6.
- (en)Cavicchioli R, Curmi P, Saunders N, Thomas T (2003). "Pathogenic Archaea: Do They Exist?". Bioessays 25 (11): 1119-28.
- (en)Lepp P, Brinig M, Ouverney C, Palm K, Armitage G, Relman D (2004). "Methanogenic Archaea and Human Periodontal Disease". Proc Natl Acad Sci USA 101 (16): 6176-81.
- Guillaume Lecointre et Hervé Le Guyader, Classification phylogénétique du vivant, t. 1, 4e édition, Paris, Belin, , 583 p. (ISBN 978-2-7011-8294-0, BNF 45150617), p. 73-74.
- (en)Kelman, Z. (2000) DNA replication in the third domain (of life). Curr Protein Pept Sci 1: 139-154.
Voir aussi
Articles connexes
Liens externes
- Ressources relatives au vivant :
- Ressource relative à la santé :
- Notices dans des dictionnaires ou encyclopédies généralistes :
- (en) Référence Catalogue of Life : Archaea (consulté le )
- (fr+en) Référence ITIS : Archaea (consulté le )
- (en) Référence NCBI : Archaea (taxons inclus) (consulté le )
- (en) Référence Tree of Life Web Project : Archaea (consulté le )
- (en) Référence uBio : site déclaré ici indisponible le 7 avril 2023
- (en) Référence World Register of Marine Species : taxon Archaea Woese, Kandler & Wheelis, 1990 (+ liste embranchements + liste classes) (consulté le )
- Lifemap NCBI : consulter en ligne : arbre de vie dynamique
