SARS-CoV-2
Le SARS-CoV-2 (acronyme anglais de severe acute respiratory syndrome coronavirus 2), soit coronavirus 2 du syndrome respiratoire aigu sévère, est le virus responsable de la Covid-19. Son acronyme est parfois partiellement francisé en SRAS-CoV-2[Note 1] - [Note 2]. Ce coronavirus hautement pathogène a été découvert en dans la ville de Wuhan (province de Hubei, en Chine).
Le SARS-CoV-2 est un virus à ARN monocaténaire de polarité positive du groupe IV de la classification Baltimore. C'est une souche nouvelle de l'espèce SARSr-CoV, dans le genre betacoronavirus qui regroupe entre autres les SARS-CoV-1 et MERS-CoV.
.jpg.webp)
Dans un contexte de faible immunité collective, le SARS-CoV-2 circule presque toute l'année. Avec le temps, il est possible que le SARS-CoV-2 circule sur un mode épidémique plus saisonnier, entre janvier et mai dans les zones à climat tempéré, tout comme le HCoV-NL63, le HCoV-229E et le HCoV-OC43, des coronavirus responsables de simples rhumes.
Le SARS-CoV-2 est principalement transmis par les microgouttelettes et aérosols et a un tropisme particulier pour le système respiratoire supérieur (nez, trachée) et inférieur (bronches, poumons). Un site complémentaire de réplication primaire est le système digestif, en particulier l’estomac et les intestins. Le SARS-CoV-2 peut se disséminer dans l’organisme via les neurones. Lorsque le SARS-CoV-2 atteint le système nerveux central, il peut se produire une perte totale ou partielle d’odorat (anosmie). Le SARS-CoV-2 a de nombreux sites secondaires de réplication : le système cardiovasculaire, le système immunitaire, le système endocrinien, le système urinaire, le système reproducteur et les glandes sudoripares de la peau.
Le principal récepteur cellulaire utilisé par le SARS-CoV-2 pour infecter des cellules est l’enzyme ACE2. Ce récepteur est reconnu par la protéine S du SARS-CoV-2 qui opère l'essentiel du processus d’entrée du virus dans une cellule. Le SARS-CoV-2 possède au total environ 29 protéines virales. Certaines d’entre elles sont spécialisées dans le détournement de la machinerie de la cellule infectée. D’autres participent activement à la réplication du génome viral. Le SARS-CoV-2 peut infecter des cellules par fusion directe ou en étant absorbé par une cellule via un processus d’endocytose. Le SARS-CoV-2 a aussi la faculté de fusionner des cellules infectées avec les cellules non infectées avoisinantes, formant des « syncytia », c’est-à-dire des cellules géantes englobant des dizaines de cellules productrices de virus.
Présentant une vitesse actuelle de mutation de l’ordre de 5,2 à 8,1 × 10−3 substitutions par site et par an, le SARS-CoV-2 est l'un des virus qui mutent le plus vite au monde.
La réponse immunitaire face au SARS-CoV-2 diffère d'un patient à l'autre : 40 % sont asymptomatiques, 40 % développent une Covid légère, 15 % une forme modérée pouvant conduire à un Covid long, et 5 % une Covid sévère pouvant nécessiter des soins de réanimation. La réponse immunitaire innée et adaptative des formes sévères de Covid-19 est globalement contre-productive et génère autant de dégâts dans l'organisme que le virus.
L'origine du SARS-CoV-2 n'est pas résolue. Le réservoir animal des sarbecovirus se trouve chez les chauve-souris asiatiques du genre Rhinolophus (les rhinolophes). Son adaptation à l'humain pourrait résulter d’un passage direct des chauves-souris aux humains, mais il est également possible qu'elle se soit faite par une transmission impliquant un hôte intermédiaire encore indéterminé, ou soit le fruit d'un gain de fonction en laboratoire[2].
Coronavirus 2 du syndrome respiratoire aigu sévère
Virologie
Classification
Le virus SARS-CoV-2 forme une souche virale génétiquement distincte des autres coronavirus humains comme le MERS-CoV ou d'autres plus bénins, mais il appartenant à la même espèce biologique que le SARS-CoV-1, le virus du SRAS[4]: comme celui-ci il appartient à l'espèce SARSr-CoV (severe acute respiratory syndrom-related coronavirus), dans le sous-genre Sarbecovirus[5] - [6] - [7], le genre Betacoronavirus et la famille Coronaviridae[4].
Morphologie et génome
La morphologie des virions est typique de celle des coronavirus[8], notamment par son halo de protubérances constituées de polymères de protéines virales spiculées (« spikes » en anglais), qui leur a donné leur nom de « virus à couronne ». Le diamètre du virion de SARS-CoV-2 est compris entre 60 et 140 nm[9], contre 50 à 80 nm pour celui du SARS-CoV-1[10] - [8] - [Note 3].
Le génome du SARS-CoV-2 est constitué d'un ARN simple-brin de 29 903 nucléotides[11] - [12] - [13]. En ce qui concerne l'homologie des nucléotides, le SARS-CoV-2 est à 79,5 % identique à celui du SARS-CoV[14] et à 50 % identique à celui du MERS-CoV[15].
Filiation génétique
Les sarbecovirus ont pour réservoir animal les chauves-souris du genre Rhinolophus, les rhinolophes.
Le SARS-CoV-2 présente des similitudes avec les Betacoronavirus trouvés chez des chauves-souris et des pangolins[16] - [17].
L'arbre phylogénétique de la branche des coronavirus apparentés au SARS-CoV-2, tel qu'obtenu sur la base du gène RdRp, est le suivant[18] - [19] - [20] :
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
SARS-CoV-1, proche à 79 % du SARS-CoV-2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
- Animal hôte :
- chauve-souris
- pangolin
- humain
En matière d'homologie, le SARS-CoV-2 est :
- à 96 % identique à celui de BetaCoV/bat/Yunnan/RaTG13/2013, un coronavirus d'une chauve-souris chinoise du genre Rhinolophe : Rhinolophus affinis[27] - [28] et à près de 97 % de BANAL-52 découvert récemment au Laos[29] - [30] ;
- à 91 % identique à un coronavirus présent chez les pangolins javanais[31] - [32] - [33] - [34] - [35] - [36].
Le SARS-CoV-2 est très proche de la souche RaTG13 (un sarbecovirus qui pourrait s'être adapté aux rongeurs[37] - [38]). Mais sa protéine S en est très différente. Le domaine de liaison au récepteur (RBD) du SARS-CoV-2 est en revanche très proche de celui de BANAL-52.
Mais le SARS-CoV-2 présente aussi deux innovations déterminantes pour son adaptation à l'humain :
- La mutation T372A dans le RBD facilite la liaison au récepteur ACE2 humain[39] (mais aussi du pangolin et de R. macrotis (it)), on la trouve dans tous les SARS-CoV-2 et seulement chez eux. Ce qui pourrait en partie expliquer pourquoi la protéine S du SARS-CoV-2 a une affinité pour le récepteur ACE2 humain 10 à 20 fois plus élevée que celle du virus du SARS-CoV-1[40] - [41].
- L'insertion d'une séquence de 4 acides aminés -S[PRRA]RSV- au site de clivage (-RS-) affichant une séquence -RRAR- qui permet chez l'humain l'activation du clivage par la furine[42]. De telles séquences se retrouvent dans d'autres coronavirus humains mais pas chez les sarbecovirus, SARS-CoV-1 inclus (Le MERS-CoV, un merbecovirus humain, a pour sa part une séquence différente -PRSVRSV-).
Description
Structure protéique du virus
Comme d'autres coronavirus, le SARS-CoV-2 possède quatre protéines structurales :
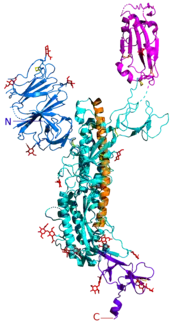
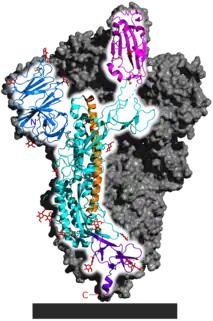
- Protéine S (dite protéine spike, spicule ou protéine spiculaire) : elle forme les péplomères, protubérances de la « couronne » caractéristique des coronavirus. Ces péplomères jouent un rôle clé dans la liaison du virus à un ou plusieurs récepteurs en surface d'une cellule[43]. La protéine S semble être l'un des principaux déterminants du tropisme du SARS-CoV-2[44].
La publication du génome viral a permis de modéliser sa structure tridimensionnelle (voir ci-contre). Elle a aussi été décrite au niveau atomique par la microscopie électronique cryogénique[41] - [45] ; - Protéine E (enveloppe) ;
- Protéine M (membrane) ;
- Protéine N (nucléocapside) ; c'est elle qui enveloppe et protège l'ARN viral (le code génétique du virus).
Les protéines S, E et M constituent, ensemble, l'enveloppe virale[43].
Génome
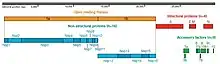
Le génome du SARS-CoV-2 contient 11 gènes[46] reconnaissant 15 Open Reading Frame (ORF) permettant de produire entre 29 et 33 protéines virales après protéolyse. Initialement 29 protéines virales avaient été identifiées[47]. Fin 2020 au moins 4 autres protéines virales supplémentaires ont été proposées (ORF2b, ORF3c, ORF3d et ORF3d-2)[48].
Le génome du SARS-CoV-2 a une coiffe à son extrémité 5' et une queue polyadénylée à son extrémité 3'[49] :
- à son extrémité 5', sont placés les gènes ORF1a et ORF1b codant des protéines non structurales (NSP). ORF1a et ORF1b représentent deux tiers du génome et se chevauchent légèrement et sont donc parfois confondus et appelés ORF1ab. ORF1ab est considéré comme un gène unique codant 16 protéines dont plusieurs sont des enzymes qui ont un rôle essentiel dans la réplication et l’expression du génome[50] ;
- à son extrémité 3', sont placés les dix gènes qui codent les protéines structurales et accessoires. Le SARS-CoV-2 a quatre gènes spécifiques à des protéines structurales : S, M, E et N. Le gène de la protéine N intègre également les ORF permettant de produire deux protéines accessoires : 9b et 9c[46]. En plus, le SARS-CoV-2 a six gènes associés à des protéines accessoires : 3a, 6, 7a, 7b, 8, et 10. Le gène de l'ORF3a code également l'essentiel de la protéine ORF3b[51] - [52] et de 3 autres protéines virales (ORF3c, ORF3d et ORF3d-2). Il est également suggéré que l'ORF codant la protéine S produit une seconde protéine baptisée ORF2b[48].
Les protéines accessoires du SARS-CoV-2 divergent en partie de celles du SARS-CoV-1[46] :
- les SARS-CoV-2 et SARS-CoV-1 ont en commun les protéines 3a, 6, 7a, 7b, 9b et 9c ;
- les protéines 3b et 8 du SARS-CoV-2 sont différentes des protéines 3b, 8a et 8b du SARS-CoV-1. Les protéines ORF3b et ORF8 seraient avec la protéine N, les premières cibles des anticorps produits par les lymphocytes B après une infection au SARS-CoV-2. Et ce bien avant que des anticorps ne soient produits contre des fragments de la protéine S. Ce qui laisse à penser que les protéines ORF3b et ORF8 sont d’une grande importance dans la pathogénèse[53] ;
- le SARS-CoV-2 aurait 5 protéines accessoires inédites : 10, 2b, 3c, 3d et 3d-2[48].
| ORF | Nucléotide début | Nucléotide fin | Nombre de codons | Protéine |
|---|---|---|---|---|
| ORF1a | 266 | 13 483 | 4 405 | Polyprotéines Pp1a et Pp1ab |
| ORF1b | 13 484 | 21 555 | 2 691 | Polyprotéine Pp1ab |
| ORF2 | 21 563 | 25 384 | 1 273 | Péplomère (protéine S) + ORF2b |
| ORF2b[48] | 21 744 | 21 860 | 39 | La protéine ORF2b est localisée aux codons 60 à 99 de la protéine S - protéine inédite |
| ORF3a | 25 393 | 26 220 | 275 | Protéines ORF3a + ORF3c + ORF3d + ORF3d-2 + ORF3b |
| ORF3c[48] | 25 457 | 25 579 | 41 | La protéine ORF3c est localisée aux codons 21 à 62 de ORF3a - protéine inédite |
| ORF3d[48] | 25 524 | 25 694 | 57 | La protéine ORF3d est localisée aux codons 43 à 100 de ORF3a - protéine inédite |
| ORF3d-2[48] | 25 596 | 25 694 | 33 | La protéine ORF3d-2 est localisée aux codons 67 à 100 de ORF3a - protéine inédite |
| ORF3b | 25 814 | 26 281[51] | 156 | ORF3b est à cheval sur ORF3a et ORF4. ORF3b est initialement tronquée et a 4 paliers d'évolution (4 codons STOP). Stade 1 initial : ORF3b1 de 22 codons (localisée aux codons 141-163 d’ORF3a). Stade 2 : ORF3b1+2 de 56 codons (localisée aux codons 141-197 d’ORF3a). Stade 3 : ORF3b1+2+3 de 78 codons (localisée aux codons 141-219 d’ORF3a). Stade 4 : ORF3b1+2+3+4 de 156 codons (localisée au codon 141 d’ORF3a jusqu’au codon 13 d’ORF4)[51] |
| ORF4 | 26 245 | 26 472 | 75 | Protéine d'enveloppe (protéine E) |
| ORF5 | 26 523 | 27 191 | 222 | Protéine membranaire (protéine M) |
| ORF6 | 27 202 | 27 387 | 61 | Protéine ORF6 |
| ORF7a | 27 394 | 27 759 | 121 | Protéine ORF7a |
| ORF7b | 27 756 | 27 887 | 43 | Protéine ORF7b |
| ORF8 | 27 894 | 28 259 | 121 | Protéine ORF8 |
| ORF9 | 28 274 | 29 533 | 419 | Phosphoprotéine de capside (protéine N) + Protéine ORF9b + Protéine ORF9c |
| ORF9b | 28 284 | 28 572 | 97 | ORF9b correspond aux codons 4 à 101 de l’ORF9 |
| ORF9c | 28 729 | 28 950 | 73 | ORF9c correspond aux codons 152 à 225 de l’ORF9 |
| ORF10 | 29 558 | 29 674 | 38 | Protéine ORF10 - protéine inédite |
Cycle de réplication
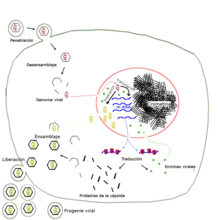
Comme c'est généralement le cas des virus, le cycle de réplication du SARS-CoV-2 se traduit par (1) l’entrée du virus à l’intérieur de la cellule, (2) le détournement de la machinerie cellulaire par le virus, (3) la réplication du virus par la cellule et/ou la réalisation de taches bien précises par la cellule répondant aux besoins du virus.
Entrée dans une cellule
La protéine S du SARS-CoV-2 est le principal acteur dans l'infection des cellules. La protéine S est constituée de deux sous-unités fonctionnelles : la sous-unité S1 permet la liaison du virus au récepteur de la cellule hôte et la sous-unité S2 assure la fusion de l’enveloppe virale avec la membrane cellulaire[54]. La sous-unité S1 contient deux domaines (le RBD et le NTD) qui chacun reconnaît des récepteurs cellulaires différents. Le principal domaine utilisé jusqu'à présent par le SARS-CoV-2 est le RBD. Le RBD permet la liaison au récepteur ACE2[55] - [56] - [57]. Le NTD est un domaine secondaire. Le NTD permet au SARS-CoV-2 de s'attacher aux récepteurs L-SIGN (en) s'exprimant fortement dans les poumons, le foie, les reins et les ganglions lymphatiques, ou aux récepteurs DC-SIGN (en), répandus dans les cellules dendritiques, matures ou non[58] - [59].
Une fois que la sous-unité S1 est liée à un récepteur (le plus souvent ACE2), deux clivages protéolytiques successifs sont nécessaires. Le premier clivage coupe la protéine S en S1 et S2. Le second appelé S2' libère l'extrémité du peptide de fusion (FP) qui comme son nom l'indique amorce la fusion du virion avec la cellule. Une des originalités de la protéine S du SARS-CoV-2 est qu’elle intègre une séquence d'activation atypique pour un coronavirus[60] au site de clivage S1 / S2, similaire aux séquences observées dans les virus de grippe. Ainsi, à l’instar du virus de la grippe, la protéine S du SARS-CoV-2 peut être clivée à l’aide d’une protéase qui se trouve en abondance dans le plasma humain : la furine. Les scientifiques suggèrent que cette capacité à pouvoir effectuer le clivage S1 / S2 par la furine est la raison pour laquelle le SARS-CoV-2 a causé une épidémie humaine[61].
Détournement de la cellule
Concrètement la fusion avec une cellule consiste en l'introduction du génome viral avec la protéine N du SARS-CoV-2 dans la cellule. Une fois dans la cellule, la protéine N bloque la production d’interféron[62] - [63]. Les interférons sont des molécules de signalisation qui permettent d'alerter les cellules voisines qu'une infection est en cours[64]. Dans le même temps, l’ARN viral est traduit par les ribosomes de la cellule hôte qui se mettent à produire des protéines virales[65] - [52].
Une fois synthétisées par les ribosomes, une grande partie des 16 protéines non structurales du SARS-CoV-2 s'assemblent pour former le Complexe Réplicase-Transcriptase (CRT) indispensable à la réplication en série du génome viral. D'autres protéines non structurales sont en charge de prendre le contrôle de la machinerie traductionnelle de la cellule et d'éteindre la traduction « normale » qui jusque là fonctionne, empêchant ainsi la synthèse de protéines interférons par les ribosomes. Nsp1 se fixe ainsi au ribosome de la cellule hôte et bloque tous les ARN messagers de la cellule, sauf ceux liés au génome du virus[66]. D'autres protéines virales sont plus ou moins spécialisées dans l'inhibition des interférons : nsp3[67], ORF3b[51], ORF6[68] - [69] - [70], ORF9b[71] - [72] et ORF9c[73] - [74] - [75]. La protéine du SARS-CoV-2 ayant la plus forte activité anti-interféron semble être ORF3b. Et il est suggéré qu'au gré des mutations à venir, la protéine ORF3b pourrait progressivement exercer une activité anti-interféron de plus en plus forte[51].
Des protéines virales vont aussi exercer une activité empêchant la synthèse d'un antigène. Un antigène est une molécule permettant aux globules blancs de reconnaître un pathogène et de lutter contre en mobilisant la mémoire immunitaire. Les protéines virales ORF8[76] - [77] et ORF3a[78] empêchent la synthèse d'un antigène par la cellule infectée. Tandis que la protéine S active des récepteurs sur la cellule infectée (récepteurs HLA-E) qui protègent la cellule infectée contre une destruction par les lymphocytes NK[79] - [80].
Production des virions
Après avoir pris le contrôle de la cellule infectée, et alors que le Complexe Réplicase-Transcriptase (CRT) réplique en série le génome viral, les ribosomes sont mobilisés pour produire en série des protéines virales structurales. Ces protéines s’assemblent entre elles dans le lumen (l’intérieur) d’un compartiment dérivé du réticulum endoplasmique[81]. Cette étape est appelée le bourgeonnement[81]. D’abord, une protéine N (nucléocapside) se fixe à une copie d'ARN et l’empaquette dans une protéine M (membrane) qui donne sa forme au virion[81]. Ensuite des protéines S sont incorporées[81]. La protéine M dirige la plupart des interactions protéine-protéine nécessaires à l'assemblage des virus après sa liaison à la nucléocapside[82]. La protéine E contribue à l’assemblage et à la libération du virion hors de la cellule infectée, en suivant la voie de sécrétion (appareil de Golgi, puis vésicules sécrétoires)[52]. Le virion quitte le milieu intracellulaire par exocytose, et est prêt à infecter une autre cellule.
Syncytia
Dans des organes tels que les poumons, les reins ou le foie, les cellules épithéliales sont étroitement compactées, l’espace extracellulaire est très limité. Le SARS-CoV-2 y tire un avantage. Les cellules infectées peuvent fusionner avec les cellules non infectées avoisinantes, formant des « syncytia », c’est-à-dire des cellules géantes englobant des dizaines de cellules productrices de virus[83]. Ce processus est médié par la protéine S nouvellement synthétisée qui s'accumule à la surface de la cellule infectée. Mais la formation de syncytia peut se faire aussi par « fusion from without (FFWO) », c’est-à-dire par des cellules infectées qui n’ont pas encore répliqué la protéine S[84].
Ce mode d’infection permet au SARS-CoV-2 d’infecter vite un grand nombre de cellules avec une faible quantité de protéines S. Lorsque le SARS-CoV-2 infecte de nouvelles cellules au sein d’un syncytium, les anticorps se révèlent d’une faible efficacité[84]. En revanche, IFITM (en), (interferon-inducible transmembrane protein), une protéine transmembranaire produite en présence d'interférons, peut bloquer cette fusion. Mais son effet peut être contrecarré par TMPRSS2 qui facilite la formation de syncytia[83].
Primaire
Les voies respiratoires supérieures (nez, pharynx) et inférieures (bronches, poumons) sont le principal site de réplication primaire. Un site complémentaire de réplication primaire est le système digestif, en particulier l’estomac et les intestins. Des particules du SARS-CoV-2 sont facilement détectées dans les selles[85], ce qui a permis de suivre le développement de l'épidémie via des analyses d'eaux d'égouts[86]. Le SARS-CoV-2 se réplique néanmoins moins bien dans le système digestif que dans le système respiratoire[87]. L’infection du système digestif se fait probablement par auto-infection, c'est-à-dire par absorption de mucus nasal infecté.
Secondaire
Les sites de réplication secondaires du SARS-CoV-1 dans les formes sévères sont le cœur, les glandes sudoripares de la peau, les reins et le système endocrinien qui régule les hormones (glandes surrénales, la parathyroïde et l’hypophyse)[88] - [89]. Pour le SARS-CoV-2, les formes sévères (et modérées[90] ?), sont susceptibles d’infecter ces mêmes organes, mais également :
- le système immunitaire : le SARS-CoV-2 peut se répliquer dans le système lymphatique au niveau de la bouche (tonsille palatine), du cou (cervical lymph nodes[90]), des poumons (hilar lymph nodes[91]) et des artères (paraaort. lymph nodes)[87]. Le SARS-CoV-2 peut infecter la rate qui est un organe de stockage de globules rouges et de lymphocytes[91] - [92] - [93]. Le SARS-CoV-2 est également capable de se répliquer dans l’appendice qui fabrique des lymphocytes pour les intestins[87] - [94] ;
- le système nerveux central : le SARS-CoV-2 peut se répliquer dans la moëlle osseuse (colonne vertébrale) qui est le lieu de production des globules blancs[87]. Le SARS-CoV-2 peut également se reproduire dans le cerveau. Il a été détecté dans le lobe frontal, l’hippocampe, le cervelet et le pont de Varole[87] - [95]. Du fait de leur neurotropisme, les coronavirus humains peuvent sur des sujets ayant une prédisposition génétique, contribuer au développement ou à l’aggravation de maladies neurologiques comme la sclérose en plaques, la maladie d’Alzheimer, ou encore des encéphalites récurrentes[96] ;
- le système génital : le SARS-CoV-2 peut migrer et se reproduire dans les testicules et l’utérus[87], alors que le SARS-CoV-1 en était incapable[88] - [89].
Globules blancs
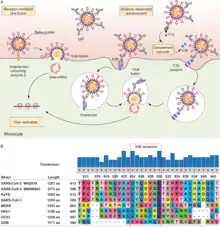
Le SARS-CoV-2 est capable de se répliquer dans certains globules blancs. In vitro, face à des globules blancs, le SARS-CoV-2 infecte en priorité les monocytes (44,3 %), les lymphocytes T CD4+ (14,2 %), les lymphocytes T CD8 (13,5 %) et les lymphocytes B (7,58 %). In vivo, l’infection des globules blancs par le SARS-CoV-2 est différente. Dans le cas de Covid sévère, des monocytes et lymphocytes B peuvent être infectés et, dans une moindre mesure, des lymphocytes T[97] - [98].
Des virus tels que les coronavirus peuvent infecter les globules blancs, par un mécanisme connu sous le nom de facilitation dépendante des anticorps (ADE), comme cela a été mis en évidence avec le SARS-CoV-1 et le MERS-CoV[99]. Même si aucun cas d'une telle ADE n'a pu être constaté pour l'instant avec le SARS-CoV-2, ce risque incite à la prudence tant dans la recherche vaccinale que pour les traitements expérimentaux à base de sérums de patients infectés ou d'anticorps monoclonaux[100].
Dans le cas du SARS-CoV-1, la séquence activant les anticorps facilitants est l’épitope peptidique « LYQDVNC » localisé sur le fragment « SD2 » de la protéine S. Cette séquence est présente dans la souche originelle du SARS-CoV-2 (celle de Wuhan) et correspond aux codons de la protéine S : L611, Y612, Q613, D614, V615, N616, C617[99]. La mutation D614G sur la protéine S, qui est devenue dominante parmi les variants du SARS-CoV-2, a transformé la séquence « LYQDVNC » en « LYQGVNC », et semblerait empêcher le phénomène d'anticorps facilitants[101].
D'autres mécanismes pouvant générer des anticorps facilitants ont été proposés pour le SARS-CoV-2. Certains anticorps ciblant le RBD de la protéine S du SARS-CoV-2 seraient susceptibles d'activer le récepteur FcγRIIB[102], reconnu par les lymphocytes B. Tandis que certains anticorps ciblant le NTD[103] - [104] - [105], pourraient activer :
- le récepteur FcγRIIa reconnu par les macrophages, les neutrophiles, les eosinophiles, les thrombocytes, les cellules de Langerhans ;
- le récepteur FcγRIIIa reconnu par les lymphocytes NK et les macrophages.
Chez le coronavirus du chat, un des rares coronavirus capable de se reproduire activement dans un globule blanc (les macrophages), le phénomène des anticorps facilitants induit une péritonite infectieuse (PIF). À noter que pour soigner la PIF, le GS-441524, autre nom du remdesivir, s’est montré efficace[106].
Dissémination
Après s’être répliqué localement au niveau des voies respiratoires et éventuellement dans le système digestif, le SARS-CoV-2 peut se disséminer dans l'organisme via 3 voies :
- via le transport axonal c'est-à-dire les neurones. Le SARS-CoV-2 peut infecter le nerf phrénique[87] qui est le nerf du diaphragme, le muscle principal de la ventilation pulmonaire, permettant l'inspiration. Et dans un modèle animal hamster, des antigènes du SARS-CoV-2 ont été détectés à la jonction entre le bulbe olfactif et le nerf olfactif. Autrement dit, l'infection des neurones sensoriels olfactifs se ferait via le transport axonal et serait à l'origine de la perte totale ou partielle d’odorat (anosmie) chez les patients Covid-19[107]. Il est suggéré que la neuropiline 1 pourrait faciliter le transport axonal du SARS-CoV-2[108] ;
- via un cheval de Troie : par sa capacité à reconnaître les récepteurs DC-SIGN (en) et L-SIGN (en), le SARS-CoV-2 peut infecter ou s’agripper à des globules blancs comme les monocytes, les macrophages ou les cellules dendritiques. Il est très probable que le SARS-CoV-2 les utilise comme un cheval de Troie pour migrer vers d’autres organes en empruntant soit la circulation sanguine, soit le système lymphatique[93] - [92]. Le SARS-CoV-2 peut circuler dans le système lymphatique et l'infecter, même dans des formes modérées de Covid-19[90] ;
- via une virémie : dans les formes les plus sévères du Covid-19, si le SARS-CoV-2 se réplique très activement dans les cellules endothéliales des vaisseaux sanguins (artères, veines), en particulier dans les vaisseaux des poumons et du cœur, il peut se produire une virémie[87]. Autrement dit, le virus circule dans le sang et se répand dans d’autres organes sans utiliser spécifiquement de cheval de Troie.
Incubation et charge virale
La période d’incubation est le délai entre la contamination et l’apparition des premiers symptômes de la maladie. Dans le cas d'une infection au SARS-CoV-2, la période d'incubation varie entre deux et quatorze jours[109]. La période d'incubation médiane est de 5 jours, et 97,5 % de ceux qui développent des symptômes le font en moins de 11,5 jours[110].

Bien que des études aient suggéré que le pic de charge virale dans les voies respiratoires supérieures (nez, pharynx) soit atteint avant l'apparition des premiers symptômes de la maladie, il semble que le pic se produise en fait peu de temps après, voire 3 à 5 jours après. Ce pic est suivi d'une clairance continue du SARS-CoV-2 dans l’organisme. Dans les voies respiratoires supérieures, l'ordre de grandeur du pic viral est similaire entre les individus asymptomatiques et symptomatiques infectés, autour de 70 millions de copies d'ARN/mL par test PCR. Néanmoins, de manière assez similaire à la grippe, la clairance virale est plus rapide chez les individus asymptomatiques que chez ceux qui sont symptomatiques[111]. Plus de la moitié des contaminations serait le fait de porteurs asymptomatiques[112].
Les asymptomatiques peuvent tolérer un certain degré d'infection des voies respiratoires inférieures sans développer de symptômes. Indépendamment de l’âge, environ 40 à 50 % des Covid-19 asymptomatiques pourraient présenter des anomalies radiographiques des poumons, le plus souvent des nodules en verre dépoli[113] - [114]. Le verre dépoli aigu exprime un œdème, une hémorragie ou simplement une inflammation pulmonaire. Généralement il faut moins de 3 mois pour que ces nodules disparaissent[115].
Il est suggéré que le taux de létalité apparente (décès/cas confirmés) du SARS-CoV-2 serait autour de 2 %, tandis que le taux de létalité réelle (décès/cas réels) est autour de 0,8 %[116], ce qui est élevé au regard de la contagiosité du SARS-CoV-2. Les personnes développant des formes sévères sont généralement âgées ou en surpoids, ou présentent des comorbidités (hypertension, diabète, cancers, etc.). En France, en 2020, l'âge médian des décès est de 84 ans[117], et 75 % des patients décédés ont plus de 75 ans[118]. Le taux de mortalité des malades admis en réanimation est compris suivant les hôpitaux entre 10 %[119] et 40 %[120].
Une typologie à affiner pour distinguer les différentes réponses immunitaires face au SARS-CoV-2 est :
- 40 % d’asymptomatiques[121] ;
- 40 % de symptomatiques légers, type grippe ;
- 15 % de formes modérées pouvant conduire à une Covid long[122] ;
- 5 % de formes sévères pouvant nécessiter des soins de réanimation[123].
Covid léger
De manière probablement assez similaire à la grippe, l'ARN viral du SARS-CoV-2 finit par être détecté par des globules blancs « sentinelles » comme les cellules dendritiques ou les macrophages. La détection du virus conduit à la sécrétion d'interférons de type I (IFN), de chimiokines et de cytokines pro-inflammatoires[124], induisant un état antiviral généralisé. Sur le site de l'infection des globules blancs spécifiques à la réponse innée sont recrutés. Les plus importants sont les lymphocytes NK qui pilotent la clairance virale. Les monocytes et les neutrophiles vont aider à éliminer les cellules mortes infectées[124].
Chez les Covid-19 asymptomatiques, ces mécanismes de défense du système immunitaire inné pourraient être suffisants pour venir à bout du SARS-CoV-2. Chez les Covid-19 légèrement symptomatiques, la clairance ultime vient avec l’immunité adaptative où interviennent les lymphocytes T et les lymphocytes B. L'immunité adaptative ou la mémoire immunitaire est activée dès qu'un globule blanc « sentinelle » présente un antigène, c'est-à-dire un fragment du SARS-CoV-2, aux lymphocytes T dans un ganglion lymphatique[125]. Les lymphocytes sont les seuls globules blancs à avoir une mémoire[126]. L’immunité adaptative s’appuie sur :
- l’immunité cellulaire qui implique les lymphocytes T dont les 3 fonctions sont de (1) coordonner la réponse immunitaire avec les lymphocytes T CD4, (2) tuer les cellules infectées avec les lymphocytes T CD8, et (3) réguler l’intensité et la durée de la réponse ;
- l’immunité humorale c’est-à-dire sur les lymphocytes B producteurs d’anticorps.
La très grande majorité de la population mondiale a déjà contracté un rhume à cause d’un coronavirus bénin comme le HCoV-NL63, le HCoV-229E ou encore le HCoV-OC43[127]. À la suite de cette infection, des lymphocytes T à mémoire ont conservé une immunité cellulaire de combat et sont capables de la mobiliser contre le SARS-CoV-2, une fois que l'antigène leur a été présenté. Il s’agit donc d’une immunité cellulaire croisée[128] - [129] - [130]. En revanche il est suggéré qu'il n'existe pas d'immunité croisée pour les lymphocytes B qui produisent des anticorps[131]. Dans le cas d'une infection par le SARS-CoV-2, seuls les anticorps ciblant la protéine S du SARS-CoV-2 se révéleraient neutralisants[105]. Les anti-S commencent à être détectés en sérologie, environ deux semaines après l’infection. Les anticorps contre la protéine S ciblent seulement quelques fragments spécifiques : le RBD représente entre 65 et 77 % des anti-S, et le NTD représente entre 6 et 20 % des anti-S[104].
Les résultats de plusieurs équipes mettent en évidence que tous les patients infectés ne développent pas de réponse anticorps. Le taux d’anticorps était significativement plus élevé chez les patients plus âgés[128]. L’ensemble évoque que l’on peut guérir de l’infection en l’absence d’anticorps et que ceci est particulièrement vrai pour les plus jeunes et les patients asymptomatiques, faisant évoquer l’importance de l’immunité innée et de l’immunité cellulaire dans la clairance virale[128].
Covid sévère
Dans les formes modérées de Covid-19 et particulièrement dans celles sévères, la réponse innée est inefficiente. Elle se caractérise par un taux d’interférons de type I et III relativement bas[132]. Et surtout dans la semaine consécutive à l’infection, il est observé chez ces patients une chute des lymphocytes T, avec une lymphopénie en forme de V. Toutes les situations de stress aigu peuvent se compliquer de lymphopénie via l’augmentation des taux circulants de cortisol par le système endocrinien et spécifiquement par les glandes corticosurrénales. Alors que chez un individu en bonne santé, le taux de cortisol est généralement autour de 375 nmol/L[133], chez les patients Covid-19 en état de stress, ce taux atteint rapidement 620 nmol/L[134] - [135].
La raréfaction des lymphocytes est rapidement compensée par une synthèse massive de neutrophiles (neutrophilie)[136] - [137]. Les neutrophiles qui se substituent aux lymphocytes pour lutter contre le SARS-CoV-2 libèrent dans les tissus infectés des « NEToses », c’est-à-dire des fibres composées d'ADN et de protéines, dont la fonction est de piéger des micro-organismes pathogènes (bactéries ou autres). Un excès de neutrophiles contribue à la thrombogénèse, c'est-à-dire à la formation de caillots sanguins[138].
La réponse immunitaire innée et adaptative des formes sévères de Covid-19 est globalement contre-productive et génère autant de dégâts dans l'organisme que le virus. Du fait de la lymphopénie, les lymphocytes T sont recrutés en nombre réduit. Ces lymphocytes T sur-expriment le récepteur CD69, la protéine Tim-3 (en) et le facteur de transcription aiolos (en), ce qui a pour effet une production massive d’interférons gamma (IFN-γ)[132] - [139] - [140]. En réponse à l’élévation du taux d’IFN-γ, les monocytes et macrophages sécrètent massivement de l’IP-10 (en), une chimiokine appelée également CXCL10. L’élévation du taux de CXCL10 annonce le syndrome de détresse respiratoire aiguë de la Covid-19[141].
La concomitance de l’apparition des anticorps, deux semaines après l’infection, avec l’orage cytokinique, ainsi que la corrélation positive entre le taux d’anticorps et la sévérité de l’infection a fait envisager l’hypothèse que les anticorps puissent être impliqués dans les mécanismes physiopathologiques[128]. Dans une étude auprès de 15 patients Covid-19 brésiliens admis en soins intensifs, la moitié des patients présentaient une infection des globules blancs[97] - [98]. Très peu, sinon aucun des lymphocytes T était infecté. En revanche les lymphocytes B étaient particulièrement ciblés par le SARS-CoV-2[97] - [98].
Peut-être en lien avec des anticorps non neutralisants et facilitants, il est observé une prolifération de monocytes / macrophages qui sécrètent des niveaux élevés de cytokines et chimiokines notamment des IL-6, IL-8 et IL-10[142]. Les formes sévères nécessitant une hospitalisation en unité de soins intensifs se démarquent en particulier par des concentrations plus élevées de IP-10/CXCL10 (en), MCP-1/CCL2 et TNFα[141].
Les taux de cytokines de la Covid-19 sévère ne sont pas spécialement élevés, en comparaison d’un choc septique ou d'une infection respiratoire aiguë sévère classique[143]. Pourtant il se produit bien un orage cytokinique avec la Covid-19, ce qui concrètement se traduit par un processus incontrôlé de mort cellulaire par pyroptose (en), apoptose et nécroptose des tissus infectés[144]. La mort cellulaire est mesurable par le taux sérique de lactate déshydrogénase (LDH). Chez plus de 95 % des patients décédés de la Covid, le taux de LDH est au-dessus du seuil de tolérance qui est fixé à 250 U/L[145]. Et ce processus de destruction cellulaire est induit par le cocktail de cytokines de la Covid-19[144]. Il est suggéré que la production concomitante de TNFα et d’IFNγ induit une production délétère d'oxyde nitrique, à l’origine d'une mort cellulaire incontrôlable et in fine du syndrome de détresse respiratoire aiguë[144]. L’utilisation à forte dose de corticoïdes pour soigner des Covid sévères inhibe les IFNγ[146] et par conséquent pourrait empêcher la production mortelle d'oxyde nitrique.
Mutation et variants
D'abord réputé stable[147], le SARS-CoV-2 s’avère en fait un virus extrêmement instable[148] - [149].
Les mutations de la protéine S sont celles qui ont été le plus médiatisées. La première mutation d'importance est D614G qui a favorisé l'infectivité du SARS-CoV-2[150]. À partir de décembre 2020, le variant anglais se distingue par au moins 17 modifications (mutations ou délétions), toutes protéines virales confondues, un record[151]. La plus connue des mutations est N501Y qui a amélioré la liaison du RBD avec le récepteur ACE2[151]. Le variant anglais multiplie par deux l’infectivité du virus[152]. En parallèle apparaissent les variants sud-africain et brésilien qui ont la particularité de partager avec le variant anglais la mutation N501Y. Mais ces deux variants contiennent surtout des mutations comme E484K qui affaiblissent l’efficacité des anticorps des vaccins de première génération et facilitent les réinfections au SARS-CoV-2[153].
Au total, fin mars 2021, sur les 1 273 codons de la protéine S, il est recensé 28 mutations (2 %) qui se propagent[154] :
- 11 d’entre elles (40 %) concernent le NTD : mutations aux codons 18, 69, 70, 80, 138, 144, 215, 222, 241, 242 et 243 ;
- 6 d’entre elles (20 %) concernent le RBD : mutations aux codons 417, 439, 452, 477, 484 et 501 ;
- 4 concernent les SD1 et SD2 : mutations aux codons 570, 614, 677 et 681.
Le NTD est le fragment le plus instable de la protéine S, autrement dit celui qui mute le plus rapidement. Il est anticipé que la prochaine mutation majeure sur le NTD se produise sur le codon 248. Cette mutation pourrait encore affaiblir davantage les anticorps des vaccins de première génération (AstraZeneca, Pfizer, Moderna…)[155] - [156].
En dehors de la protéine S, fin mars 2021, le SARS-CoV-2 concentre de fortes mutations sur :
- ORF9c : sur les 73 codons de cette protéine, sept (10 %) sont en pleine évolution : 194, 199, 202, 203, 204, 205 et 220[154]. La protéine ORF9c est essentielle au SARS-CoV-2 pour déréguler à son profit les gènes des cellules infectées[73] - [74] - [75] ;
- ORF9b : sur ses 97 codons, quatre (4 %) mutent assez fortement : 10, 16, 32 et 70[154]. Outre son activité anti-interférons, la protéine ORF9b bloque l’apoptose (autodestruction de la cellule)[71] - [72] ;
- ORF8 : sur ses 121 codons, sept (6 %) ont déjà des mutations bien avancées : 27, 52, 68, 73, 84 et 92[154]. La protéine ORF8 permet au SARS-CoV-2 de bloquer la présentation d’un antigène par le CMH-I des cellules infectées, ce qui retarde la réponse immunitaire adaptative[76] - [77] ;
- ORF3b : sur les 156 codons de cette protéine, seulement quatre connaissent une forte évolution : 171, 172, 174 et 223[154]. Mais ORF3b est la protéine du SARS-CoV-2 exerçant la plus forte activité anti-interférons. Et surtout ORF3b est tronquée. L’ORF3b qui la code contient quatre codons-stop qui devraient sauter au fur et à mesure des mutations et permettre progressivement à cette protéine d’exercer une activité anti-interférons de plus en plus forte[51].
Finalement, il existe des milliers de variants du SARS-CoV-2, certains influencent négativement la santé des humains, les diagnostics et les vaccins[157]. Globalement, les variants du SARS-CoV-2 qui s'imposent sont ceux qui sont les plus contagieux ou qui échappent aux vaccins de première génération.
| Virus | Vitesse de mutation (Substitutions/site/an) | |
|---|---|---|
| Mutation la + rapide | Polio[158] | ∼1,0 × 10−2 |
| SARS-CoV-2[148] - [149] | 5,2 à 8,1 × 10−3 | |
| VIH-1[159] | 3 à 8 × 10−3 | |
| Grippe[159] | 2 à 8 × 10−3 | |
| Virus de l'hépatite C[160] | 1,5 × 10−3 | |
| Virus de l'hépatite A[161] | 1,21 à 2 × 10−3 | |
| Ebola[162] | 1,2 × 10−3 | |
| Dengue[159] | 7 à 9 × 10−4 | |
| Rougeole[163] | 6,0 à 6,5 × 10−4 | |
| Virus de la rage[164] | 5 × 10−4 | |
| Virus de l'encéphalite japonaise[165] | 3,5 à 5,3 × 10−4 | |
| HCov-229E[166] | 3 à 8 × 10−4 | |
| Virus de la fièvre jaune[159] | 2 à 5 × 10−4 | |
| Varicelle[167] | 1,82 × 10−5 à 3,8 × 10−6 | |
| Virus de l'hépatite B | ~10−6 | |
| HPV-18[168] | ∼4,5 × 10−7 | |
| Mutation la plus lente | EBV[169] | ~10−9 |
Nom du virus et de la maladie
D'abord dénommé « coronavirus de Wuhan »[170] puis « nouveau coronavirus 2019 » (2019-nCoV), son nom officiel SARS-CoV-2 (pour « severe acute respiratory syndrome coronavirus 2 », en anglais) a été défini le par l'International Committee on Taxonomy of Viruses (ICTV)[1]. La forme longue en français de l’acronyme SARS-CoV-2 est désignée par l’OMS « coronavirus 2 du syndrome respiratoire aigu sévère »[171], tandis que l’Office québécois de la langue française le désigne « coronavirus du syndrome respiratoire aigu sévère 2 » et francise partiellement l'acronyme en SRAS-CoV-2[170] - [Note 1] - [Note 2].
Le groupe d'étude Nidovirales de l'ICTV a proposé le nom SARS-CoV-2 au terme d'une analyse taxonomique poussée[4]. Ce travail montre notamment que le nouveau coronavirus appartient à la même espèce biologique (SARSr-CoV) que SARS-CoV-1 qui avait causé l'épidémie de SRAS en 2003, même si le syndrome observé en 2019 diffère du SRAS à proprement parler.
Simultanément, l'OMS donne à la maladie liée au virus le nom officiel de « maladie à coronavirus 2019 » (COVID-19[171] - [172], de l'anglais coronavirus disease 2019) qui avant était informellement dénommée « pneumonie de Wuhan ». À noter que Covid-19 désigne la maladie et non le coronavirus, d'où des recommandations de l'employer au féminin[173] - [174].
Même si nommer différemment la maladie et l'agent qui la cause est habituel (exemple : le VIH cause le sida), et si la définition des espèces biologiques dépend d'autres éléments que ceux purement conjoncturels (exemple : Botrytis cinerea cause la pourriture grise mais aussi la pourriture noble recherchée pour la vinification des sauternes), l'apparition des deux noms le même jour a d'abord suscité quelques incompréhensions tant dans le public peu averti mais sensibilisé par une situation de crise[175], que dans la communauté scientifique[176] - [177].
Selon l'historien Frédéric Vagneron, « c’est sans doute la première fois dans l’histoire que l’on a détecté un virus avant même de donner un nom à la maladie qu’il provoque »[178].
Ressources scientifiques
L'émergence de SARS-CoV-2, jusque là inconnu de la communauté scientifique, a donné lieu en quelques semaines à une explosion sans précédent de production scientifique, à son partage à l'échelle planétaire et à son application pour la mise au point de tests de dépistage, de vaccins et de traitements, pour le bénéfice public mais soulevant parfois des questions de fiabilité[179] - [180].
Les archives de prépublications, notamment bioRxiv et medRxiv, et divers forums de chercheurs, permettent une diffusion rapide — mais non formellement approuvée par les pairs — de l'information scientifique concernant SARS-CoV-2[181]. BioRxiv a publié le premier preprint sur SARS-CoV-2 (alors nommé 2019-nCoV) le [182] et medRxiv le [183].
Les premières publications scientifiques validées par les pairs datent du [184]. Plusieurs grandes revues et grands éditeurs scientifiques, devant l'urgence de la situation, ont décidé de rendre disponibles hors abonnement certaines publications scientifiques sur le nouveau coronavirus et la pneumonie associée. C'est notamment le cas de Elsevier[185], The Lancet[186], The New England Journal of Medicine[187], Science[188], Springer-Nature[189] ou encore Wiley[190].
Le 2 avril 2020, la Commission européenne a ouvert un portail de données dédié[191], appuyé sur l'infrastructure ELIXIR (en), afin de faciliter la collecte et le partage des données de recherche disponibles[192] : séquences, données d'expression, protéines, structures, littérature, autres. Cette ressource sert au passage de pilote pour la mise en place du dispositif European Open Science Cloud (EOSC).
Une plateforme internationale en open-data, GISAID, est destinée à recueillir les données sur le séquençage du génome du virus, et qui a par ailleurs étendu sa collecte à des données épidémiologiques. Ces données sont exploitées par un projet lui aussi en accès libre, NextStrain[193]. Au , GISAID recense plus de 63 000 séquences du génome. Il semblerait toutefois qu'en raison d'enjeux financiers ou de pouvoir, le partage des données se tarisse après le pic de la pandémie. Selon Mediapart (à la date de juillet 2020), la France n'aurait publié que « 394 génomes dans cette base, soit à peine 1 % du nombre total de génomes partagés au niveau international »[194].
Illustration iconique
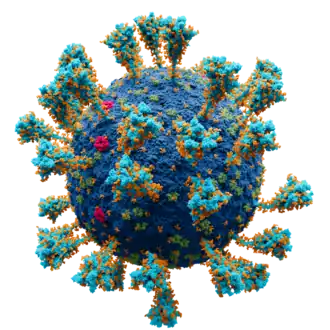
.png.webp)

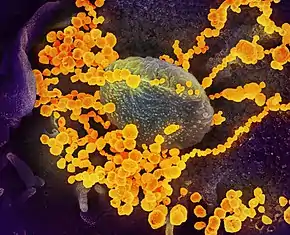
La modélisation de la particule (virion) de SARS-CoV-2 par les illustrateurs médicaux Alissa Eckert et Dan Higgins, des centres pour le contrôle et la prévention des maladies, les principales agences fédérales des États-Unis en matière de protection de la santé publique, s'est imposée aux médias du monde entier comme l'image iconique du virus ; quand bien même des illustrations plus précises sont apparues ultérieurement. En pleine pandémie, elle fait la Une de la presse et des émissions télévisuelles ; les gens se l'approprient à travers des déguisements, des peluches ou des piñatas cathartiques. Elle en devient le symbole[195].
Diffusée sous licence libre au tout début de l'épidémie à partir de , il s'agit de la première modélisation du virus créée afin d'alerter la population américaine.
Basée sur une photographie en noir & blanc issue du Centre chinois pour le contrôle et la prévention des maladies de Pékin, son rendu est colorisé et stylisé, et n'est pas strictement fidèle à la réalité scientifique[195]. En effet, la lumière polarisée, la texture granuleuse, la rondeur parfaite de la cellule ainsi que les couleurs chaudes sont des mises en scène. Ainsi, le rouge des protéines S évoque inconsciemment le danger et le sang ; les grains jaunes des protéines E et orange des protéines M sont gommés pour mettre en valeur les protéines S qui figurent l'identité du virus. Selon l'historienne de l'art Fleur Hopkins-Loféron, cette illustration est l'héritière de l'image du virus du SIDA : une boule rougeoyante inquiétante ornée de piquants agressifs[195].
En 2020, cette modélisation reçoit le prix Beazley décerné par le Design Museum de Londres[195].
Notes et références
Notes
- L'acronyme SRAS-CoV-2 est presque uniquement utilisé au Canada et en Suisse romande.
- L'OMS, en français, recommande l'usage du nom officiel défini par l'ICTV [c'est-à-dire SARS-CoV-2] (voir Appellation de la maladie à coronavirus 2019 (COVID-19) et du virus qui la cause, § Quel est le nom utilisé par l'OMS pour le virus ?).
- Dans tous les cas, le virion est toujours d'une taille largement suffisante pour être assez facilement visible au microscope électronique, y compris sur des coupes minces, sans toutefois qu'on puisse le distinguer par sa forme d'autres coronavirus.
Références
- (en) « Information • News • 2020 », voir § Naming the 2019 Coronavirus, ICTV, (consulté le ).
- (en) Yujia Alina Chan, « The Emergence of the Spike Furin Cleavage Site in SARS-CoV-2 », Molecular Biology and Evolution, (lire en ligne)
- (en) Megan Scudellari, « How the coronavirus infects cells — and why Delta is so dangerous », Nature, vol. 595, no 7869, , p. 640–644 (ISSN 0028-0836 et 1476-4687, DOI 10.1038/d41586-021-02039-y, lire en ligne, consulté le )
- (en) Alexander E. Gorbalenya, Susan C. Baker, Ralph S. Baric, Raoul J. de Groot, Christian Drosten, Anastasia A. Gulyaeva, Bart L. Haagmans, Chris Lauber, Andrey M. Leontovich, Benjamin W. Neuman, Dmitry Penzar, Stanley Perlman, Leo L.M. Poon, Dmitry Samborskiy, Igor A. Sidorov, Isabel Sola et John Ziebuhr, « Severe acute respiratory syndrome-related coronavirus – The species and its viruses, a statement of the Coronavirus Study Group », bioRxiv, (DOI 10.1101/2020.02.07.937862, lire en ligne, consulté le ).
- (en) « Phylogeny of SARS-like betacoronaviruses », nextstrain (consulté le ).
- (en) David S Hui, Esam I Azhar, Tariq A Madani, Francine Ntoumi, Richard Kock, Osman Dar, Giuseppe Ippolito, Timothy D Mchugh, Ziad A Memish, Christian Drosten, Alimuddin Zumla et Eskild Petersen, « The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health - The latest 2019 novel coronavirus outbreak in Wuhan, China », International Journal of Infectious Diseases, vol. 91, , p. 264-266 (PMID 31953166, DOI 10.1016/j.ijid.2020.01.009, lire en ligne, consulté le ).
 .
. - (en) Antonio C. P. Wong, Xin Li, Susanna K. P. Lau et Patrick C. Y. Woo, « Global Epidemiology of Bat Coronaviruses », Viruses, vol. 11(2), no 174, (DOI 10.3390/v11020174, lire en ligne, consulté le ).
- (en) Na Zhu, Dingyu Zhang, Wenling Wang et Xingwang Li, « A Novel Coronavirus from Patients with Pneumonia in China, 2019 », New England Journal of Medicine, , NEJMoa2001017 (ISSN 0028-4793 et 1533-4406, DOI 10.1056/NEJMoa2001017, lire en ligne, consulté le ).
- (en) Na Zhu, Dingyu Zhang, Wenling Wang et Xingwang Li, « A Novel Coronavirus from Patients with Pneumonia in China, 2019 », New England Journal of Medicine, vol. 382, no 8, , p. 727–733 (ISSN 0028-4793 et 1533-4406, PMID 31978945, PMCID PMC7092803, DOI 10.1056/NEJMoa2001017, lire en ligne, consulté le ).
- (en) Cynthia S. Goldsmith et Sara E. Miller, « Modern Uses of Electron Microscopy for Detection of Viruses », Clinical Microbiology Reviews, vol. 22, no 4, , p. 552–563 (ISSN 0893-8512 et 1098-6618, PMID 19822888, PMCID PMC2772359, DOI 10.1128/CMR.00027-09, lire en ligne, consulté le ).
- (en) « New-type coronavirus causes pneumonia in Wuhan: expert », XinhuaNet, (lire en ligne, consulté le ).
- (en) « CoV2020 », platform.gisaid.org (consulté le ).
- (en) SARS-CoV, B. S. (2020). Mutation and Recombination[PDF].
- (en) Elaine Y P Lee, Ming-Yen Ng et Pek-Lan Khong, « COVID-19 pneumonia: what has CT taught us? », The Lancet Infectious Diseases, vol. 20, , p. 384-385 (lire en ligne, consulté le ).
- (en) Roujian Lu, Xiang Zhao, Juan Li, Peihua Niu et Bo Yang, « Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding », The Lancet, vol. 395, no 10224, , p. 565-574 (lire en ligne, consulté le ).
- Félix Roudaut, « Coronavirus : qu’est-ce que la maladie de Kawasaki qui inquiète le monde médical ? », sur rtl.fr, (consulté le ).
- (en) Benvenuto D. et al., « The 2019-new Coronavirus epidemic: evidence for virus evolution », bioRxiv.org, (lire en ligne).
- Hong Zhou, Jingkai Ji, Xing Chen, Yuhai Bi, Juan Li, Qihui Wang, Tao Hu, Hao Song, Runchu Zhao, Yanhua Chen, Mingxue Cui, Yanyan Zhang, Alice C. Hughes, Edward C. Holmes et Weifeng Shi, « Identification of novel bat coronaviruses sheds light on the evolutionary origins of SARS-CoV-2 and related viruses », Cell, , S0092867421007091 (DOI 10.1016/j.cell.2021.06.008)
- (en) S Wacharapluesadee, CW Tan, P Maneeorn, P Duengkae, F Zhu, Y Joyjinda, T Kaewpom, WN Chia, W Ampoot, BL Lim, K Worachotsueptrakun, VC Chen, N Sirichan, C Ruchisrisarod, A Rodpan, K Noradechanon, T Phaichana, N Jantarat, B Thongnumchaima, C Tu, G Crameri, MM Stokes, T Hemachudha et LF Wang, « Evidence for SARS-CoV-2 related coronaviruses circulating in bats and pangolins in Southeast Asia. », Nature Communications, vol. 12, no 1, , p. 972 (PMID 33563978, PMCID 7873279, DOI 10.1038/s41467-021-21240-1).
- (en) Vibol Hul, Deborah Delaune, Erik A. Karlsson, Alexandre Hassanin, Putita Ou Tey, Artem Baidaliuk, Fabiana Gámbaro, Vuong Tan Tu, Lucy Keatts, Jonna Mazet, Christine Johnson, Philippe Buchy, Philippe Dussart, Tracey Goldstein, Etienne Simon-Lorière et Veasna Duong, « A novel SARS-CoV-2 related coronavirus in bats from Cambodia », sur bioRxiv, (DOI 10.1101/2021.01.26.428212), p. 2021.01.26.428212.
- (en) Shin Murakami, Tomoya Kitamura, Jin Suzuki, Ryouta Sato, Toshiki Aoi, Marina Fujii, Hiromichi Matsugo, Haruhiko Kamiki, Hiroho Ishida, Akiko Takenaka-Uema, Masayuki Shimojima et Taisuke Horimoto, « Detection and Characterization of Bat Sarbecovirus Phylogenetically Related to SARS-CoV-2, Japan », Emerging Infectious Diseases, vol. 26, no 12, , p. 3025–3029 (DOI 10.3201/eid2612.203386).
- (en) Hong Zhou, Xing Chen, Tao Hu, Juan Li, Hao Song, Yanran Liu, Peihan Wang, Di Liu, Jing Yang, Edward C. Holmes, Alice C. Hughes, Yuhai Bi et Weifeng Shi, « A Novel Bat Coronavirus Closely Related to SARS-CoV-2 Contains Natural Insertions at the S1/S2 Cleavage Site of the Spike Protein », Current Biology, vol. 30, no 11, , p. 2196–2203.e3 (DOI 10.1016/j.cub.2020.05.023).
- (en) Tommy Tsan-Yuk Lam, Na Jia, Ya-Wei Zhang, Marcus Ho-Hin Shum, Jia-Fu Jiang, Hua-Chen Zhu, Yi-Gang Tong, Yong-Xia Shi, Xue-Bing Ni, Yun-Shi Liao, Wen-Juan Li, Bao-Gui Jiang, Wei Wei, Ting-Ting Yuan, Kui Zheng, Xiao-Ming Cui, Jie Li, Guang-Qian Pei, Xin Qiang, William Yiu-Man Cheung, Lian-Feng Li, Fang-Fang Sun, Si Qin, Ji-Cheng Huang, Gabriel M. Leung, Edward C. Holmes, Yan-Ling Hu, Yi Guan et Wu-Chun Cao, « Identifying SARS-CoV-2-related coronaviruses in Malayan pangolins », Nature, vol. 583, no 7815, , p. 282–285 (DOI 10.1038/s41586-020-2169-0).
- (en) Ping Liu, Jing-Zhe Jiang, Xiu-Feng Wan, Yan Hua, Linmiao Li, Jiabin Zhou, Xiaohu Wang, Fanghui Hou, Jing Chen, Jiejian Zou et Jinping Chen, « Are pangolins the intermediate host of the 2019 novel coronavirus (SARS-CoV-2)? », PLOS Pathogens, vol. 16, no 5, , e1008421 (DOI 10.1371/journal.ppat.1008421).
- (en) H Zhou, X Chen, T Hu, J Li, H Song, Y Liu, P Wang, D Liu, J Yang, EC Holmes, AC Hughes, Y Bi et W Shi, « A Novel Bat Coronavirus Closely Related to SARS-CoV-2 Contains Natural Insertions at the S1/S2 Cleavage Site of the Spike Protein. », Current biology : CB, vol. 30, no 11, , p. 2196-2203.e3 (PMID 32416074, DOI 10.1016/j.cub.2020.05.023).
- (en) Zhou P, Yang XL, Wang XG, Hu B, Zhang L, Zhang W, Si HR, Zhu Y, Li B, Huang CL, Chen HD, Chen J, Luo Y, Guo H, Jiang RD, Liu MQ, Chen Y, Shen XR, Wang X, Zheng XS, Zhao K, Chen QJ, Deng F, Liu LL, Yan B, Zhan FX, Wang YY, Xiao GF, Shi ZL, « Addendum: A pneumonia outbreak associated with a new coronavirus of probable bat origin », Nature, vol. 588, no 7836, , E6 (PMID 33199918, DOI 10.1038/s41586-020-2951-z, lire en ligne).
- (en) Peng Zhou, Xing-Lou Yang, Xian-Guang Wang et Ben Hu, « A pneumonia outbreak associated with a new coronavirus of probable bat origin », Nature, (ISSN 0028-0836 et 1476-4687, DOI 10.1038/s41586-020-2012-7, lire en ligne, consulté le ).
- (en) Fan Wu, Su Zhao, Bin Yu et Yan-Mei Chen, « A new coronavirus associated with human respiratory disease in China », Nature, vol. 579, no 7798, , p. 265–269 (ISSN 0028-0836 et 1476-4687, PMID 32015508, PMCID PMC7094943, DOI 10.1038/s41586-020-2008-3, lire en ligne, consulté le ).
- Marc Gozlan, « Des coronavirus de chauves-souris très proches du SARS-CoV-2 identifiés au Laos », sur Le Monde, (consulté le ).
- DOI 10.21203/rs.3.rs-871965/v1
- Hervé Morin, « Coronavirus : le pangolin a-t-il pu servir d’hôte intermédiaire ? », Le Monde, (lire en ligne). L’université d’agriculture de Chine du Sud a annoncé qu'un séquençage du virus trouvés chez des pangolins montrait qu'il était homologue à 99 % à celui du SARS-CoV-2. Le pangolin aurait pu servir d'espèce réservoir ou hôte entre la chauve-souris et l'humain, une hypothèse discutée par plusieurs spécialistes.
- Makarenkov V. et al. Horizontal gene transfer and recombination analysis of SARS-CoV-2 genes helps discover its close relatives and shed light on its origin, BMC Ecology and Evolution 21, 2021
- Expert reaction to statement from South China Agricultural University that research has identified the pangolin as a possible coronavirus host, Science Media Center, 7/02/2020.
- (en) Tommy Tsan-Yuk Lam, Marcus Ho-Hin Shum, Hua-Chen Zhu et Yi-Gang Tong, « Identifying SARS-CoV-2 related coronaviruses in Malayan pangolins », Nature, (ISSN 0028-0836 et 1476-4687, DOI 10.1038/s41586-020-2169-0, lire en ligne, consulté le ).
- (en) Lamia Wahba, Nimit Jain, Andrew Z. Fire et Massa J. Shoura, « An Extensive Meta-Metagenomic Search Identifies SARS-CoV-2-Homologous Sequences in Pangolin Lung Viromes », mSphere, vol. 5, no 3, , e00160–20, /msphere/5/3/mSphere160–20.atom (ISSN 2379-5042, DOI 10.1128/mSphere.00160-20, lire en ligne, consulté le ).
- (en) Tao Zhang, Qunfu Wu et Zhigang Zhang, « Probable Pangolin Origin of SARS-CoV-2 Associated with the COVID-19 Outbreak », Current Biology, vol. 30, no 7, , p. 1346–1351.e2 (PMID 32197085, PMCID PMC7156161, DOI 10.1016/j.cub.2020.03.022, lire en ligne, consulté le ).
- (en) Rossana Segreto, « The genetic structure of SARS‐CoV‐2 does not rule out a laboratory origin », BioEssays, (lire en ligne, consulté le ).
- (en) Huihui Mou, « Mutations from bat ACE2 orthologs markedly enhance ACE2-Fc neutralization of SARS-CoV-2 », bioRxiv, (lire en ligne, consulté le ).
- DOI 10.1016/j.cell.2021.07.007
- (en) Yun Chen, Yao Guo, Yihang Pan et Zhizhuang Joe Zhao, « Structure analysis of the receptor binding of 2019-nCoV », Biochemical and Biophysical Research Communications, vol. 525, no 1, , p. 135–140 (PMID 32081428, PMCID PMC7092824, DOI 10.1016/j.bbrc.2020.02.071, lire en ligne, consulté le )
- (en) Daniel Wrapp, Nianshuang Wang, Kizzmekia S. Corbett et Jory A. Goldsmith, « Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation », Science, vol. 367, no 6483, , p. 1260–1263 (ISSN 0036-8075 et 1095-9203, DOI 10.1126/science.abb2507, lire en ligne, consulté le )
- Coutard B, Valle C, de Lamballerie X, Canard B, Seidah NG, Decroly E, « The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade », Antiviral Research, vol. 176, , p. 104742 (PMID 32057769, PMCID 7114094, DOI 10.1016/j.cub.2020.03.022).
- (en) Canrong Wu, Yang Liu, Yueying Yang et Peng Zhang, « Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods », Acta Pharmaceutica Sinica B, , S2211383520302999 (DOI 10.1016/j.apsb.2020.02.008, lire en ligne, consulté le )
- Astrid Vabret et Fabien Miszczak, « Changement de tropisme des coronavirus », Revue Francophone des Laboratoires, tropisme des virus : de l'animal à l'homme ?, vol. 2010, no 423, , p. 63–68 (ISSN 1773-035X, DOI 10.1016/S1773-035X(10)70561-X, lire en ligne, consulté le )
- Mandelbaum RF (19 février 2020). "[Scientists Create Atomic-Level Image of the New Coronavirus's Potential Achilles Heel]". Gizmodo. consulté le 13 mars 2020
- (en) Francis K. Yoshimoto, « The Proteins of Severe Acute Respiratory Syndrome Coronavirus-2 (SARS CoV-2 or n-COV19), the Cause of COVID-19 », The Protein Journal, vol. 39, no 3, , p. 198–216 (ISSN 1875-8355, PMID 32447571, PMCID 7245191, DOI 10.1007/s10930-020-09901-4, lire en ligne, consulté le ).
- (en) Chittaranjan Baruah, « Sequence Analysis and Structure Prediction of SARS-CoV-2 Accessory Proteins 9b and ORF14: Evolutionary Analysis Indicates Close Relatedness to Bat Coronavirus », BioMed Research International, (lire en ligne, consulté le ).
- (en) Irwin Jungreis, « Conflicting and ambiguous names of overlapping ORFs in the SARS-CoV-2 genome: A homology-based resolution », Virology, (lire en ligne, consulté le ).
- (en) Philip V’kovski, « Coronavirus biology and replication: implications for SARS-CoV-2 », Nature, (lire en ligne, consulté le ).
- (en) Peter Schmidtke, « SARS-CoV-2 - part 2 - From the viral genome to protein structures », discngine, (lire en ligne, consulté le ).
- (en) Yoriyuki Konno, « SARS-CoV-2 ORF3b Is a Potent Interferon Antagonist Whose Activity Is Increased by a Naturally Occurring Elongation Variant », Cell Rep, (lire en ligne, consulté le ).
- Jean-Claude Hervé et Naoum Salamé, « Réplication et expression de l’information génétique au cours du cycle infectieux du coronavirus », Institut français de l'éducation, (lire en ligne, consulté le ).
- (en) Asmaa Hachim, « ORF8 and ORF3b antibodies are accurate serological markers of early and late SARS-CoV-2 infection », Nature Immunology, (lire en ligne, consulté le ).
- Imane Jamai Amir, « Covid-19 : virologie, épidémiologie et diagnostic biologique », Option/Bio., (lire en ligne, consulté le ).
- (en) Tomer Meirson, « Structural basis of SARS-CoV-2 spike protein induced by ACE2 », Bioinformatics., (lire en ligne, consulté le ).
- (en) Fatemeh Pourrajab, « Molecular Basis for Pathogenicity of Human Coronaviruses », Infect Drug Resist., (lire en ligne, consulté le ).
- (en) Wenfei Song, « Cryo-EM structure of the SARS coronavirus spike glycoprotein in complex with its host cell receptor ACE2 », PLoS Pathog., (lire en ligne, consulté le ).
- (en) Nader Rahimi, « C-type Lectin CD209L/L-SIGN and CD209/DC-SIGN: Cell Adhesion Molecules Turned to Pathogen Recognition Receptors », Biology, (lire en ligne, consulté le ).
- (en) Wai Tuck Soh, « The N-terminal domain of spike glycoprotein mediates SARS-CoV-2 infection by associating with L-SIGN and DC-SIGN », Biorxiv, (lire en ligne, consulté le ).
- « Covid : les mutations qui rendent le variant delta du virus plus contagieux et plus inquiétant », BBC News Brasil, (consulté le )
- Erwan Sallard, « Retrouver les origines du SARS-CoV-2 dans les phylogénies de coronavirus », Med Sci (Paris), (lire en ligne, consulté le ).
- (en) Yong Hu, « The Severe Acute Respiratory Syndrome Coronavirus Nucleocapsid Inhibits Type I Interferon Production by Interfering with TRIM25-Mediated RIG-I Ubiquitination », J Virol., (lire en ligne, consulté le ).
- V. Bonny, « COVID-19 : physiopathologie d’une maladie à plusieurs visages », Rev Med Interne., (lire en ligne, consulté le ).
- Ghizlane Maarifi, « La réponse interféron : Un grand pouvoir implique de grandes responsabilités », EDP Sciences, (lire en ligne, consulté le ).
- (en) « An Overview of Their Replication and Pathogenesis; Section 2 Genomic Organization », Methods in Molecular Biology, Springer, vol. 1282, , p. 1–23 (ISBN 978-1-4939-2438-7, PMID 25720466, PMCID 4369385, DOI 10.1007/978-1-4939-2438-7_1).
- Franck Martin, « Une structure en "épingle à cheveux" contrôle la traduction du génome du virus SARS-CoV-2 », CNRS, (lire en ligne, consulté le ).
- (en) Donghyuk Shin, « Papain-like protease regulates SARS-CoV-2 viral spread and innate immunity », Nature, (lire en ligne, consulté le ).
- (en) Lisa Miorin, « SARS-CoV-2 Orf6 hijacks Nup98 to block STAT nuclear import and antagonize interferon signaling », PNAS, (lire en ligne, consulté le ).
- (en) Amin Addetia, « SARS-CoV-2 ORF6 disrupts nucleocytoplasmic transport through interactions with Rae1 and Nup98 », bioRxiv, (lire en ligne, consulté le ).
- (en) Yoichi Miyamoto, « SARS-CoV-2 ORF6 disturbs nucleocytoplasmic trafficking to advance the viral replication », bioRxiv, (lire en ligne, consulté le ).
- « Une équipe internationale de chercheurs a identifié des vulnérabilités communes aux coronavirus SARS-CoV-2, SARS-CoV-1 et MERS-CoV », CNRS, (lire en ligne, consulté le ).
- (en) He-wei Jiang, « SARS-CoV-2 Orf9b suppresses type I interferon responses by targeting TOM70 », Nature, (lire en ligne, consulté le ).
- (en) Fangfang Lu, « SARS-CoV-2 ORF9c: a mysterious membrane-anchored protein that regulates immune evasion? », Nature Reviews Immunology, (lire en ligne, consulté le ).
- (en) Ana Dominguez Andres, « SARS-CoV-2 ORF9c Is a Membrane-Associated Protein that Suppresses Antiviral Responses in Cells », University of Oxford - Immunology Network, (lire en ligne, consulté le ).
- (en) Nima Hemmat, « The roles of signaling pathways in SARS-CoV-2 infection; lessons learned from SARS-CoV and MERS-CoV », Archives of Virology, (lire en ligne, consulté le ).
- (en) Matthew D. Park, « Immune evasion via SARS-CoV-2 ORF8 protein? », Nature Reviews Immunology, (lire en ligne, consulté le ).
- (en) Y. Zhang, « The ORF8 Protein of SARS-CoV-2 Mediates Immune Evasion through Potently Downregulating MHC-I », University of Oxford - Immunology Network, (lire en ligne, consulté le ).
- (en) Guangyan Miao, « ORF3a of the COVID-19 virus SARS-CoV-2 blocks HOPS complex-mediated assembly of the SNARE complex required for autolysosome formation », Developmental Cell, (lire en ligne, consulté le ).
- (en) Daria Bortolotti, « SARS-CoV-2 Spike 1 Protein Controls Natural Killer Cell Activation via the HLA-E/NKG2A Pathway », Cells., (lire en ligne, consulté le ).
- Mathilde Allard, « Fonctions immunologiques de la molécule HLA-E :Implication en Cancérologie et en Transplantation », École Doctorale « Biologie Santé » Nantes – Angers, (lire en ligne, consulté le ).
- Matthew Borok, « Le cycle viral de SARS-CoV-2 », L'Arbre des connaissances, (lire en ligne, consulté le ).
- (en) Coronaviruses, vol. 1282, Springer, coll. « Methods in Molecular Biology », , 1–23 p. (ISBN 978-1-4939-2438-7, PMID 25720466, DOI 10.1007/978-1-4939-2438-7_1), « Coronaviruses: an overview of their replication and pathogenesis ».
- (en) Julian Buchrieser, « Syncytia formation by SARS‐CoV‐2‐infected cells », EMBO J, (lire en ligne, consulté le ).
- (en) Samuel A. Theuerkauf, « Quantitative assays reveal cell fusion at minimal levels of SARS-CoV-2 spike protein and fusion from without », iScience, (lire en ligne, consulté le ).
- (en) David L. Jones, « Shedding of SARS-CoV-2 in feces and urine and its potential role in person-to-person transmission and the environment-based spread of COVID-19 », Science of The Total Environment, (lire en ligne, consulté le ).
- (en) Smruthi Karthikeyan, Joshua I. Levy, Peter De Hoff et Greg Humphrey, « Wastewater sequencing reveals early cryptic SARS-CoV-2 variant transmission », Nature, (ISSN 0028-0836 et 1476-4687, DOI 10.1038/s41586-022-05049-6, lire en ligne, consulté le )
- (en) Stefanie Deinhardt-Emmer, « Early postmortem mapping of SARS-CoV-2 RNA in patients with COVID-19 and correlation to tissue damage », biorxiv, (lire en ligne [PDF], consulté le ).
- (en) L. He, « Expression of elevated levels of pro-inflammatory cytokines in SARS-CoV-infected ACE2+ cells in SARS patients: relation to the acute lung injury and pathogenesis of SARS », Journal of Pathology, (lire en ligne, consulté le ).
- (en) Yanqing Ding, « Organ distribution of severe acute respiratory syndrome (SARS) associated coronavirus (SARS-CoV) in SARS patients: implications for pathogenesis and virus transmission pathways », Journal of Pathology, (lire en ligne, consulté le ).
- (en) Ernesto Roldán-Santiago, « Severe Acute Respiratory Syndrome Coronavirus 2 Spreads to Lymph Nodes and Strongly Expands CD4+ Effector Memory RA Cells in a Patient With Mild Coronavirus Disease 2019 », Clinical Infectious Diseases, (lire en ligne, consulté le ).
- (en) Xiu-Wu Bian, « Autopsy of COVID-19 patients in China », National Science Review, (lire en ligne).
- (en) Y. Chen, « The Novel Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2) Directly Decimates Human Spleens and Lymph Nodes », University of Oxford - Immunology Network, (lire en ligne, consulté le ).
- (en) Matthew D. Park, « Macrophages: a Trojan horse in COVID-19? », Nature Reviews Immunology, (lire en ligne, consulté le ).
- (en) Jessica S. Meyer, « Acute appendicitis in four children with SARS-CoV-2 infection », Journal of Pediatric Surgery Case Reports, (lire en ligne, consulté le ).
- (en) Huiwen Zheng, « Virulence and pathogenesis of SARS-CoV-2 infection in rhesus macaques: A nonhuman primate model of COVID-19 progression », PLoS Pathog, (lire en ligne, consulté le ).
- Marc Desforges, « Coronavirus humains respiratoires neuro-invasifs et neurotropes : agents neurovirulents potentiels », Virologie, (lire en ligne, consulté le ).
- (en) Mariana Borsa, « Attacking the defence: SARS-CoV-2 can infect immune cells », Nature Reviews Immunology, (lire en ligne, consulté le ).
- (en) Marjorie C. Pontelli, « Infection of human lymphomononuclear cells by SARS-CoV-2 », University of Oxford - Immunology Network, (lire en ligne, consulté le ).
- (en) Jiong Wang, « The potential for antibody-dependent enhancement of SARS-CoV-2 infection: Translational implications for vaccine development », J Clin Transl Sci., (lire en ligne, consulté le ).
- « Vaccins contre la COVID19 : doit-on s'inquiéter du risque de maladie aggravée chez les personnes vaccinées ? », sur VIDAL (consulté le )
- (en) Reviany V. Nidom, « Investigation of the D614G mutation and antibody-dependent enhancement sequences in indonesian SARS-CoV-2 isolates and comparison to southeast Asian isolates », Systematic Reviews in Pharmacy, (lire en ligne, consulté le )
- (en) Dapeng Li, « The functions of SARS-CoV-2 neutralizing and infection-enhancing antibodies in vitro and in mice and nonhuman primates », bioRxiv, (lire en ligne, consulté le ).
- Etienne Decroly, « Rôle du site de clivage par la furine et du domaine de reconnaissance du récepteur ACE2 dans l'émergence du SARS-CoV-2 », IHU Méditerranée-Infection, (lire en ligne, consulté le ).
- (en) Matthew McCallum, « N-terminal domain antigenic mapping reveals a site of vulnerability for SARS-CoV-2 », Cell, (lire en ligne, consulté le ).
- (en) Luca Piccoli, « Mapping Neutralizing and Immunodominant Sites on the SARS-CoV-2 Spike Receptor-Binding Domain by Structure-Guided High-Resolution Serology », Cell, (lire en ligne, consulté le ).
- Stéphane Korsia-Meffre, « Remdésivir : une molécule testée pour une maladie du chat, la fièvre Ebola, et maintenant l’infection à SARS-CoV-2 », Vidal, (lire en ligne, consulté le ).
- Marc Gozlan, « Covid-19 et perte d’odorat : du nouveau sur la persistance du SARS-CoV-2 et son potentiel neuroinvasif », Réalités biomédicales - Le Monde, (lire en ligne, consulté le ).
- (en) Bindu S. Mayi, « The role of Neuropilin-1 in COVID-19 », PLoS Pathog., (lire en ligne, consulté le )
- (en) Knvul Sheik et Roni Caryn Rabin, « The Coronavirus: What Scientists Have Learned So Far », The New York Times, (lire en ligne, consulté le ).
- (en) Stephen A. Lauer, Kyra H. Grantz, Qifang Bi et Forrest K. Jones, « The Incubation Period of Coronavirus Disease 2019 (COVID-19) From Publicly Reported Confirmed Cases: Estimation and Application », Annals of Internal Medicine, (ISSN 0003-4819, DOI 10.7326/M20-0504, lire en ligne, consulté le ).
- (en) Muge Cevik, « SARS-CoV-2, SARS-CoV, and MERS-CoV viral load dynamics, duration of viral shedding, and infectiousness: a systematic review and meta-analysis », Lancet Microbe, (lire en ligne, consulté le ).
- (en) Tapiwa Ganyani, Cecile Kremer, Dongxuan Chen et Andrea Torneri, « Estimating the generation interval for COVID-19 based on symptom onset data », bioRxiv, Infectious Diseases (except HIV/AIDS), (DOI 10.1101/2020.03.05.20031815, lire en ligne, consulté le ).
- (en) Eng Eong Ooi, « Asymptomatic SARS-CoV-2 infection », Lancet Infectious Diseases, (lire en ligne, consulté le ).
- (en) Rasha Mostafa Mohamed Ali, « Radiological findings spectrum of asymptomatic coronavirus (COVID-19) patients », Egypt J Radiol Nucl Med., (lire en ligne, consulté le ).
- Gilbert Ferretti, « Nodule en verre dépoli : quelle prise en charge en 2014 ? », CHU Grenoble, (lire en ligne, consulté le ).
- Bruno Laporte, « Covid-19 : Mortalité, létalité, morbidité », Twitter, (lire en ligne, consulté le ).
- Anaïs Thiébaux, « Victimes du coronavirus : qui en meurt, à quel âge, avec quelle maladie ? », Le Journal des femmes, (lire en ligne, consulté le ).
- (en) « Coronavirus statistics: Latest numbers on COVID-19 cases and deaths », Euronews, (lire en ligne).
- Luc Peillon, « Covid-19 : Quel est le taux de mortalité des malades du Covid-19 en réanimation? », Libération, (lire en ligne, consulté le ).
- « Covid-19 : la mortalité en réanimation beaucoup plus forte qu'annoncée, selon une étude », sur L'express, .
- (en) Daniel P. Oran, « Prevalence of Asymptomatic SARS-CoV-2 Infection », Annals of Internal Medicine, (lire en ligne, consulté le ).
- Ferney, « Les mystères du Covid long », lacroix.fr, (consulté le ).
- (en) Aaron J. Wilk, « A single-cell atlas of the peripheral immune response in patients with severe COVID-19 », Nat Med., (lire en ligne, consulté le ).
- (en) Akiko Iwasaki, « Innate immunity to influenza virus infection », Nature Reviews Immunology, (lire en ligne, consulté le ).
- « INFOGRAPHIE. Comment le vaccin protège l'organisme », Sciences et Avenir, (lire en ligne, consulté le ).
- « De mémoire de lymphocyte… C’est quoi être immunisé ? », INSERM, (lire en ligne, consulté le ).
- A. Vabret, « Coronavirus humains (HCoV) », Pathol Biol (Paris), (DOI 10.1016/j.patbio.2008.02.018, lire en ligne, consulté le ).
- Jean-Daniel Lelièvre, « Aspects immunologiques et virologiques de l’infection par le SARS-CoV-2 », Haute Autorité de Santé (HAS) de la République française, (lire en ligne, consulté le ).
- Pascal Meylan, « Immunité cellulaire contre SARS-CoV-2 : substrat de l’immunité croisée entre coronavirus saisonniers et SARS-CoV-2 ? », Rev Med Suisse, (lire en ligne, consulté le ).
- Diane Cacciarella, « Covid-19 : une immunité croisée avec le rhume ? », Pourquoi Docteur ?, (lire en ligne, consulté le ).
- (en) Isabelle Sermet-Gaudelus, « Prior infection by seasonal coronaviruses does not prevent SARS-CoV-2 infection and associated Multisystem Inflammatory Syndrome in children », medRxiv, (lire en ligne, consulté le ).
- (en) Stefania Varchetta, « Unique immunological profile in patients with COVID-19 », Nature - Cellular & Molecular Immunology, (lire en ligne, consulté le ).
- (en) Suzanne Brown, « A time‐adjusted cortisol cut‐off can reduce referral rate for Synacthen stimulation test whilst maintaining diagnostic performance », Clin Endocrinol (Oxf), (lire en ligne, consulté le ).
- (en) Tricia Tan, « Association between high serum total cortisol concentrations and mortality from COVID-19 », Lancet Diabetes Endocrinol., (lire en ligne, consulté le ).
- (en) N.S. Panesar, « What caused lymphopenia in SARS and how reliable is the lymphokine status in glucocorticoid-treated patients? », Med Hypotheses., (lire en ligne, consulté le ).
- (en) Didier Payen, « A Longitudinal Study of Immune Cells in Severe COVID-19 Patients », Front Immunol., (lire en ligne, consulté le ).
- (en) Prisca Mutinelli-Szymanski, « Neutrophil:lymphocyte ratio predicts short-term outcome of COVID-19 in haemodialysis patients », Clinical Kidney Journal, (lire en ligne, consulté le ).
- (en) A. Arcanjo, « The emerging role of neutrophil extracellular traps in severe acute respiratory syndrome coronavirus 2 (COVID-19) », Nature - Sci Rep, (lire en ligne, consulté le ).
- Marc Gozlan, « Pneumonie Covid-19 : une inflammation qui tourne en boucle », Réalités biomédicales - Le Monde, (lire en ligne).
- (en) Katarina Radulovic, « CD69 regulates type I IFN-induced tolerogenic signals to mucosal CD4 T cells that attenuate their colitogenic potential », J Immunol., (lire en ligne).
- (en) Francesca Coperchini, « The cytokine storm in COVID-19: An overview of the involvement of the chemokine/chemokine-receptor system », Cytokine Growth Factor Rev., (lire en ligne, consulté le ).
- (en) Willianne Hoepel, « High titers and low fucosylation of early human anti-SARS-CoV-2 IgG promote inflammation by alveolar macrophages », Science Translational Medicine, (lire en ligne, consulté le ).
- Céline Deluzarche, « Coronavirus : la fausse piste de "l'orage cytokinique" », sur Futura-sciences.com (consulté le ).
- (en) Rajendra Karki, « Synergism of TNF-α and IFN-γ Triggers Inflammatory Cell Death, Tissue Damage, and Mortality in SARS-CoV-2 Infection and Cytokine Shock Syndromes », Cell, (lire en ligne, consulté le ).
- (en) Brandon Michael Henry, « Lactate dehydrogenase levels predict coronavirus disease 2019 (COVID-19) severity and mortality: A pooled analysis », Am J Emerg Med., (lire en ligne, consulté le ).
- (en) Xiaoyu Hu, « Inhibition of IFN-γ Signaling by Glucocorticoids », J Immunol, (lire en ligne, consulté le ).
- « Coronavirus chinois : les raisons pour lesquelles il ne faut pas paniquer », sur LExpress.fr, (consulté le ).
- (en) Marta Giovanetti, « The first two cases of 2019‐nCoV in Italy: Where they come from? », J Med Virol., (lire en ligne, consulté le ).
- (en) Malik Sallam, « Temporal increase in D614G mutation of SARS-CoV-2 in the Middle East and North Africa », Heliyon, (lire en ligne, consulté le ).
- (en) Ewen Callaway, « The coronavirus is mutating — does it matter? », Nature, vol. 585, , p. 174-177 (DOI 10.1038/d41586-020-02544-6, lire en ligne [PDF]).
- Loïc Mangin, « Quelles mutations du SARS-CoV-2 doit-on craindre ? », Pour la Science, (lire en ligne, consulté le ).
- Richard Gray, « Comment les mutations du Covid-19 changent la donne de la pandémie », BBC Future, (lire en ligne, consulté le ).
- Marc Gozlan, « Covid-19 : le défi des nouveaux variants », Réalités biomédicales - Le Monde, (lire en ligne, consulté le ).
- (en) « Genomic epidemiology of novel coronavirus - Global subsampling », Nextstrain, (lire en ligne, consulté le ).
- Céline Deluzarche, « Coronavirus : l'efficacité des anticorps est réduite à zéro en moins de 80 jours », Futura Santé, (lire en ligne, consulté le ).
- (en) Emanuele Andreano, « SARS-CoV-2 escape in vitro from a highly neutralizing COVID-19 convalescent plasma », biorxiv, (lire en ligne, consulté le ).
- « Contagiosité, circulation, vaccin... Que sait-on de la nouvelle variante du virus ? », lexpress.fr, (lire en ligne).
- (en) Gene V. Gavrilin, « Evolution of Circulating Wild Poliovirus and of Vaccine-Derived Poliovirus in an Immunodeficient Patient: a Unifying Model », J Virol., (lire en ligne, consulté le ).
- (en) Raphaëlle Klitting, « What Does the Future Hold for Yellow Fever Virus? (II) », Genes, (lire en ligne, consulté le ).
- (en) Andres Moreira-Soto, « Cross-order host switches of hepatitis C-related viruses illustrated by a novel hepacivirus from sloths », Virus Evolution, (lire en ligne, consulté le ).
- (en) Eleonora Cella, « Genotype I hepatitis A virus introduction in Italy: Bayesian phylogenetic analysis to date different epidemics », J Med Virol, (lire en ligne, consulté le ).
- (en) Denis Malvy, « Ebola virus disease », The Lancet, (lire en ligne, consulté le ).
- (en) Yuki Furuse, « Origin of measles virus: divergence from rinderpest virus between the 11th and 12th centuries », Virology Journal, (lire en ligne, consulté le ).
- (en) Kentaro Tohma, « Phylogeographic analysis of rabies viruses in the Philippines », Infection, Genetics and Evolution, (lire en ligne, consulté le ).
- (en) Amy J. Schuh, « Phylogeography of Japanese Encephalitis Virus: Genotype Is Associated with Climate », PLoS Negl Trop Dis., (lire en ligne, consulté le ).
- (en) Krzysztof Pyrc, « Mosaic Structure of Human Coronavirus NL63, One Thousand Years of Evolution », J Mol Biol., (lire en ligne, consulté le ).
- (en) Daniel P. Depledge, « Evolution of Cocirculating Varicella-Zoster Virus Genotypes during a Chickenpox Outbreak in Guinea-Bissau », J Virol., (lire en ligne, consulté le ).
- (en) Cadhla Firth, « Using Time-Structured Data to Estimate Evolutionary Rates of Double-Stranded DNA Viruses », Mol Biol Evol., (lire en ligne, consulté le ).
- (en) Eric R. Weiss, « Early Epstein-Barr Virus Genomic Diversity and Convergence toward the B95.8 Genome in Primary Infection », J Virol., (lire en ligne, consulté le ).
- « SRAS-CoV-2 », Grand Dictionnaire terminologique, Office québécois de la langue française (consulté le ).
- « Appellation de la maladie à coronavirus 2019 (COVID-19) et du virus qui la cause », sur who.int, Organisation mondiale de la santé, (consulté le ).
- (en) « Novel Coronavirus (2019-nCoV) Situation Report – 22 » [PDF], sur who.int, Organisation mondiale de la santé, (consulté le ) : « Following WHO best practices for naming of new human infectious diseases, which were developed in consultation and collaboration with the World Organisation for Animal Health (OIE) and the Food and Agriculture Organization of the United Nations (FAO), WHO has named the disease COVID-19, short for “coronavirus disease 2019.” », p. 1.
- « COVID-19 », Grand Dictionnaire terminologique, Office québécois de la langue française (consulté le ).
- « Le covid 19 ou La covid 19 | Académie française », sur academie-francaise.fr (consulté le ).
- « Épidémie de Covid-19 : la difficulté de bien nommer le virus et la maladie », Le Monde.fr, (lire en ligne, consulté le ).
- (en) Shibo Jiang, Zhengli Shi, Yuelong Shu et Jingdong Song, « A distinct name is needed for the new coronavirus », The Lancet, , S0140673620304190 (DOI 10.1016/S0140-6736(20)30419-0, lire en ligne, consulté le ).
- (en) Yuntao Wu, Wenzhe Ho, Yaowei Huang et Dong-Yan Jin, « SARS-CoV-2 is an appropriate name for the new coronavirus », The Lancet, , S0140673620305572 (DOI 10.1016/S0140-6736(20)30557-2, lire en ligne, consulté le ).
- Sébastien Rouet, « Covid-19, Ebola, peste noire… nos sociétés face aux maladies, avec l’historien Frédéric Vagneron », sur geo.fr, (consulté le ).
- Hervé Morin, Pascale Santi, David Larousserie, Sandrine Cabut et Nathaniel Herzberg, « Comment le Covid-19 chamboule la recherche scientifique », Le Monde.fr, (lire en ligne
 , consulté le ).
, consulté le ). - (en-GB + fr) Conseil français de l'Intégrité Scientifique, « L’intégrité scientifique, la science ouverte et la crise sanitaire », sur ENRIO, (consulté le )
- (en) « COVID-19 SARS-CoV-2 preprints from medRxiv and bioRxiv », sur bioRxiv (consulté le ).
- (en) Tianmu Chen, Jia Rui, Qiupeng Wang et Zeyu Zhao, « A mathematical model for simulating the transmission of Wuhan novel Coronavirus », bioRxiv, Systems Biology, (DOI 10.1101/2020.01.19.911669, lire en ligne, consulté le ).
- (en) Jonathan M Read, Jessica RE Bridgen, Derek AT Cummings et Antonia Ho, « Novel coronavirus 2019-nCoV: early estimation of epidemiological parameters and epidemic predictions », medRxiv, Infectious Diseases (except HIV/AIDS), (DOI 10.1101/2020.01.23.20018549, lire en ligne, consulté le ).
- (en) Chaolin Huang, Yeming Wang, Xingwang Li et Lili Ren, « Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China », The Lancet, , S0140673620301835 (DOI 10.1016/S0140-6736(20)30183-5, lire en ligne, consulté le ).
- (en) « Novel Coronavirus Information Center », sur Elsevier Connect (consulté le ).
- (en) « COVID-19 Resource Centre », sur thelancet.com (consulté le ).
- (en) « Coronavirus (Covid-19) — NEJM », sur New England Journal of Medicine (consulté le ).
- (en) « Coronavirus », sur Science | AAAS (consulté le ).
- (en) « Coronavirus », sur Springer Nature (consulté le ).
- (en) « Novel Coronavirus - Wiley Online Library », sur Novel Coronavirus (consulté le ).
- Le « COVID-19 Data Portal »
- (en) Georges Lobo, « European COVID-19 Data Platform », sur joinup.ec.europa.eu, (consulté le ).
- (en) Claire Jarvis, « How genomic epidemiology is tracking the spread of COVID-19 locally and globally », sur acs.org, .
- Lise Barnéoud, « Données épidémiologiques: la pénurie cachée », sur mediapart.fr, .
- (fr) « Portrait de l'ennemi public n°1 », émission Le dessous des images du 28 novembre 2022, présentée par Sonia Devillers, Arte, 12 minutes (visionner en ligne)
Voir aussi
Bibliographie
- (en) Yixuan Wang, Yuyi Wang, Yan Chen et Qingsong Qin, « Unique epidemiological and clinical features of the emerging 2019 novel coronavirus pneumonia (COVID-19) implicate special control measures », Journal of Medical Virology, (lire en ligne).
- (en) Parham Habibzadeh et Emily K. Stoneman, « The Novel Coronavirus: A Bird's Eye View », The International Journal of Occupational and Environmental Medicine, vol. 11, no 2, , p. 65–71 (ISSN 2008-6520 et 2008-6814, PMID 32020915, PMCID PMC7205509, DOI 10.15171/ijoem.2020.1921, lire en ligne, consulté le )
Articles connexes
- Maladie à coronavirus 2019 - Pandémie de Covid-19
- Variants du SARS-CoV-2
- Test diagnostique du SARS-CoV-2
- SARS-CoV-2 chez les animaux
- Coronavirus lié au syndrome respiratoire aigu sévère (espèce)
- Enzyme de conversion de l'angiotensine 2 (ACE2), porte d'entrée du virus
- Virologie
- Syndrome respiratoire aigu sévère (SRAS)
- SARS-CoV (coronavirus du SRAS)
- Coronavirus du syndrome respiratoire du Moyen-Orient
Liens externes
- Ressources relatives à la santé :
- (en) Medical Subject Headings
- (cs + sk) WikiSkripta
- Ressources relatives au vivant :
- CDC chinois (Chinese Center for Disease Control and Prevention)
- Institut de Biologie des Agents pathogènes (Institute of Pathogen Biology)
- Carte de l'épidémie actualisée en temps réel par le Center for Systems Science and Engineering de la Johns Hopkins University de Baltimore
- (en-US) Gardner L., « Mapping the Wuhan Coronavirus (2019-nCoV) », sur systems.jhu.edu, (consulté le )
- Questions fréquentes sur les nouveaux coronavirus et Rapports quotidiens de situation sur le nouveau Coronavirus (2019-nCoV) [en anglais] par l'Organisation Mondiale de la Santé
- Fiche-maladie régulièrement actualisée par l'Institut Pasteur
- « Coronavirus 2019-nCoV : le point sur la situation en France et dans le monde », sur le site du Dictionnaire Vidal (consulté le ).
- "Analyses bioinformatiques du coronavirus 2019-nCoV : pourquoi et comment ?", article rédigé par deux bioinformaticiens sur bioinfo-fr.net, 05 février 2020
- "Conseil scientifique COVID-19", Avis du Conseil scientifique COVID-19 (.fr)
- "Infection au nouveau Coronavirus (SARS-CoV-2), COVID-19, France et Monde", Chiffres officiels français, des derniers jours.
- Zsuzsanna Varga, Andreas J. Flammer, Peter Steiger, Martina Haberecker, Rea Andermatt, Annelies Zinkernagel et al., « Electron microscopy of SARS-CoV-2: a challenging task – Authors' reply », (consulté le ).