Haplogroupe T (Y-ADN)
En génétique humaine, l’haplogroupe T est un haplogroupe de l'ADN du chromosome Y humain. De 2002 à 2008, il fut connu comme haplogroupe K2. Ne pas confondre avec l'haplogroupe T de l'ADN mitochondrial, du même nom.
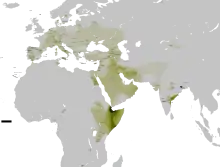
| Date d'origine | 39 000-48 000 années avant le présent |
|---|---|
| Place d'origine | Asie de l'Ouest[1] |
| Ancêtre | LT |
| Descendants | T1, T* |
| Mutations définies | L206, M184/Page34/USP9Y+3178, M193, M272, Page129 |
| Plus hautes fréquences | Kurukh, Bauris, Chians, Allemands Stilfser/Tyroliens, Somalis, Arabes de Somalie et de Djibouti, Saccensi/Siciliens, Eivissencs, Juifs du Nord-Est Portugal, Rajus, Mahli, Zoroastriens de Kerman, Bakhtiaris, Sud-Égyptiens |
Le polymorphisme à évènement unique qui définit ce clade est généralement considéré comme un polymorphisme nucléotidique singulier (sigle anglais SNP) M184. D'autres SNP, L206, M193, M272 sont couramment considérés comme équivalents par la phylogénétique.
Origines
« On pense que T1-M70 originaire d'Asie après l'émergence des mutations K-M9 (45–30 ky) (Underhill et al. 2001a) et T-M184. Comme on peut déduire des données collectées (Underhill et al. 2000; Cruciani et al. 2002; Semino et al. 2002; present study), Les individus T1-M70, quelque temps plus tard, allèrent vers le sud en Afrique. Alors que ce haplogroupe a une fréquence relativement haute en Égypte, Oman, Tanzanie, Éthiopie ([Scozzari et al. 1997, 1999])" »
— J. R. Luis et al.: The Levant versus the Horn of Africa: Evidence for Bidirectional Corridors of Human Migrations (Errata), American Journal of Human Genetics, 74: 532-544.
L'analyse de la structure de son réseau en Europe suggère que l'haplogroupe T est « un ancien lignage indigène européen diversifié, plutôt que des immigrants récents du Moyen-Orient ou d'Afrique » (King et al. 2007:585)[2]
La présence en Europe de lignages appartenant aux subclades T1a ou T1a1 (Ancien T1b) reflète probablement de multiples épisodes de flux génique. Les haplogroupes T1a* sont issus probablement d'un flux génique plus ancien[3].
Distribution haplogroupe T1
L'haplogroupe T1 (M70, M184, M193, M272) est trouvé à une petite majorité chez les indigènes Kurukh, Bauris & Lodha en Inde; et une forte minorité de Rajus et Mahli en Inde, Somalis, Sud Égyptiens et Foulbé en Afrique, Chians, Allemands Stilfser/Tyroliens Saccensi/Siciliens, Eivissencs et Juifs du Nord-Est Portugal en Europe, Zoroastriens, Bakhtiaris au Moyen-Orient, et les Xibes en Extrême-Orient.
Puisque l'haplogroupe T1 n'est pas associé avec les lignages R1, G et J qui sont entrés en Afrique venant d'Eurasie relativement récemment, Luis et al. (2004) suggère que la présence du clade sur le continent africain peut, comme pour les représentants de R1b-V88*, pointer à une plus ancienne arrivée depuis l'Asie. Le Levant plus que l'Arabie du Sud apparaît être la principale route d'accès, car les haplogroupes égyptiens et turcs sont considérablement plus vieux en âge (13 700 avant présent et 9 000 avant présent, respectivement) que ceux trouvés à Oman (seulement 1 600 avant présent). Selon les auteurs, la distribution moderne suivant un modèle en tâches pour l'haplogroupe T1 à l'intérieur de l'Afrique peut donc représenter les restes d'une présence précoce plus étendue du clade. Les expansions plus tardives de populations portant des lignages E1b1b, E1b1a, G et J peuvent les avoir fait très largement surpasser en importance numérique les porteurs des clades T dans de nombreuses régions[4].
Mendez et al. (2011) ont observé qu'une notable fréquence d'un échantillon Lemba d'Afrique du Sud (18 %) portait exclusivement l'haplogroupe T1b-L131 qui est considéré originaire du Proche-Orient. T1b* partage une même période estimée d'expansion avec les T1* Somalis[3].
La distribution de l'haplogroupe T1 dans beaucoup de parties de l'Europe est diffuse ou régionalisée ; par exemple, l'haplogroupe T1 fut trouvé pour 1,7 % (10/591) dans un ensemble de six échantillons d'hommes du Sud-Ouest de la Russie, mais il était complètement absent d'un ensemble de huit échantillons totalisant 637 individus de la moitié nord de la Russie européenne[5].
Distribution haplogroupe T (hors T1)
FTDNA, une société commerciale de généalogie génétique expose une carte qui montre une relativement haute fréquence de l'haplogroupe T chez quelques Aborigènes australiens. Probablement ces populations correspondent à celles antérieurement rapportées dans plusieurs études comme K*(M9) avec une fréquence proche de 30 % en Australie du Nord. Selon FTDNA, leur haplogroupe T est défini par le SNP M184 tandis que M70 définit T1.
Asie du Sud
L'haplogroupe T1-M70 a été détecté chez :
| Population | Langue | Région | Membres/Nombre | Pourcentage | Source | Notes |
| Kurukh | Yerukala (dravidien) | Andhra Pradesh | 10/18 | 55,6 % | [6] | |
| Bauris | Bengali (indo-aryen) | Bengale-Occidental | 10/19 | 52,6 % | [6] | K* a 6/19, si M70- mais M184+, alors pourrait être 84,2 % |
| Lodhas | Lodha (Sora–Juray–Gorum munda) | Bengale-Occidental | 2/4 | 50 % | [6] | |
| Rajus | Telougou (dravidien) | Andhra Pradesh | 3/19 | 15,9 % | [6] | |
| Maheli | Mahali (Kherwari munda) | Bengale-Occidental | 2/13 | 15,3 % | [6] | |
| Chenchus | Chenchu (dravidien) | Andhra Pradesh | 3/20 | 15 % | [6] | K* a 7/20, si M70- mais M184+, alors pourrait être 50 % |
| Kare Vokkal | Kannada (dravidien) | Karnataka | 4/30 | 13,3 % | [7] | K* a 3/30, si M70- mais M184+, alors pourrait être 23,3 % |
| Banjaras | Lambadi (indo-aryen) | Andhra Pradesh | 2/18 | 11,1 % | [6] | |
| Gonds | Gondi (dravidien) | South Uttar Pradesh | 4/38 | 10,6 % | [8] - [9] | |
| Gonds | Gondi (dravidien) | Madhya Pradesh | 10/139 | 7,2 % | [8] - [9] | |
| Indiens | langues en Inde | Inde du Sud | 18/305 | 5,9 % | [6] | |
| Maheli | Mahali (Kherwari munda) | Jamshedpur du Jharkhand; Purulia, Midnapore & autres endroits du Bengale-Occidental | 2/38 | 5,3 % | [6] - [10] | Deux échantillons d'études différentes groupés ensemble |
| Chenchus | Chenchu (Dravidien) | Andhra Pradesh | 3/61 | 4,9 % | [6] - [11] | Échantillons de Trivedi et al. and Kivisild et al. |
| Banjaras | Lambadi (indo-aryen) | Andhra Pradesh | 2/53 | 3,8 % | [6] - [11] | Deux échantillons d'études différentes groupés ensemble |
| Indiens | Langues en Inde | Inde de l'Est | 14/367 | 3,8 % | [6] | |
| Gujaratis | Gujarati (Indo-Aryen) | Gujarat | 1/29 | 3,4 % | [11] | |
| Lodha | Lodha (Sora–Juray–Gorum Munda | Midnapore et autres endroits Bengale-Occidental | 2/71 | 2,8 % | [6] - [10] - [12] | Trois échantillons d'études différentes groupés ensemble |
Avec K-M9+, non confirmé mais probablement T1-M70+ : 56,6 % (30/53) des Kunabhis au Karnataka[13], 32,5 % (13/40) des Kammas en Andhra Pradesh[14], 26,8 % (11/41) de Brahmanes en Visakhapatnam[14], 25 % (1/4) de Kattunaiken en Inde du Sud[15], 22,4 % (11/49) de Telougous en Andhra Pradesh[16], 20 % (1/5) de Ansâri en Asie du Sud[17], 10 % (2/20) de Poroja en Andhra Pradesh[14], 9,8 % (5/51) de Pandits au Cachemire[8], 8,2 % (4/49) de Gujars au Cachemire[8], 7,7 % (1/13) de Siddis (migrants venus d'Éthiopie) en Andhra Pradesh[14], 5,5 % (3/55) de Adi en Inde du Nord[18], 5,5 % (7/128) de Pardhans à Adilabad[16], 5,3 % (2/38) de Brahmanes du Bihar[8], 4,3 % (1/23) de Bagatas en Andhra Pradesh[14], 4,2 % (1/24) de Valmiki en Andhra Pradesh[14], 3,6 % (2/56) de Syed en Asie du Sud[17], 3,1 % (1/32) de Brahmanes au Maharashtra[8], 3,1 % (2/64) de Brahmanes au Gujarat[8], 2,9 % (1/35) de Rajputs en Uttar Pradesh[19], 2,3 % (1/44) de Brahmanes au Peruru[14], et 1,7 % (1/59) de Manghi au Maharashtra[16].
Aussi dans les Desasth-Brahmanes au Maharashtra (1/19 ou 5,3 %) et de Chitpavan-Brahmanes au Konkan (1/21 ou 4,8 %) de Chitpavan-Brahmanes au Konkan (2/66 ou 3 %).
Europe
Avec K-M9+, non confirmé, mais probablement T1-M70+ : 14 % (3/23) de Russes de l'oblast de Yaroslavl[40], 12,5 % (3/24) d'Italiens à Matera[41], 10,3 % (3/29) d'Italiens à Avezzano[41], 10 % (3/30) de Tyroliens du Val di Non[41], 10 % (2/20) d'Italiens de Pescara[41], 8,7 % (4/46) d[Italians de Bénévent[41], 8,3 % [4/48) d'Italiens de Cilento[41], 8,3 % (7/84) d'Italiens de l'Ouest Campanie[42], 7,8 % (4/51) d'Italiens du Sud Latium[42], 7,4 % (2/27) d'Italiens de Paola en Calabre[41], 7,3 % (11/150) d'Italiens en Centre-Sud Italie[43], 7,1 % (8/113) de Serbes en Serbie[44], 7 % (6/86) de Sardes à Tempio[45], 4,7 % (2/42) d'Aroumains en Roumanie[46], 3,7 % (3/82) d'Italiens à Biella[47], 3,7 % (1/27) d'Andalous de la province de Cordoue[28], 3,3 % (2/60) de Léonais de la province de León|[28], 3,2 % (1/31) d'Italiens à Postua[47], 3,2 % (1/31) d'Italiens à Cavaglià[47], 3,1 % (3/97) de Calabrais à Reggio de Calabre,|[33]. 2,8 % (1/36) de Russes dans une partie de la Russie[48], 2,8 % (2/72) d'Italiens en Sud Apulie[49], 2,7 % (1/37) de Calabrais à Cosenza[50], 2,6 % (3/114) de Serbes à Belgrade[51], 2,5 % (1/40) de Russes à Pskov[40], 2,4 % (1/42) de Russes à Kalouga[40], 2,2 % (2/89) de Transylvains à Miercurea-Ciuc[52], 2,2 % (2/92) d'Italiens à Trino, province de Verceil[47], 1,9 % (2/104) d'Italiens à Brescia[53], 1,9 % (2/104) de Roumains en Roumanie[54], 1,7 % (4/237) de Serbes et de Monténégrins en Serbie et au Monténégro[55], 1,7 % (1/59) d'Italiens dans les Marches[49], 1,7 % (1/59) de Calabrais à Catanzaro[50], 1,6 % (3/183) de Grecs en Grèce du Nord[56], 1,3 % (2/150) de Suisses-allemands dans le canton de Zurich[57], 1,3 % (1/79) d'Italiens en Sud Toscane et Nord Latium[49], 1,1 % (1/92) de Néerlandais à Leyde[58], 0,8 % (1/132) d "Andalous" dans le Nord-Ouest de la Tunisie[59], 0,5 % (1/185) de Serbes à Novi Sad (Voivodine)[60], 0,5 % (1/186) de Polonais en Polésie[61] et 0,4 % (1/234) d'Allemands à Halle, Saxe-Anhalt[62].
D'autres endroits où on a trouvé une significative proportion d'individus haplogroupe T ; Trentin (2/67 ou 3 %), vallée de l'Isarco (1/34 ou 2,9 %), Münster (3/102 ou 2,9 %), Mariña de Lugo (1/34 ou 2,9 %), Heraklion (3/104 ou 2,9 %), Roslavl (3/107 ou 2,8 %), Munich (3/112 ou 2,7 %), Ourense (1/37 ou 2,7 %), Livny (3/110 or 2,7 %), Biella (3/114 ou 2,6 %), Entre Douro et Vouga (6/228 ou 2,6 %), Porto (3/118 ou 2,5 %), Urbino (1/40 ou 2,5 %), Péninsule Ibérique (16/629 ou 2,5 %), Blekinge/Kristianstad (1/41 ou 2,4 %), Biélorussie (1/41 ou 2,4 %), Modène (3/130 ou 2,3 %), îles Féroé (2/89 ou 2,2 %), République tchèque et Slovaquie (1/45 or 2,2 %), Provence-Alpes-Côte d'Azur (1/45 ou 2,2 %), Pristen (1/45 ou 2,2 %), Cáceres (2/91 ou 2,2 %), Brac (1/47 ou 2,1 %), Satakunta (1/48 ou 2,1 %), Ouest-Croatie (2/101 ou 2 %), Ukraine (1/50 ou 2 %), Greifswald (2/104 ou 1,9 %), Moldaves de Sofia (1/54 ou 1,9 %), Uppsala (1/55 ou 1,9 %), Lublin (2/112 ou 1,8 %), Serpa dans le district de Beja (1/54 ou 1,8 %), Grecs Macédoine grecque|Macédoniens] (1/57 ou 1,8 %), Nea Nikomedeia (1/57 ou 1,8 %), Sesklo/Dimini (1/57 ou 1,8 %), Lerne/Franchthi (1/57 ou 1,8 %), Açores (2/121 ou 1,7 %), Viana do Castelo (1/59 ou 1,7 %), Brabant-Septentrional (1,54 %), Midi-Pyrénées (1/67 ou 1,5 %), Belgorod (2/143 ou 1,4 %), Sardaigne (1/77 ou 1,3 %), district de Burzyansky en Bachkirie (1/80 ou 1,3 %), Hambourg (2/161 or 1,2 %), Cosaques du Kouban (1/90 ou 1,1 %) Rostock (1/96 ou 1 %), Magdebourg (1/100 ou 1 %) et Stockholm (2/228 ou 0,9 %)[63] - [64] - [65] - [66] - [67] - [68] - [6] - [42] - [31] - [69] - [70] - [71] - [72] - [73] - [74] - [75] - [76] - [77] - [78] - [79] - [80] - [81] - [82] - [83] - [84] - [85] - [20] - [86] - [87] - [88] - [89] - [90] Selon les résultats du projet ADN Italie de la compagnie FTDNA de tests génétiques, 3,9 % d'Italiens appartiennent à cet haplogroupe[91]. Approximativement 3 % de Juifs séfarades et 2 % de Juifs Ashkenazim appartiennent à l'haplogroupe T[92].
Moyen-Orient et Caucase
Non confirmé mais probablement T1-M70+ : 28 % (7/25) des Lezguiens au Daghestan[95], 21,7 % (5/23) des Ossètes à Zamankul[109], 14 % (7/50) des Iraniens à Ispahan[95], 13 % (3/23) des Ossètes à Zil'ga[109], 12,6 % (11/87) de Kurdes Kurmandji dans l'est de la Turquie[110], 11,8 % (2/17) d ArabesPalestiniens en Palestine[17], 8,3 % (1/12) d'Iraniens de Chiraz[111], 8,3 % (2/24) d'Ossètes à Alagir[109], 8 % (2/25) of Kurdes Kurmandji en Géorgie[110], 7,5 % (6/80) d'Iraniens à Téhéran[95] - [112], 7,4 % (10/135) d ArabesPalestiniens dans un village en Israël[17], 7 % (10/143) of d ArabesPalestiniens ein Israël et en Palestine[17], 5 % (1/19) de Tchétchènes en Tchétchénie[95] - [112], 4,2 % (3/72) d'Azeris en Azerbaïdjan[95] - [112], 4,1 % (2/48) d'Iraniens à Ispahan[112], 4 % (4/100) d'Arméniens en Arménie[95] - [112], 4 % (1/24) de Bédouins en Israël[17] et 2,6 % (1/39) de Turcs à Ankara[112].
Afrique
Non confirmé mais probablement T1-M70+ : 9,7 % (3/31) de Datogs en Centre Tanzanie[125], 5,8 % (4/69) de Kordofaniens au Kordofan[17], 5,6 % (1/18) de Touaregs à Gorom-Gorom[132], 4,8 % (5/105) de Tunisiens à Sfax[133], 4,8 % (3/63) de Libyens à Tripoli[134], 2,6 % (1/39) de Hutus au Rwanda[135] 2,1 % (1/47) de Berbères à Sejnane[136], 1,9 % (1/53) d'Ovimbundus en Angola[137], et 1,5 % (1/68) de Mozabites à Ghardaia[138].
Extrême-Orient
| Population | Langue | Région | Membres/Nombre | Pourcentage | Source | Notes |
| Xibe | Xibe (Langues toungouses) | Xinjiang | 1/8 | 12,5 % | [139] - [140] | |
| Kazakhs | Kazakh (langues turques) | Sud-Ouest Altaï | 1/30 | 3,3 % | [141] | |
| Ouïghours | Ouïghour (langues turques) | Xinjiang | 1/48 | 2,1 % | [142] |
Non confirmé mais probablement T1-M70+ : 4,9 % (2/41) de Xibe du [Xinjiang][143], 2 % (4/204) de Hui au Liaoning[144], et 0,9 % (1/113) de Bidayuh au Sarawak[145].
Subclades
Arbre
Cet arbre phylogénétique des subclades de l'haplogroupe T se base sur l'arbre ISOGG 2011.
- T (L206, M184/PAGES00034/USP9Y+3178, M193, M272, PAGES00129)
- T1 (M70/PAGES00046, PAGES00078)
- T1a (L162/PAGES00021, L299)
- T1a1 (L208/PAGES00002)
- T1a1a (M320)
- T1a1b (p. 77)
- T1a1c (p. 330)
- T1a1d (p. 321)
- T1a1d1 (p. 317)
- T1a1 (L208/PAGES00002)
- T1b (L131)
- T1b1 (p. 322, p. 328)
- T1b1a (p. 327)
- T1b1 (p. 322, p. 328)
- T1a (L162/PAGES00021, L299)
- T1 (M70/PAGES00046, PAGES00078)
Voir aussi
Références
- Underhill PA, et al. (2001), Maori origins, Y-chromosome haplotypes and implications for human history in the Pacific., Hum Mutat. 2001 Apr;17(4):271-80.
- Turi E. King et al, "Thomas Jefferson’s Y Chromosome Belongs to a Rare European Lineage, " "AMERICAN JOURNAL OF PHYSICAL ANTHROPOLOGY" (2007),
- F.L. Mendez et al., "Increased Resolution of Y Chromosome Haplogroup T Defines Relationships among Populations of the Near East, Europe, and Africa" BioOne Human Biology 83(1):39-53, (2011)
- J. R. Luis et al.: The Levant versus the Horn of Africa: Evidence for Bidirectional Corridors of Human Migrations (Errata), American Journal of Human Genetics, 74: 532-544.
- Oleg Balanovsky, Siiri Rootsi, Andrey Pshenichnov, Toomas Kivisild, Michail Churnosov, Irina Evseeva, Elvira Pocheshkhova, Margarita Boldyreva, Nikolay Yankovsky, Elena Balanovska, et Richard Villems, "Two Sources of the Russian Patrilineal Heritage in Their Eurasian Context", The American Journal of Human Genetics, 82, 236–250, janvier 2008. Les Russes du Sud-Ouest étaient des villes suivantes : Roslavl, Livny, Pristen, Repievka et Belgorod; avec compris les Cosaques du Kouban de la République des Adygués.
- R. Trivedi, Sanghamitra Sahoo, Anamika Singh, G. Hima Bindu, Jheelam Banerjee, Manuj Tandon, Sonali Gaikwad, Revathi Rajkumar, T Sitalaximi, Richa Ashma, G. B. N. Chainy et V. K. Kashyap, "High Resolution Phylogeographic Map of Y-Chromosomes Reveal the Genetic Signatures of Pleistocene Origin of Indian Populations"
- Anish M. Shah et al., ["Indian Siddis: African Descendants with Indian Admixture"] 2011
- S.Sharma et al. "The Indian origin of paternal haplogroup R1a1* substantiates the autochthonous origin of Brahmins and the caste system", Journal of Human Genetics (2009)
- Clyde Winters "Y-Chromosome evidence of an African origin of Dravidian agriculture", International Journal of Genetics and Molecular Biology (2010)
- Vikrant Kumar et al. "Y-chromosome evidence suggests a common paternal heritage of Austro-Asiatic populations"
- T. Kivisild et al. "The Genetic Heritage of the Earliest Settlers Persists Both in Indian Tribal and Caste Populations"
- Sanghamitra Sengupta et al. "Polarity and Temporality of High-Resolution Y-Chromosome Distributions in India Identify Both Indigenous and Exogenous Expansions and Reveal Minor Genetic Influence of Central Asian Pastoralists"
- K.Tangaraj et al. "Y-Chromosomal STR Haplotypes in Two Endogamous Tribal Populations of Karnataka, India", "'American Academy of Forensic Sciences'" (2007)
- G. Venkata et al. "Y-chromosome SNP haplotypes suggest evidence of gene flow among caste, tribe, and the migrant Siddi populations of Andhra Pradesh, South India"]
- R. Cordaux et al. "Independent Origins of Indian Caste and Tribal Paternal Lineages"]
- I. Thanseem et al. "Genetic affinities among the lower castes and tribal groups of India: inference from Y chromosome and mitochondrial DNA S1 ", "'BMC Genetics'" (2006)
- Elise M. S. Belle et al. "Y chromosomes of self-identified Syeds from the Indian subcontinent show evidence of elevated Arab ancestry but not of a recent common patrilineal origin"]
- Richard Cordaux et al. "The Northeast Indian Passageway: A Barrier or Corridor for Human Migrations?", Society for Molecular Biology and Evolution (2004)
- P. Majumder et al., Ethnic populations of India as seen from an evolutionary perspective]
- V. Onofri et al., "Y-chromosome genetic structure in sub-Apennine populations of Central Italy by SNP and STR analysis", Springer-Verlag (2007)
- C. Robino et al., "Y-chromosomal STR haplotypes in a population sample from continental Greece, and the islands of Crete and Chios", Forensic Science International (2004)
- I. Pichler et al., "Genetic Structure in Contemporary South Tyrolean Isolated Populations Revealed by Analysis of Y-Chromosome, mtDNA, and Alu Polymorphisms", Human Biology, août 2006, v. 78, no 4, p. 441–464.
- Stefania Turrina et al., "Y-chromosomal STR haplotypes in a Northeast Italian population sample using 17plex loci PCR assay", Springer-Verlag (2006)
- Cornelia Di Gaetano, Nicoletta Cerutti, Francesca Crobu et al., "Differential Greek and northern African migrations to Sicily are supported by genetic evidence from the Y chromosome", European Journal of Human Genetics (2009) 17, 91–99
- Pierre A. Zalloua, Daniel E. Platt, Mirvat El Sibai et al., "Identifying Genetic Traces of Historical Expansions: Phoenician Footprints in the Mediterranean, " American Journal of Human Genetics (2008), doi:10.1016/j.ajhg.2008.10.012. cf. "Table S3. Haplogroup Numbers and Frequencies" in the online data supplement.
- Inês Nogueiro et al., Phylogeographic analysis of paternal lineages in NE Portuguese Jewish communities, Am J Phys Anthropol. 2009 Nov 16.
- Rosaria Scozzari et al., Human Y-chromosome variation in the Western Mediterranean area: implications for the peopling of the region, Human Immunology 2001
- Carlos Flores, Nicole Maca-Meyer, Ana M González, Peter J Oefner, Peidong Shen, Jose A Pérez, Antonio Rojas, Jose M Larruga et Peter A Underhill, "Reduced genetic structure of the Iberian peninsula revealed by Y-chromosome analysis: implications for population demography, " European Journal of Human Genetics (2004) 12, 855–863 & 2004 Nature Publishing Group
- Laisel Martinez, Peter A Underhill, Lev A Zhivotovsky et al., "Paleolithic Y-haplogroup heritage predominates in a Cretan highland plateau, " European Journal of Human Genetics (2007) 15, 485–493
- N. Maca-Meyer, P. Sánchez-Velasco, C. Flores et al., "Y Chromosome and Mitochondrial DNA Characterization of Pasiegos, a Human Isolate from Cantabria (Spain)", Annals of Human Genetics (2003) 67, 329–339
- Sandra Beleza et al."Micro-Phylogeographic and Demographic History of Portuguese Male Lineages", "Annuals of Human Genetics" (2006)
- Giuseppe Calcagno et al. "Y-Chromosome short tandem repeat (STR) haplotypes in a Campania population sample", "'Clin Chem Lab Med'" (2005)
- V.Rodriguez et al., "Genetic sub-structure in western Mediterranean populations revealed by 12 Y-chromosome STR loci, " International Journal of Legal Medicine (2008), "Table S2. Y-chromosome STR haplotypes in 554 unrelated men from the western Mediterranean region" in the online data supplement.
- Decorte R. et al., "'YHRD'"
- Ambrosio B. et al., "'Y-STR genetic diversity in autochthonous Andalusians from Huelva and Granada provinces (Spain), " Forensic Sci International Genetics (2011)
- R. Montiel et al."Analysis of Y-chromosome Variability and its Comparison with mtDNA Variability Reveals Different Demographic Histories Between Islands in the Azores Archipelago (Portugal)", Annals of Human Genetics (2005)
- Jan Zastera et al., "Assembly of a large Y-STR haplotype database for the Czech population and investigation of its substructure, " Forensic Science International: Genetics (2010)
- Kristin L. Young et al., "Paternal Genetic History of the Basque Population of Spain, " Human Biology, 83(4):455-475 (2011)
- Susann Seiberling et al., "Allelverteilung Y-chromosomaler Short TandemRepeats in Vorpommern, " Greifswald, Institut für Medizinische Mikrobiologie (2005)
- Boris A. Malyarchuk et al. "Differentiation of Mitochondrial DNA and Y Chromosomes in Russian Populations", Human Biology, Volume 76, Number 6 (2005)
- F. Di Giacomo et al. "Clinal patterns of human Y chromosomal diversity in continental Italy and Greece are dominated by drift and founder effects", " ScienceDirect (2003)
- Capelli et al. "Y chromosome variation in the Italian peninsula is clinal and supports an admixture model for the Mesolithic-Neolithic encounter", "'ScienceDirect'" (2006)
- Cesare Rapone "Y chromosome haplotypes in Central-South Italy: Implication for reference database", " Forensic Science International (2006)
- Marijana Pericic "High-Resolution Phylogenetic Analysis of Southeastern Europe Traces Major Episodes of Paternal Gene Flow Among Slavic Populations", " Oxford Journals (2005)
- Contu et al."Y-Chromosome Based Evidence for Pre-Neolithic Origin of the Genetically Homogeneous but Diverse Sardinian", "'PubMed Central'" (2008)
- Bosch et al."Paternal and maternal lineages in the Balkans show a homogeneous landscape over linguistic barriers, except for the isolated Aromuns.", "'University College London'" (2006)
- N. Cerutti et al. "Population data for Y-chromosome STR haplotypes from Piedmont (Italy)", "'Forensic Science International'" (2005)
- "'AMERICAN JOURNAL OF PHYSICAL ANTHROPOLOGY'" (2008)
- Cristian Capelli et al. "A 9-loci Y chromosome haplotype in three Italian populations ", "'Forensic Science International: Genetics'" (2005)
- V. Rodríguez et al. "Genetic sub-structure in western Mediterranean populations revealed by 12 Y-chromosome STR loci", "'J Legal Med'" (2008)
- Lovorka Barac Lauc et al. "Y chromosome STR polymorphisms in a Serbian population sample", "'Forensic Science International'" (2004)
- Balazs Egyed. "Population genetic study in two Transylvanian populations using forensically informative autosomal and Y-chromosomal STR markers", Forensic Science International (2005)
- Nicoletta Cerri et al. "YPopulation data for 12 Y-chromosome STRs in a sample from Brescia (northern Italy)", "'Elsevier Ireland Ltd.'" (2005)
- Ligia Elena Barbarii et al. "Y-chromosomal STR haplotypes in a Romanian population sample", "'Springer-Verlag'" (2003)
- Miljana Stevanovic "Human Y-specific STR haplotypes in population of Serbia and Montenegro", Forensic Science International (2006)
- Leda Kovatsi et al. "Population genetics of Y-chromosome STRs in a population of Northern Greeks", "'Forensic Science International: Genetics'" (2009)
- C. Haas et al. "Y-chromosome STR haplotypes in a population sample from Switzerland (Zurich area)", "'Forensic Science International'" (2005)
- Heike Rodig et al. "Evaluation of haplotype discrimination capacity of 35 Y-chromosomal STR loci", "'Forensic Science International'" (2007)
- Lotfi Cherni et al. "Y-chromosomal STR haplotypes in three ethnic groups and one cosmopolitan population from Tunisia", "'Elsevier Ireland Ltd.'" (2005)
- Igor S. Veselinovic et al. "Allele frequencies and population data for 17 Y-chromosome STR loci in a Serbian population sample from Vojvodina province", "'Forensic Science International'" (2007)
- Witold Pepinski et al. "Population genetics of Y-chromosome STRs in a population of Podlasie, northeastern Poland", "'Elsevier Ireland Ltd.'" (2004)
- Uta-Dorothee Immel et al. "Y chromosome polymorphisms and haplotypes in South Saxony-Anhalt (Germany)", Forensic Science International (2005)
- Alicia M Cadenas, Lev A Zhivotovsky, Luca L Cavalli-Sforza, Peter A Underhill and Rene J Herrera, "Y-chromosome diversity characterizes the Gulf of Oman, " European Journal of Human Genetics (2007), 1 - 13
- M. Regueiro et al.: "Iran: Tricontinental Nexus for Y-Chromosome Driven Migration, " Human Heredity, 2006, vol. 61, p. 132–43.
- Cinnioglu, Cengiz, et al., "Excavating Y-Chromosome Haplotype Strata in Anatolia, " Human Genetics, 2004, vol. 114, p. 127–48.
- Sanghamitra Sahoo, Anamika Singh, G. Himabindu, Jheelam Banerjee, T. Sitalaximi, Sonali Gaikwad, R. Trivedi, Phillip Endicott, Toomas Kivisild, Mait Metspalu, Richard Villems, et V. K. Kashyap, "A prehistory of Indian Y chromosomes: Evaluating demic diffusion scenarios, " Proceedings of the National Academy of Sciences of the United States of America. Published online on January 13, 2006, 10.1073/pnas.0507714103. (cf. Supporting Figure 3 in online data supplement)
- Pierre A. Zalloua, Yali Xue, Jade Khalife, Nadine Makhoul, Labib Debiane, Daniel E. Platt, Ajay K. Royyuru, Rene J. Herrera, David F. Soria Hernanz, Jason Blue-Smith, R. Spencer Wells, David Comas, Jaume Bertranpetit, Chris Tyler-Smith, and The Genographic Consortium, "Y-Chromosomal Diversity in Lebanon Is Structured by Recent Historical Events, " The American Journal of Human Genetics 82, 873–882, avril 2008.
- Sadaf Firasat, Shagufta Khaliq, Aisha Mohyuddin, Myrto Papaioannou, Chris Tyler-Smith, Peter A Underhill and Qasim Ayub, "Y-chromosomal evidence for a limited Greek contribution to the Pathan population of Pakistan, " European Journal of Human Genetics (2007) 15, 121–126.
- Mirvat El Sibai et al., "Geographical Structure of the Y-chromosomal Genetic Landscape of the Levant: A coastal-inland contrast", Annals of Human Genetics (2009), "Table S1. General information and haplotype and haplogroup assignment of samples genotyped in this study" in the online data supplement.
- Battaglia et al. "Y-chromosomal evidence of the cultural diffusion of agriculture in southeast Europe", " European Journal of Human Genetics (in press) (2009)
- Rita Gonçalves et al."Y-chromosome Lineages from Portugal, Madeira and A¸cores Record Elements of Sephardim and Berber Ancestry", "'University of Madeira'" (2005)
- Karlsson et al."Y-chromosome diversity in Sweden – A long-time perspective", "European Journal of Human Genetics" (2006)
- Varzari et al."Searching for the Origin of Gagauzes: Inferences from Y-Chromosome Analysis", "American Journal of Human Biology" (2008)
- Semino et al."The Genetic Legacy of Paleolithic Homo sapiens sapiens in Extant Europeans: A Y Chromosome Perspective", "Science" (2000)
- Kivisild et al."The Genetic Heritage of the Earliest Settlers Persists Both in Indian Tribal and Caste Populations", "American Journal of Human Genetics" (2003)
- Tove H. Jorgensen et al."The origin of the isolated population of the Faroe Islands investigated using Y chromosomal markers", "Human Genetics" (2003)
- Verónica Gomes et al."Refining the analysis of Y-chromosomal diversity in Alentejo (Portugal)", "Elsevier Ireland Ltd" (2008)
- Marin A.et al." Biodemographic and molecular analysis of an isolated Alpine population (Postua)", "International Journal of Anthropology" (2005)
- M.Brion et al." Hierarchical analysis of 30 Y-chromosome SNPs in European populations", "Springer-Verlag" (2004)
- G.Ferri et al." Slow and fast evolving markers typing in Modena males (North Italy)", "'Forensic Science Internationa'" (2009)
- R.J. King et al., "Differential Y-chromosome Anatolian Influences on the Greek and Cretan Neolithic, " Annals of Human Genetics (2008)
- D. Kasperaviciute et al., "Y Chromosome and Mitochondrial DNA Variation in Lithuanians, " Annals of Human Genetics (2004)
- T. Lappalainen, U. Hannelius et al., "Population Structure in Contemporary Sweden—A Y-Chromosomal and Mitochondrial DNA Analysis, " Annals of Human Genetics (2009)
- P. Grignani et al., "Highly informative Y-chromosomal haplotypes by the addition of three new STRs DYS437, DYS438 and DYS439," Springer-Verlag (2000)
- S. Gaikwad et al., "Molecular insight into the genesis of ranked caste populations of western India based upon polymorphisms across non-recombinant and recombinant regions in genome, " Genome Biology (2005)
- Zoë H. Rosser et al."Y-Chromosomal Diversity in Europe Is Clinal and Influenced Primarily by Geography, Rather than by Language", "'American Journal of Human Genetics'" (2000)
- Mark G Thomas et al."New genetic evidence supports isolation and drift in the Ladin communities of the South Tyrolean Alps but not an ancient origin in the Middle", European Journal of Human Genetics (2008)
- Irene Pichler et al., "Drawing the history of the Hutterite population on a genetic landscape: inference from Y-chromosome and mtDNA genotypes", "'European Journal of Human Genetics'" (2010)
- E. Ramos-Luis et al., "Phylogeography of French male lineages, " Forensic Science International: Genetics Supplement Series (2009)
- Maria Gaibar et al. "STR genetic diversity in a Mediterranean population from the south of the Iberian Peninsula", Annals of Human Biology (2010)
- Italy DNA Project blog, "What a difference a year makes" (posted Tuesday, September 04, 2007), based on data from the Italy DNA Project at Family Tree DNA,
- Nicholas Wade, "Study Raises Possibility of Jewish Tie for Jefferson," The New York Times (28 février 2007)
- Zahra Lashgary et al, "Y chromosome diversity among the Iranian religious groups: A reservoir of genetic variation", Annals of Human Biology (2011)
- L. Roewer et al, "A Y-STR database of Iranian and Azerbaijanian minority populations, " "Forensic Science International: Genetics 4" (2009),
- Nasidze et al."Mitochondrial DNA and Y-Chromosome Variation in the Caucasus", Annals of Human Genetics (2004)
- Yonan et al., "Y-chromosome diversity in the Assyrian christians, " (2009)
- N. Al-Zahery, O. Semino, G. Benuzzi, C. Magri, G. Passarino, A. Torroni, and A.S. Santachiara-Benerecetti, "Y-chromosome and mtDNA polymorphisms in Iraq, a crossroad of the early human dispersal and of post-Neolithic migrations, " Molecular Phylogenetics and Evolution (2003)
- L. QUINTANA-MURCI et al., "Y-chromosome speci®c YCAII, DYS19 and YAP polymorphisms in human populations: a comparative study", Annals of Human Genetics (1999)
- Mukherjee N., Nebel A., Oppenheim A. and Majumder P. P., "High-resolution analysis of Y-chromosomal polymorphisms reveals signatures of population movements from Central Asia and West Asia into India, " J. Genet. 80, 125–135 (2001)
- Ana Teresa Fernandes et al., "Y-chromosomal STRs in two populations from Israel and the Palestinian Authority Area: Christian and Muslim Arabs, " Forensic Science International: Genetics (2011)
- Michael E. Weale et al., "Armenian Y chromosome haplotypes reveal strong regional structure within a single ethno-national group, " Journal of Human Genetics (2009)
- Khaled K. Abu-Amero, Ali Hellani, Ana M. Gonzalez et al., "Saudi Arabian Y-Chromosome diversity and its relationship with nearby regions, " BMC Genetics 2009, 10:59 doi:10.1186/1471-2156-10-59
- Marc Haber et al., "Influences of history, geography, and religion on genetic structure: the Maronites in Lebanon, " European Journal of Human Genetics 2010
- Mohammad et al. "Genetic structure of nomadic Bedouin from Kuwait", UKPMC (2009)
- M.-R. Rafiee et al., "Analysis of Y-Chromosomal Short Tandem Repeat (STR) Polymorphism in an Iranian Sadat Population", Russian Journal of Genetics (2009)
- Roy M. Regueiro et al., "Iran: Tricontinental Nexus for Y-Chromosome Driven Migration, " Human Heredity 2006, DOI: 10.1159/000093774
- Roy J King et al., "The coming of the Greeks to Provence and Corsica: Y-chromosome models of archaic Greek colonization of the western Mediterranean, " BMC Evolutionary Biology 2011, 11:69 doi:10.1186/1471-2148-11-69
- Oleg Balanovsky et al., "Parallel Evolution of Genes and Languages in the Caucasus Region, " Molecular Biology and Evolution 2011
- Nasidze et al. "Genetic Evidence Concerning the Origins of South and North Ossetians", Annals of Human Genetics (2004)
- Nasidze et al. "MtDNA and Y-chromosome Variation in Kurdish Groups", Annals of Human Genetics (2005)
- R. Spencer Wells et al., "The Eurasian Heartland: A continental perspective on Y-chromosome diversity, " The National Academy of Sciences, 2001
- Nasidze et al. "Haplotypes from the Caucasus, Turkey and Iran for nine Y-STR loci", "'Elsevier Ireland Ltd'" (2003)
- Giuseppe Iacovacci, Eugenia D’Atanasio, Ornella Marini et Alfredo Coppa, « Forensic data and microvariant sequence characterization of 27 Y-STR loci analyzed in four Eastern African countries », Forensic Science International: Genetics, vol. 27, , p. 123–131 (ISSN 1872-4973, DOI 10.1016/j.fsigen.2016.12.015, lire en ligne, consulté le )
- (en) « Variation in Y chromosome, mitochondrial DNA and labels of identity on Ethiopia », sur UCL, Thèse Doctorale
- U.D. Immel et al., "Y-chromosomal STR haplotypes in an Arab population from Somalia" (2009)
- Fulvio Cruciani, Piero Santolamazza, Peidong Shen et al., "A Back Migration from Asia to Sub-Saharan Africa Is Supported by High-Resolution Analysis of Human Y-Chromosome Haplotypes, " American Journal of Human Genetics 70:1197–1214, 2002
- « Insights into the Middle Eastern paternal genetic pool in Tunisia: high prevalence of T-M70 haplogroup in an Arab population »,
- Margurethe Stenersen et al., "Kurdish (Iraq) and Somalian population data for 15 autosomal and 9 Y-chromosomal STR loci, " Elsevier (2004)
- Elizabeth T Wood et al., "Contrasting patterns of Y chromosome and mtDNA variation in Africa: evidence for sex-biased demographic processes, " European Journal of Human Genetics (2005)
- Juan J Sanchez, Charlotte Hallenberg, Claus Børsting, Alexis Hernandez and Niels Morling, "High frequencies of Y chromosome lineages characterized by E3b1, DYS19-11, DYS392-12 in Somali males, " European Journal of Human Genetics (2005) 13, 856–866
- Barbara Arredi, Estella S. Poloni, Silvia Paracchini et al., "A Predominantly Neolithic Origin for Y-Chromosomal DNA Variation in North Africa, " American Journal of Human Genetics 75:338–345, 2004
- Ghada A. Omran et al., "Diversity of 17-locus Y-STR haplotypes in Upper (Southern) Egyptians, " Forensic Science International: Genetics Supplement Series2007
- Cesare de Filippo et al., "Y-chromosomal variation in Sub-Saharan Africa, insights into the history of Niger–Congo groups", Oxford University Press, 2010
- Andreas O. Tillmar et al., "Population data of 12 Y-STR loci from a Somali population" (2009)
- Sarah A. Tishkoff et al. "Supplementary Table 2: Y chromosome genotype data", "'Mol. Biol. Evol.'" (2007)
- Called "Wairak" and misidentified as Bantu in the studies.
- Wanwalai Charoenchote et al. "AmpFℓSTR® Identifiler™ STR Allele Frequencies and PowerPlex® Y-STR Haplotype Frequencies of the Meru Population of Northern Tanzania", University of Nevada (2004)
- Krishna R Veeramah et al. "Little genetic differentiation as assessed by uniparental markers in the presence of substantial language variation in peoples of the Cross River region of Nigeria", BMC Evolutionary Biology (2010)
- Samir Elmrghni et al. "Population genetic data for 17 Y STR markers from Benghazi (East Libya)", "'Forensic Science International: Genetics'" (2011)
- Franz Manni et al. "Y-Chromosome Analysis in Egypt Suggests a Genetic Regional Continuity in Northeastern Africa", Human Biology, volume 74, no 5, octobre 2002, p. 645-658 (2002)
- Verónica Gomes et al. "Digging deeper into East African human Y chromosome lineages", "'Hum Genet'" (2010)
- Luisa Pereira et al. "Linking the sub-Saharan and West Eurasian gene pools: maternal and paternal heritage of the Tuareg nomads from the African Sahel", European Journal of Human Biology (2010)
- Imen Ayadi et al. "Haplotypes for 13 Y-chromosomal STR loci in South Tunisian population (Sfax region)", 'Elsevier Ltd.' (2005)
- U.-D. Immel et al. "Y-chromosomal STR haplotypes in an Arab population from Libya", "'Elsevier B.V.'" (2005)
- ALESSANDRA CAGLIÀ et al. "A Study of Y-Chromosome Microsatellite Variation in Sub-Saharan Africa: A Comparison between FST and RST Genetic Distances", Human Biology, volume 75, no 3. (2003)
- Sabeh Frigi et al. "Data for Y-chromosome haplotypes defined by 17 STRs (AmpFLSTR1 YfilerTM) in two Tunisian Berber communities", "'Elsevier Ireland Ltd.'" (2005)
- Miguel M. Melo et al. "Y-STR haplotypes in three ethnic linguistic groups of Angola population", "'Forensic ScienceInternational:Genetics'" (2010)
- E. Bosch et al. "Y chromosome STR haplotypes in four populations from northwest Africa", Int J Legal Med (2000)
- R.Sanghamitra Sengupta et al, "Polarity and Temporality of High-Resolution Y-Chromosome Distributions in India Identify Both Indigenous and Exogenous Expansions and Reveal Minor Genetic Influence of Central Asian Pastoralists, " "The American Journal of Human Genetics" (2006),
- R. Howard M. Cann et al, "A Human Genome Diversity Cell Line Panel, " Science (2002), "Table 1" in the online data supplement.
- Dulik MC, Osipova LP, Schurr TG, "Y-Chromosome Variation in Altaian Kazakhs Reveals a Common Paternal Gene Pool for Kazakhs and the Influence of Mongolian Expansions, " "PLoS ONE" (2011),
- Zhong et al, "Extended Y-chromosome investigation suggests post-Glacial migrations of modern humans into East Asia via the northern route, " "Society for Molecular Biology and Evolution" (2010),
- Yali Xue, Tatiana Zerjal, Weidong Bao et al., "Male Demography in East Asia: A North–South Contrast in Human Population Expansion Times, " Genetics 172: 2431–2439 (April 2006)
- Rufeng Bai et al. "Y-chromosomal STRs haplotypes in Chinese Hui ethnic group samples", "'Elsevier Ireland Ltd.'" (2008)
- Yuet Meng Chang et al. "Haplotype diversity of 17 Y-chromosomal STRs in three native Sarawak populations (Iban, Bidayuh and Melanau) in East Malaysia 'Elsevier Ireland Ltd.' (2008)