Hybridation entre les humains archaïques et modernes
Il existe des preuves d'hybridation entre les humains archaïques et modernes pendant le Paléolithique moyen et au début du Paléolithique supérieur. Plusieurs épisodes d'hybridation indépendants se sont produits. Certaines populations humaines sont ainsi issues de l'hybridation avec des Néandertaliens, des Dénisoviens, ainsi qu'avec plusieurs espèces humaines encore non identifiées.
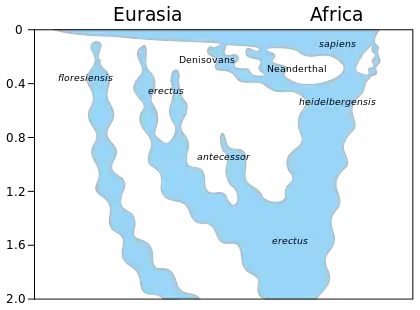
L'ADN néandertalien représente entre 1,5 et 2,1 % du génome des Eurasiens, alors qu'il est absent ou significativement plus rare chez les peuples subsahariens. Dans les populations mélanésiennes actuelles, la part d'ADN dénisovien atteint entre 2,3 et 3,7 %. Des analyses d'ADN non-comparatives récentes – en l'absence de découverte d'une autre espèce – suggèrent que les populations africaines ont également reçu un apport génétique d'une espèce humaine africaine disparue.
Les preuves génétiques montrent que l'évolution humaine ne peut pas être considérée comme une évolution cloisonnée consécutive à la divergence de différentes lignées. Les formes modernes et archaïques se sont en effet croisées de nombreuses fois en Afrique, au Moyen-Orient et en Asie, et le flux génétique allait dans les deux sens[2] - [3]. Certains gènes acquis par hybridation ont notamment pu être sélectionnés chez Homo sapiens en raison de leur caractère adaptatif[2]. Toutefois, l'hybridation n'a pas été jusqu'au brassage complet des différents groupes humains, puisque chacun d'eux semble avoir finalement conservé son identité propre. De ce fait, l'évolution humaine récente se présente plutôt a posteriori comme une évolution de groupes distincts avec des épisodes ponctuels d'hybridation.
Historique
L'hypothèse, portant alternativement le nom de métissage, d'hybridation, de mélange ou de théorie des origines hybrides, a été discutée depuis la découverte des premiers restes de Néandertal au XIXe siècle, bien que les premiers auteurs pensaient que les Néandertaliens étaient des ancêtres directs des hommes modernes. Thomas Huxley suggéra que beaucoup d'Européens portaient des traces d'ancêtres néandertaliens mais considérait les caractéristiques de l'homme de Néandertal comme des traits primitifs. Postulant qu'ils appartenaient à une « étape du développement de l'espèce humaine, les antécédents à la différenciation de toutes les races existantes », il considérait que « nous devrions les retrouver dans la plus basse de ces races, partout dans le monde, et au commencement de chacune d'entre elles »[4].
Hans Peder Steensby, dans son article Racestudier i Danmark (« Études sur les races au Danemark ») de 1907, rejeta la comparaison des Néandertaliens avec des singes ainsi qu'une éventuelle infériorité et, tout en mettant en avant que l'homme moderne avait des origines variées, suggéra la théorie de l'hybridation comme la meilleure explication d'un certain nombre d'observations scientifiques d'alors[5].
Au milieu du XXe siècle, Carleton Coon considérait que les caucasiens avaient deux origines : la première du Paléolithique supérieur (mélange entre l’H. sapiens et l’H. neanderthalensis) et la seconde méditerranéenne (purement H. sapiens). Il répéta sa théorie dans un ouvrage de 1962 appelé « The Origin of Races »[6].
Néandertaliens
Néandertal et Eurasiens
En 2010, le séquençage du génome des Néandertaliens révéla que ces derniers partageaient plus d'allèles avec les populations eurasiennes (par exemple, françaises, han ou papoue-néo-guinéenne) qu'avec les populations subsahariennes (tel que les Yorubas et les San)[7]. D'après l'étude, cet écart de similarité génétique s'expliquerait par un flux de gènes récent des Néandertaliens vers les humains modernes après la migration hors de l'Afrique[7]. En 2013, les mêmes chercheurs estimèrent la part de gènes néandertaliens chez les Eurasiens actuels à 1,5 – 2,1 %[8]. De 20 à 40 % du génome néandertalien aurait survécu au sein des populations eurasiatiques modernes[9] - [10] - [11].
Sur la base du spectre de la fréquence allélique, il a été montré que l'hybridation récente était le modèle le plus probable pour expliquer le différentiel de similarités génétiques entre Africains et non-Africains modernes par rapport aux Néandertaliens[7] - [12]. L'hybridation récente est également soutenue par les analyses des déséquilibres de liaison[13].
Au travers de l'étendue des déséquilibres de liaison, il a été estimé que le transfert d'allèles entre les Néandertaliens et les premiers ancêtres des Eurasiens a eu lieu entre 65 000 et 47 000 ans AP[13]. En conjonction avec les preuves fossiles et archéologiques, le transfert aurait eu lieu quelque part dans l'ouest de l'Eurasie, probablement au Moyen-Orient[13].
2 % d’ADN Néandertalien ont été découverts dans le génome de l'Homme de Ust-Ishim. La date du croisement Néandertal - Homo sapiens remonterait de 7 000 à 13 000 ans avant l’existence de l'Homme de Ust-Ishim, daté d'environ 45 000 ans AP[14].
Une étude révèle que les gènes néandertaliens identifiés dans le génome des Européens actuels ont une structure qui n'est pas proche des gènes des Néandertaliens européens tardifs[15]. Le génome néandertalien des Eurasiens actuels est plus proche des Néandertaliens de Mezmaiskya (Caucase) que de ceux de Denisova (Altaï) ou de Vindija (Croatie)[8].
Asie
Des études récentes montrent qu'il y a légèrement plus d’ADN néandertalien chez les populations d'Asie de l’est que chez les Européens[9] - [16] - [17] - [18]. Cela signifie que deux événements distincts de transfert de gènes ont eu lieu chez l'homme moderne et que les premiers ancêtres des populations de l’Asie de l'est ont connu plus d'hybridations que les premiers ancêtres des Européens après la divergence des deux groupes[9] - [16] - [17]. Ceci correspondrait à 20,2 % (soit entre 13,4 et 27,1 % avec un intervalle de confiance de 95 %) d'apport supplémentaire néandertalien dans le second transfert génétique des populations d'Asie de l’est[9]. Il est aussi possible, bien que cela soit moins plausible, que la différence soit causée par une dilution de l'apport néandertalien chez les Européens à la suite de migrations tardives d'Afrique[16]. Ces résultats pourraient également résulter d'une sélection négative inférieure en Asie de l’est par rapport à l'Europe[18]. Il a aussi été observé qu'il y avait une variation petite mais significative du taux de mélange au sein des populations européennes, tandis que ces variations ne sont pas significatives dans les populations d'Asie de l’est[9].
Afrique du Nord
Une étude de 2012 a montré que les populations nord-africaines ont un taux d'hybridation situé entre celui des Eurasiens (le plus élevé) et des populations subsahariennes (le plus bas)[19]. Cette étude a également montré une grande variation entre les Nord-Africains eux-mêmes, en fonction principalement de l'importance de l'ascendance eurasienne ou subsaharienne[19]. Cependant, il semblerait que ce résultat n'ait pas pour seule cause l'introgression eurasienne[19].
Afrique subsaharienne
L'apport des Néandertaliens a également été retrouvé, épisodiquement mais significativement, chez les Maasaï, un peuple est-africain[17]. Après avoir identifié les origines africaines et non africaines au sein des Maasaï, il a pu être déduit qu'un apport récent de gènes d'humains modernes non africains (postérieur à l'hybridation avec les Néandertaliens) est à l'origine de cette contribution génétique car environ 30 % du génome des Maasaï a des origines non africaines remontant à environ une centaine de générations[17].
En 2020, la comparaison du génome d'un Néandertalien de l'Altaï avec 2 504 génomes modernes incluant cinq sous-populations africaines montre qu'elles comportent une moyenne de 17 mégabases d'origine néandertalienne, soit 0,3 % de leur génome. Une partie au moins de ces gènes néandertaliens procure une amélioration du système immunitaire et une protection contre le rayonnement ultraviolet, et a dû être retenue sélectivement. La moitié environ de l'ADN néandertalien a sans doute été apportée par des hommes modernes revenus en Afrique au cours des vingt derniers millénaires après une hybridation partielle avec des Néandertaliens ; le reste pourrait ne pas être réellement d'origine néandertalienne mais avoir été acquis par les Néandertaliens de l'Altaï par hybridation avec des hommes modernes au Moyen-Orient il y a plus de 100 000 ans, à l'occasion d'une sortie d'Afrique précoce[20] - [21]. Cependant, cette affirmation selon laquelle l'ascendance néandertalienne se trouve répandue parmi les Africains subsahariens est contestée par certains généticiens, comme David Reich[22].
Variations au sein du génome
L'ADN mitochondrial de l'Homme moderne ne montre pas d'influence néandertalienne[23] - [24]. Les diverses hypothèses envisageables sont que l'ADN mitochondrial néandertalien portait des mutations néfastes qui conduisait à l'extinction du porteur, que les hybrides issus des mères néandertaliennes étaient élevés dans les groupes néandertaliens et s'éteignirent avec eux, ou que les femelles néandertaliennes et les mâles sapiens n'avaient pas de progéniture fertile[23]. L'absence de preuve d'ADN mitochondrial néandertalien chez les humains modernes ne discrédite pas la théorie de l'hybridation. En effet, une étude a produit en 2012 un modèle qui a démontré qu'une croissance exponentielle est compatible avec la survie d'un seul ADN mitochondrial ou une lignée de chromosome Y, si le taux de croissance est dans un intervalle étroit et supercritique. Par conséquent, même si les Néandertaliens et les Africains modernes appartenaient à la même population hybride et même si la population pouvait croître exponentiellement à un taux réduit, le résultat le plus probable serait toujours que tous les humains descendent d'une seule femme (ADN mitochondrial - Ève mitochondriale) et d'un seul homme (chromosome Y - Adam Y-chromosomique). Le modèle estimait qu'il n'y a que 7 % de probabilité pour que les humains puissent avoir soit l'ADN mitochondrial, soit le chromosome Y d'origine néandertalienne[25].
Des études récentes ont montré la présence de grandes régions génomiques avec un apport néandertalien réduit chez l'humain moderne du fait de sélection négative[9] - [18], partiellement causée par l'infertilité des hybrides mâles[18]. Ces grandes régions de faible apport néandertalien sont plus nombreuses sur le chromosome X — avec jusqu'à cinq fois moins d'ascendance néandertalienne par rapport aux autosomes — et contiennent un nombre relativement important de gènes relatifs aux testicules, signifiant que les humains modernes ont relativement peu de gènes néandertaliens sur le chromosome X ou exprimés dans les testicules, confirmant le fait que l'infertilité masculine est affectée par une quantité disproportionnée de gènes sur le chromosome X[18]. De plus, aucune trace de gène issus de chromosomes Y néandertalien n'a été identifiée chez les hommes modernes[26] - [27].
Gènes néandertaliens sélectionnés chez Sapiens
Cette hybridation ancienne a des conséquences sur les populations actuelles : les études génétiques menées dans le laboratoire de Lluis Quintana-Murci à l'institut Pasteur ont ainsi montré des différences entre Africains et Européens, dans leurs réactions immunitaires face à certains stimuli bactériens ou viraux, différences qui ont pu être reliées à des gènes que les seconds ont hérité de Néandertal[28].
Par l'étude des allèles de divers gènes du génome néandertalien hérités par certaines populations sapiens, une autre équipe de chercheurs, dont les travaux furent publiés le 12 février 2016 dans la revue Science, ont pu établir un lien entre cet héritage génétique et diverses caractéristiques notamment sur le plan immunologique, dermatologique et neurologique. Par cette étude croisée des phénotypes et des dossiers médicaux de 28 000 personnes, ces chercheurs ont pu conclure que cet héritage génétique a une incidence sur la santé des personnes concernées[29].
- Risque de complications de la Covid-19
Une séquence génétique de 50 kb localisée sur le chromosome 3 (chr3:45 859 651-45 909 024, hg19) a été identifiée comme associée à un risque accru d'arrêt respiratoire chez les patients souffrant de Covid-19[30]. Une étude estime que cette séquence, qui se retrouve à une fréquence d'environ 30 % en Asie du Sud et d'environ 8 % en Europe, aurait été acquise par contact récent avec des Néandertaliens[31] - [32].
Épigénétique
Une étude de 2014 sur l'épigénétique de l'homme de Néandertal a dévoilé les résultats sur la méthylation complète de l'ADN de Néandertal et de l'homme de Denisova[33]. La reconstruction de la carte de la méthylation de l'ADN a permis aux chercheurs d'évaluer les niveaux d'activité des gènes dans tout le génome de Néandertal et de les comparer aux humains modernes. L'un des principaux résultats a porté sur la morphologie des membres de Néandertal. Gokhman et al. ont constaté que les changements dans les niveaux d'activité du groupe de gènes HOX étaient à l'origine de nombreuses différences morphologiques entre les hommes de Néandertal et les humains modernes, y compris les membres plus courts, les os courbes et plus encore[33].
Lagar Velho
D'après une étude de 1999, les restes de l'enfant de Lagar Velho, retrouvés dans une tombe du paléolithique supérieur au Portugal, présenteraient les traits d'une hybridation entre les Néandertaliens et les humains modernes en Ibérie[34].
Pestera cu Oase
Entre 2013 et 2015, des analyses génétiques effectuées sur les restes d’Homo sapiens trouvés en Roumanie dans la grotte de Peștera cu Oase ont montré la présence d’ADN de Néandertal en proportion élevée. Cela montre qu’il y a eu plusieurs épisodes d'hybridation entre Néandertal et Homo sapiens. Dans le cas de Oase 1, il semble que cette lignée ait disparu sans laisser de descendants. On n'en trouve pas de traces génétiques chez les Européens modernes[35] - [36] - [14].
Riparo Mezzena

Une équipe de chercheurs, dirigée par Silvana Condemi de l’université d’Aix-Marseille, a réinterprété en 2013 des observations faites sur une mandibule mise au jour en 1957 sur le site de Riparo Mezzena, en Italie. En l’examinant, l'équipe a découvert l’ébauche d’un menton, caractéristique présente chez Homo sapiens mais absente chez Néandertal. Or les chercheurs ont pu procéder à une analyse de l’ADN mitochondrial de cette mandibule et ont conclu qu'elle appartenait à un Néandertalien[37]. D’autres mandibules néandertaliennes, trouvées un peu partout en Europe (l’homme de Spy en Belgique, de La Ferrassie en Dordogne, de Las Palomas en Espagne et de la grotte de Vindija en Croatie), présenteraient cette caractéristique[38].
L'hybridation pourrait expliquer d'autres caractéristiques dites "transitionnelles" chez les Néandertaliens trouvés dans la grotte de Vindija : des bourrelets sus-orbitaires moins proéminents, un visage de dimension réduite et des incisives plus étroites[39] - [40].
Les résultats de l'équipe de Silvana Condemi concernant la mandibule de Riparo Mezzena sont toutefois contestés par une autre étude, publiée en 2016 : des analyses génétiques conduisent les auteurs de cette étude à affirmer que cette mandibule n'était pas celle d'un Néandertalien mais d'un Homo sapiens[41].
Dénisoviens
Une étude de 2010 a montré que les Mélanésiens (par exemple les habitants de la Papouasie-Nouvelle-Guinée) partagent davantage d'allèles avec les hommes de Denisova que les Eurasiens et les Africains[42]. De 2,3 à 3,7 % du génome mélanésien provient des hommes de Denisova tandis que presque aucun apport de ces humains n'est présent chez les Eurasiens et Africains[42]. L'étude montre que cet apport ne concerne pas les populations d'Asie de l'est, indiquant qu'il y avait une interaction entre les premiers ancêtres des Mélanésiens et les hommes de Denisova mais que celle-ci n'a pas eu lieu dans les régions proches du sud de la Sibérie, où le premier reste d'un homme de Denisova fut trouvé[42]. De plus, une étude de 2011 a montré que les Aborigènes d'Australie et les hommes de Denisova partageaient davantage d'allèles que les autres populations eurasiennes et africaines, confirmant l'hybridation entre les premiers ancêtres des Mélanésiens et les hommes de Denisova[43].
En 2011, une autre étude a apporté de nouvelles preuves selon lesquelles le plus haut taux d'hybridation avec les hommes de Denisova se retrouve chez les populations d'Océanie, suivi par les populations d'Asie du sud-est, et aucune parmi les populations d'Asie de l'est[44]. Il y a une part importante de matériels génétiques de l'hominidés de Denisova dans les populations d'Océanie et d'Asie du sud-est (Aborigène d'Australie, Polynésiens, Fidjiens, Indonésiens de l'est, Mamanwa et Manobo), mais pas dans certaines populations issue de l'ouest et de la partie continentale de l'Asie du sud-est (Indonésiens de l'ouest, Malaisiens, Andamanais, Asiatiques venant du continent), indiquant que l'hybridation avec les hominidés de Denisova a eu lieu en Asie du sud-est plutôt que sur le continent eurasiatique[45]. L'observation d'une forte hybridation avec les hominidés de Denisova en Océanie et le manque d'hybridation sur l'Asie continentale suggère que les premiers humains modernes et les hominidés de Denisova se sont métissés à l'est de la ligne Wallace[46].
Skoglund et Jakobsson (2011) ont observé que les Océaniens en particulier, suivis par les populations de l'Asie du Sud-Est, bénéficient d'une grande admixtion de gènes des hommes de Denisova par rapport aux autres populations[47]. De plus, les chercheurs ont trouvé des traces possibles d'admixtion Dénisovienne chez les peuples de l'Asie de l'Est, mais aucune admixtion de ces gènes chez les Amérindiens[47]. Par opposition, Prüfer et d'autres chercheurs (2013) ont trouvé que les peuples d'Asie centrale et les Amérindiens ont 0.2% de contribution génétique Dénisovienne, soit 25 fois inférieure à celle des Océaniens[48]. Les moyens de mouvement de ces gènes vers ces populations restent cependant inconnus[48]. Toutefois, Wall (2013) a énoncé qu'il ne trouvait aucune preuve d'admixtion Dénisovienne chez les peuples de l'Asie de l'Est[49]. Des trouvailles indiquent que le déplacement des gènes Dénisoviens a eu lieu entre les divers ancêtres communs des Phillipins aborigènes, Australiens aborigènes, et des peuples de Nouvelle-Guinée[44] - [50]. Ces derniers et les Australiens ont des taux d'admixtion de Dénisoviens similaires, indiquant qu'une hybridation a dû avoir lieu entre eux avant leur arrivée en Nouvelle-Guinée et en Australie pléistocène, il y a au moins 44 000 ans[44]. Il a aussi été observé que la proportion d'ascendance Océanienne chez les peuples de l'Asie du Sud-Est est proportionnelle à leur admixtion Dénisovienne, sauf dans les Philippines, où il y a une proportion plus haute d'admixtion de Dénisoviens[44]. Reich et d'autres chercheurs (2011) ont suggéré une modélisation possible dans laquelle il y aurait une importante vague de migration de l'Homme moderne (Homo Sapiens Sapiens) se dirigeant vers l'Est, dont certains étaient des ancêtres communs Philippins, de Nouvelle-Guinée ou d'Australie qui se sont hybridés avec des Dénisoviens, respectivement suivis par une divergence des ancêtres Philippins, s'hybridant ainsi avec les peuples et ancêtres de Nouvelle-Guinée et Australiens, avec une part de cette même population de la migration qui n'est pas entrée en contact avec des gènes de Dénisoviens, et l'hybridation entre les ancêtres Philippins avec une part de la population d'un mouvement de migration vers l'Est beaucoup plus tardif (l'autre partie des migrants deviendront les Asiatiques de l'Est)[44]. Il a été démontré que les Eurasiens possédaient du matériel génétique dérivé archaïque, mais en beaucoup plus basse quantité, qui chevauchait sur le matériel génétique Dénisovien, découlant du fait que les hominidés de Dénisova sont liés génétiquement aux Néandertaliens, qui ont eux-mêmes contribué au partage de gènes eurasien, au lieu de l'hybridation directe des Dénisoviens avec les ancêtres lointains de ces Eurasiens[51] - [52]. On observe de suite les restes d'un squelette de l'Homme de Tianuyan près de Zhoukoudian, en Chine. Ses 40,000 ans de PB ont démontré une contribution génétique Néandertalienne chez les humains modernes d'Eurasie, mais ne contenant aucune contribution de Dénisovien perceptible[53]. L'Homme de Tianuyan est un cousin lointain des ancêtres de plusieurs populations d'Asie et d'Amérique (Amérindiens), mais le squelette était daté à une époque ultérieure à la divergence entre les peuples d'Asie et d'Europe[53]. Le manque de composantes et d'élément génétiques des Dénisoviens chez l'Homme de Tianyuan suggère que la contribution génétique a seulement eu lieu au centre des continents[48].
En explorant les allèles HLA du système immunitaire de l'Homme de Tianuyan, la supposition que ces allèles HLA-B*73 se sont introgressés des Dénisoviens vers les Hommes modernes en Asie de l'Ouest, à cause d'un schéma de distribution et d'une divergence de l'allèle HLA-B*73 des autres allèles HLA s'est posée[54]. Chez les Hommes modernes, l'allèle HLA-B*73 est concentré en Asie de l'Ouest, mais reste rare ou absent autre part[54]. Même si HLA-B*73 n'est pas présent dans le génome Dénisovien, une étude a montré que c'était associé avec la présence de HLA-C*15:05 dérivé des Dénisoviens, explication consistente avec les statistiques montrant que 98% des Humains modernes possèdent des allèles B*73 et des allèles C*15:05[54]. Les deux allotypes HLA-A (A*02 and A*11) et les deux HLA-C (C*15 and C*12:02) Dénisoviens correspondent aux allèles communs chez l'Homme moderne, tandis qu'un des allotypes HLA-B Dénisoviens correspond à un allèle recombinant rare absent chez la majorité des humains[54]. Une contribution de ces allèles des Dénisoviens vers l'Homme moderne est envisagée, car il est peu probable qu'ils aient été préservés indépendamment pour une telle durée à cause du haut taux de mutation des allèles HLA[54].
Gènes dénisoviens sélectionnés chez Sapiens
Il a été trouvé que la variante du gène EPAS1 a été transmise des Dénisoviens vers l'Homme moderne[55]. Les variantes ancestrales régulent les niveaux d'hémoglobine pour compenser les niveux bas d'oxygène dans les hautes altitudes. Il existe cependant une mauvaise adaptation qui augmenterait la viscosité sanguine[55]. Sa variante que les Dénisoviens possédaient limite la hausse du taux d'hémoglobine, résultant ainsi dans une adaptation facilitée pour les hautes altitudes de l'Homme de Denisova[55]. La variante Dénisovienne du gène EPAS1 est répandue chez les Tibétains majoritairement, étant sélectionnée chez leurs ancêtres après la colonisation du plateau tibétain[55].
Une étude comparative du génome chez les Inuits du Groenland, à l'aide de données issues de puces de polymorphisme nucléotidique, et celui de l'Homme de Denisova a mis en évidence un phénomène d'introgression adaptative. La région du chromosome 1 où se trouvent situés les gènes WARS2 et TBX15 montre une variante divergente chez les Inuits du Groenland qui est étroitement liée à la séquence correspondante dans le génome de l'Homme de Denisova. Ces gènes sont associés à la différenciation du tissu adipeux et à la distribution de graisse corporelle chez les humains. Cette variante a été sélectionnée dans une région géographique beaucoup plus vaste que le Groenland et est associée à des changements dans l'expression de WARS2 et TBX15 dans de multiples tissus, y compris la glande surrénale et le tissu adipeux sous-cutané[56].
Autres espèces humaines archaïques
La dégradation rapide de l'ADN sous un climat tropical rend actuellement impossible l'extraction et le séquençage de l'ADN fossile sur des spécimens d'Afrique ou d'Asie tropicale de plus 15 000 ans[57].
En 2011, après avoir découvert trois régions candidates à l'introgression en recherchant des modèles inhabituels de variations – indiquant une origine différente – dans 61 régions non codantes dans deux populations de chasseurs-cueilleurs (Aka et San, qui ont présenté un fort taux d'hybridation dans les données) et un groupe d'agriculteurs ouest-africains (les Mandingues, qui n'ont présenté aucun taux d'hybridation significatif), les chercheurs ont conclu qu'environ 2 % du matériel génétique des populations subsahariennes fut introduit dans le génome humain il y a environ 35 000 ans, par des humains archaïques qui se sont séparés de la lignées d'Homo sapiens il y a 700 000 ans[58]. Après une recherche des haplotypes introgressifs dans les populations subsahariennes, il a été suggéré que cette hybridation aurait eu lieu avec des humains archaïques qui habitaient potentiellement le centre de l'Afrique[58].
En 2012, des chercheurs ont étudié la séquence génétique de quinze chasseurs-cueilleurs subsahariens issus de trois groupes – cinq pygmées (trois Baka, un Bedzan et un Bakola) du Cameroun, cinq Hadza de Tanzanie et cinq Sandawe de Tanzanie – et ont découvert des signes selon lesquels les ancêtres des chasseurs-cueilleurs se sont hybridés avec une ou plusieurs populations humaines archaïques[57] il y a probablement 40 000 ans[59]. Ils ont aussi découvert que le dernier ancêtre commun de ces 15 individus avec l'haplotype introgressif putatif remontait à 1,2-1,3 million d'années[57].
Haplogroupe A00
Un haplogroupe archaïque (ou d'anciens Homo sapiens) du chromosome Y, appelé A00, a été identifié chez les Mbo et les Bangwa (Cameroun) et leurs descendants (dont l’afro-américain Albert Perry[60]). La date de divergence de cet haplogroupe A00 avec les autres haplogroupes humains modernes est évaluée entre 254 000[61] et 275 000 ans[26]. Sa survivance dans le patrimoine génétique actuel résulterait d'une hybridation avec des humains africains peut-être antérieurs aux humains modernes (mais contemporains de l'Homo sapiens ancien de Djebel Irhoud)[62] - [63] - [64].
Introgressions de Sapiens vers Néandertal
On trouve aussi des gènes sapiens chez l'homme de Néandertal[alpha 1] - [65] - [66]. Il s'ensuit qu'on devrait pouvoir retrouver des caractéristiques de Sapiens chez des Néandertaliens.
Une étude publiée en mars 2018 portant sur le séquençage de cinq nouveaux génomes de Néandertaliens (quatre individus d'une époque tardive localisés en Europe occidentale - un en France, deux en Belgique et un en Croatie - et un plus ancien dont les ossements furent trouvés dans le Caucase russe) pose de nouvelles questions. Contrairement à ce que l'on pourrait attendre d'un phénomène d'hybridation, l'étude révèle n'avoir trouvé aucun ADN nucléaire d'Homo sapiens dans le génome de ces Néandertaliens[15].
Mais comment expliquer dans ce cas les caractéristiques transitionnelles découvertes par l'équipe de Silvana Condemi chez les Néandertaliens de traits Homo sapiens sans apport apparent de gènes de ceux-ci dans le génome de Néandertal ? Selon elle, ces faits peuvent s'expliquer si deux hypothèses sont validées : 1° que les flux géniques se sont surtout produits dans le sens Néandertaliens → Homo sapiens, 2° que les principaux évènements d'hybridation ont eu lieu hors d'Europe bien avant l'arrivée de nos ancêtres sur ce continent[67].
L'ADN mitochondrial (ADNmt) et les séquences autosomiques des Néandertaliens, des Dénisoviens et des humains modernes induisent des phylogénies en apparence contradictoires. Les génomes autosomiques montrent que les Néandertaliens et les Dénisoviens sont des groupes frères qui se sont séparés des humains modernes il y a 765 à 550 milliers d'années (ka)[68]. En revanche, les ADNmt des Néandertaliens et des humains modernes sont plus proches les uns des autres que de ceux des Dénisoviens, et ne se sont séparés qu'il y a 468 à 360 ka[69]. A contrario, les Néandertaliens archaïques de Sima de los Huesos, âgés d'environ 400 ka, avaient des ADNmt semblables à ceux des Dénisoviens[70] - [71]. Ces résultats suggèrent que les Néandertaliens portaient à l'origine un ADNmt de type dénisovien, qui a ensuite été complètement remplacé via un flux génique provenant d'une lignée ancienne étroitement apparentée aux humains modernes[69] - [71].
Les hasards de la conservation des ADN anciens ont fait qu'à une exception près (une fraction de seulement 118 kb d'un chromosome Y néandertalien[72]), tous les séquençages d'ADN néandertaliens et dénisoviens provenaient d'individus de sexe féminin. En 2020, une technique de concentration ciblant 6,9 Mb du chromosome Y humain a permis de séquencer les chromosomes Y de trois Néandertaliens (El Sidrón 1253, daté à ~53–46 ka ; Mezmaiskaya 2, 45–43 ka ; Spy 94a, 39–38 ka) et deux Dénisoviens (Denisova 8, 136–106 ka ; Denisova 4, 84–55 ka)[73]. Les résultats sont quasi identiques à ceux obtenus par l'ADN mitochondrial : un remplacement complet du chromosome Y ancestral des Néandertaliens par le chromosome Y des humains modernes, et aucun remplacement chez les Dénisoviens. Ce remplacement, effectué il y a entre 350 et 150 ka, est sans doute dû à des avantages sélectifs. En tout cas, le fait que les hommes et les femmes modernes ont participé à ce flux génique suggère qu'aussi bien les populations sapiens que neandertalensis ont accepté les enfants d'origine mixte[74] - [73].
Super-archaïques
Une étude génétique parue en février 2020, ayant analysé l'ADN fossile néandertalien (issu de spécimens de l'Altaï et de la grotte de Vindija, en Croatie) et l'ADN dénisovien (issu de la grotte de Denisova), en tire la conclusion que l'ancêtre commun de ces deux branches, appelé Néandersovien, aurait quitté l'Afrique il y a environ 750 000 ans et se serait peu après hybridé en Eurasie avec une population locale qualifiée de super-archaïque. Celle-ci serait sortie d'Afrique il y a environ 2 millions d'années, et serait donc très éloignée des Néandersoviens, ce qui n'aurait pas fait barrage à l'hybridation[75] - [76].
Les Néandersoviens se seraient subdivisés entre Eurasiens de l'Ouest, ou Néandertaliens, et Eurasiens de l'Est, ou Dénisoviens, il y a environ 700 000 ans, c'est-à-dire beaucoup plus tôt que les estimations des études précédentes. Il y aurait eu par la suite une deuxième hybridation des super-archaïques avec les seuls Dénisoviens en Asie orientale[75].
Notes et références
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Archaic human admixture with modern humans » (voir la liste des auteurs).
Notes
- Les dates extrêmes déterminées par l'étude vont de 447 000 à 806 000 ans avec une probabilité estimée à 95 % pour 588 000 ans, la date la plus probable selon cette étude.
Références
- Stringer 2012
- (en) Rebecca Rogers Ackermann, Alex Mackay et Michael L. Arnold, « The Hybrid Origin of “Modern” Humans », Evolutionary Biology, vol. 43, no 1, , p. 1–11 (ISSN 1934-2845, DOI 10.1007/s11692-015-9348-1, lire en ligne, consulté le )
- Jordana Cepelewicz, « Fossil DNA Reveals New Twists in Modern Human Origins », sur Quanta Magazine (consulté le )
- (en) T. Huxley, Collected Essays: Volume VII, Man's Place in Nature, (lire en ligne), « The Aryan Question and Pre-Historic Man »
- (en) H.P. Steensby, « Racestudier i Danmark », Geografisk Tidsskrift, vol. 9, , p. 135–145 (lire en ligne)
- (en) C.S. Coon, The Origin of Races, , p. 529
- (en) R.E. Green, Krause, J., Briggs, A.W., Maricic, T., Stenzel, U., Kircher, M. et al., « A Draft Sequence of the Neandertal Genome », Science, vol. 328, no 5979, , p. 710–722 (PMID 20448178, DOI 10.1126/science.1188021)
- (en) K. Prüfer, Racimo, F., Patterson, N., Jay, F., Sankararaman, S., Sawyer, S. et al., « The complete genome sequence of a Neanderthal from the Altai Mountains », Nature, vol. 505, no 7481, , p. 43–49 (DOI 10.1038/nature12886, Bibcode 2014Natur.505...43P)
- (en) B. Vernot et Akey, J. M., « Resurrecting Surviving Neandertal Lineages from Modern Human Genomes », Science, vol. 343, no 6174, , p. 1017–1021 (DOI 10.1126/science.1245938)
- « Here's What Happened When Neanderthals And Ancient Humans Hooked Up 80,000 Years Ago » (consulté le )
- (en) Ann Gibbons, « Neandertals and Moderns Made Imperfect Mates », Science, vol. 343, , p. 471-472 (ISSN 0036-8075 et 1095-9203, PMID 24482455, DOI 10.1126/science.343.6170.471, lire en ligne, consulté le )
- (en) M.A. Yang, Malaspinas, A.S., Durand, E.Y. et Slatkin, M., « Ancient Structure in Africa Unlikely to Explain Neanderthal and Non-African Genetic Similarity », Molecular Biology and Evolution, vol. 29, no 10, , p. 2987–2995 (DOI 10.1093/molbev/mss117)
- (en) S. Sankararaman, Patterson, N., Li, H., Pääbo, S., Reich, D et Akey, J.M., « The Date of Interbreeding between Neandertals and Modern Humans », PLoS Genetics, vol. 8, no 10, , e1002947 (PMID 23055938, PMCID 3464203, DOI 10.1371/journal.pgen.1002947)
- « Erbgut des bisher ältesten modernen Menschen entschlüsselt », sur www.mpg.de (consulté le )
- (en) Mateja Hajdinjak, Qiaomei Fu, Alexander Hübner, Martin Petr, Fabrizio Mafessoni, Steffi Grote, Pontus Skoglund, Vagheesh Narasimham, Hélène Rougier, Isabelle Crevecoeur, Patrick Semal, Marie Soressi, Sahra Talamo, Jean-Jacques Hublin, Ivan Gu\u0161i\u0107, \u017deljko Ku\u0107an, Pavao Rudan, Liubov V. Golovanova, Vladimir B. Doronichev, Cosimo Posth, Johannes Krause, Petra Korlevi\u0107, Sarah Nagel, Birgit Nickel, Montgomery Slatkin, Nick Patterson, David Reich, Kay Prüfer, Matthias Meyer, Svante Pääbo et Janet Kelso, « Reconstructing the genetic history of late Neanderthals », Nature, Springer Nature, vol. 555, no 7698, , p. 652-656 (ISSN 0028-0836, DOI 10.1038/nature26151, lire en ligne)
- (en) M. Meyer, Kircher, M., Gansauge, M.T., Li, H., Racimo, F., Mallick, S. et al., « A High-Coverage Genome Sequence from an Archaic Denisovan Individual », Science, vol. 338, no 6104, , p. 222–226 (PMID 22936568, PMCID 3617501, DOI 10.1126/science.1224344)
- (en) J.D. Wall, Yang, M.A., Jay, F., Kim, S.K., Durand, E.Y., Stevison, L.S. et al., « Higher Levels of Neanderthal Ancestry in East Asians than in Europeans », Genetics, vol. 194, no 1, , p. 199–209 (DOI 10.1534/genetics.112.148213)
- (en) S. Sankararaman, Mallick, S., Dannemann, M., Prüfer, K., Kelso, J., Pääbo, S. et al., « The genomic landscape of Neanderthal ancestry in present-day humans », Nature, vol. 507, no 7492, , p. 354–357 (DOI 10.1038/nature12961)
- (en) F. Sánchez-Quinto, Botigué, L.R., Civit, S., Arenas, C., Ávila-Arcos, M.C., Bustamante, C.D. et al., « North African Populations Carry the Signature of Admixture with Neandertals », PLoS ONE, vol. 7, no 10, , e47765 (PMID 23082212, PMCID 3474783, DOI 10.1371/journal.pone.0047765)
- (en) Michael Price, « Africans, too, carry Neanderthal genetic legacy », Science, vol. 367, no 6477, , p. 497 (DOI 10.1126/science.367.6477.497).
- (en) Lu Chen, Aaron B. Wolf, Wenqing Fu, Liming Li et Joshua M. Akey, « Identifying and Interpreting Apparent Neanderthal Ancestry in African Individuals », Cell, (DOI 10.1016/j.cell.2020.01.012).
- Carl Zimmer, « Neanderthal Genes Hint at Much Earlier Human Migration From Africa », The New York Times, (lire en ligne, consulté le )
- (en) Mason, P.H. et Short, R.V., « Neanderthal-human Hybrids », Hypothesis, vol. 9, no 1, , e1 (DOI 10.5779/hypothesis.v9i1.215, lire en ligne)
- (en) C.C. Wang, Farina, S.E. et Li, H., « Neanderthal DNA and modern human origins », Quaternary International, vol. 295, , p. 126–129 (DOI 10.1016/j.quaint.2012.02.027)
- (en) Armando Neves et Maurizio Serva, « Extremely Rare Interbreeding Events Can Explain Neanderthal DNA in Living Humans », PLoS ONE,
- Fernando L. Mendez et al., The Divergence of Neandertal and Modern Human Y Chromosomes, 2016. doi: 10.1016/j.ajhg.2016.02.023
- Génétique: l'homme de Néandertal et la femme moderne étaient incompatibles
- Quach 2016
- « The phenotypic legacy of admixture between modern humans and Neandertals, Science, Corinne N. Simonti et al., 12 février 2016 »
- (en) D. Ellinghaus et al., « Genome-wide Association Study of Severe Covid-19 with Respiratory Failure », New England Journal of Medicine, (DOI 10.1056/NEJMoa2020283).
- (en) Hugo Zeberg et Svante Pääbo, « The major genetic risk factor for severe COVID-19 is inherited from Neandertals », sur biorxiv.org, (DOI 10.1101/2020.07.03.186296)
- (en) Hugo Zeberg et Svante Pääbo, « The major genetic risk factor for severe COVID-19 is inherited from Neanderthals », Nature, , p. 1–6 (ISSN 1476-4687, DOI 10.1038/s41586-020-2818-3, lire en ligne, consulté le )
- Gokhman D, Lavi E, Prüfer K, Fraga MF, Riancho JA, Kelso J, Pääbo S, Meshorer E, Carmel L, « Reconstructing the DNA methylation maps of the Neandertal and the Denisovan », Science, vol. 344, no 6183, , p. 523–27 (PMID 24786081, DOI 10.1126/science.1250368, Bibcode 2014Sci...344..523G)
- (en) Duarte, C., Maurício, J., Pettitt, P.B., Souto, P., Trinkaus, E., Plicht, H. van der et Zilhão, J., « The early Upper Paleolithic human skeleton from the Abrigo do Lagar Velho (Portugal) and modern-human emergence in Iberia », Proceedings of the National Academy of Sciences, vol. 96, no 13, , p. 7604–9 (PMID 10377462, PMCID 22133, DOI 10.1073/pnas.96.13.7604)
- « Early European may have had Neanderthal great-great-grandparent » (consulté le )
- « Frühe Europäer haben sich mit Neandertalern vermischt », sur www.mpg.de (consulté le )
- (en) Silvana Condemi, Aurélien Mounier, Paolo Giunti et Martina Lari, « Possible Interbreeding in Late Italian Neanderthals? New Data from the Mezzena Jaw (Monti Lessini, Verona, Italy) », PLOS ONE, vol. 8, no 3, (ISSN 1932-6203, PMID 23544098, PMCID PMC3609795, DOI 10.1371/journal.pone.0059781)
- « Le menton de Néandertal : preuve d'un métissage avec l'homme moderne ? », sur le bob, l'extra-média, Cité des sciences et de l'industrie, Palais de la découverte,
- (en) Milford H. Wolpoff, Fred H. Smith, Mirko Malez et Jakov Radovčić, « Upper pleistocene human remains from Vindija cave, Croatia, Yugoslavia », American Journal of Physical Anthropology, vol. 54, , p. 499–545 (ISSN 1096-8644, DOI 10.1002/ajpa.1330540407, lire en ligne, consulté le )
- (en) Erik Trinkaus et Fred H. Smith, « Body size of the Vindija Neandertals », Journal of Human Evolution, vol. 28, , p. 201–208 (DOI 10.1006/jhev.1995.1015, lire en ligne, consulté le )
- (en) Sahra Talamo, Mateja Hajdinjak, Marcello A. Mannino, Leone Fasani, Frido Welker, Fabio Martini, Francesca Romagnoli, Roberto Zorzin, Matthias Meyer et Jean-Jacques Hublin, « Direct radiocarbon dating and genetic analyses on the purported Neanderthal mandible from the Monti Lessini (Italy) », Scientific Reports, Springer Science and Business Media LLC, vol. 6, no 1, (ISSN 2045-2322, DOI 10.1038/srep29144, lire en ligne).
- (en) D. Reich, Green, R.E., Kircher, M., Krause, J., Patterson, N., Durand, E.Y. et al., « Genetic history of an archaic hominin group from Denisova Cave in Siberia », Nature, vol. 468, no 7327, , p. 1053–1060 (PMID 21179161, DOI 10.1038/nature09710)
- (en) M. Rasmussen, Guo, X., Wang, Y., Lohmueller, K.E., Rasmussen, S., Albrechtsen, A. et al., « An Aboriginal Australian Genome Reveals Separate Human Dispersals into Asia », Science, vol. 334, no 6052, , p. 94–98 (DOI 10.1126/science.1211177)
- D. Reich, Patterson, N., Kircher, M., Delfin, F., Nandineni, M.R., Pugach, I. et al., « Denisova Admixture and the First Modern Human Dispersals into Southeast Asia and Oceania », The American Journal of Human Genetics, vol. 89, no 4, , p. 516–28 (PMID 21944045, PMCID 3188841, DOI 10.1016/j.ajhg.2011.09.005)
- (en) D. Reich, Patterson, N., Kircher, M., Delfin, F., Nandineni, M.R., Pugach, I. et al., « Denisova Admixture and the First Modern Human Dispersals into Southeast Asia and Oceania », The American Journal of Human Genetics, vol. 89, no 4, , p. 516–528 (PMID 21944045, PMCID 3188841, DOI 10.1016/j.ajhg.2011.09.005)
- (en) A. Cooper et Stringer, C.B., « Did the Denisovans Cross Wallace's Line? », Science, vol. 342, no 6156, , p. 321–323 (PMID 24136958, DOI 10.1126/science.1244869)
- (en) P. Skoglund et Jakobsson, M., « Archaic human ancestry in East Asia », Proceedings of the National Academy of Sciences, vol. 108, no 45, , p. 18301–06 (DOI 10.1073/pnas.1108181108, Bibcode 2011PNAS..10818301S)
- K. Prüfer, Racimo, F., Patterson, N., Jay, F., Sankararaman, S., Sawyer, S. et al., « The complete genome sequence of a Neanderthal from the Altai Mountains », Nature, vol. 505, no 7481, , p. 43–49 (PMID 24352235, PMCID 4031459, DOI 10.1038/nature12886, Bibcode 2014Natur.505...43P)
- J.D. Wall, Yang, M.A., Jay, F., Kim, S.K., Durand, E.Y., Stevison, L.S. et al., « Higher Levels of Neanderthal Ancestry in East Asians than in Europeans », Genetics, vol. 194, no 1, , p. 199–209 (PMCID 3632468, DOI 10.1534/genetics.112.148213)
- (en) I. Flatow et Reich, D., « Meet Your Ancient Relatives: The Denisovans », NPR,
- M. Meyer, Kircher, M., Gansauge, M.T., Li, H., Racimo, F., Mallick, S. et al., « A High-Coverage Genome Sequence from an Archaic Denisovan Individual », Science, vol. 338, no 6104, , p. 222–26 (PMID 22936568, PMCID 3617501, DOI 10.1126/science.1224344, Bibcode 2012Sci...338..222M)
- D. Reich, Green, R.E., Kircher, M., Krause, J., Patterson, N., Durand, E.Y. et al., « Genetic history of an archaic hominin group from Denisova Cave in Siberia », Nature, vol. 468, no 7327, , p. 1053–60 (PMID 21179161, PMCID 4306417, DOI 10.1038/nature09710, Bibcode 2010Natur.468.1053R)
- (en) Q. Fu, Meyer, M., Gao, X., Stenzel, U., Burbano, H.A., Kelso, J. et Paabo, S., « DNA analysis of an early modern human from Tianyuan Cave, China », Proceedings of the National Academy of Sciences, vol. 110, no 6, , p. 2223–27 (PMID 23341637, PMCID 3568306, DOI 10.1073/pnas.1221359110, Bibcode 2013PNAS..110.2223F)
- L. Abi-Rached, M. J. Jobin, S. Kulkarni, A. McWhinnie, K. Dalva, L. Gragert et al., « The Shaping of Modern Human Immune Systems by Multiregional Admixture with Archaic Humans », Science, vol. 334, no 6052, , p. 89–94 (PMID 21868630, PMCID 3677943, DOI 10.1126/science.1209202, Bibcode 2011Sci...334...89A)
- E. Huerta-Sánchez, X. Jin, Asan, Z. Bianba, B.M. Peter, N. Vinckenbosch et al., « Altitude adaptation in Tibetans caused by introgression of Denisovan-like DNA », Nature, vol. 512, , p. 194–97 (PMID 25043035, PMCID 4134395, DOI 10.1038/nature13408, Bibcode 2014Natur.512..194H)
- Fernando Racimo, David Gokhman, Matteo Fumagalli, Amy Ko, Torben Hansen, Ida Moltke, Anders Albrechtsen, Liran Carmel, Emilia Huerta-Sánchez et Rasmus Nielsen, « Archaic adaptive introgression in TBX15/WARS2 », Molecular Biology and Evolution, Oxford University Press (OUP), , msw283 (ISSN 0737-4038, DOI 10.1093/molbev/msw283, lire en ligne)
- (en) J. Lachance, Vernot, B., Elbers, C.C., Ferwerda, B., Froment, A., Bodo, J.M. et al., « Evolutionary History and Adaptation from High-Coverage Whole-Genome Sequences of Diverse African Hunter-Gatherers », Cell, vol. 150, no 3, , p. 457–469 (DOI 10.1016/j.cell.2012.07.009)
- (en) M.F. Hammer, Woerner, A.E., Mendez, F.L., Watkins, J.C. et Wall, J.D., « Genetic evidence for archaic admixture in Africa », Proceedings of the National Academy of Sciences, vol. 108, no 37, , p. 15123–15128 (PMID 21896735, PMCID 3174671, DOI 10.1073/pnas.1109300108)
- (en) E. Callaway, « Hunter-gatherer genomes a trove of genetic diversity », Nature, (DOI 10.1038/nature.2012.11076)
- L’homme qui ne descendait pas d’Adam
- Un effet de goulot récent dans la diversité du chromosome Y coïncide avec un changement global dans la culture
- Schlebusch et al., « Southern African ancient genomes estimate modern human divergence to 350,000 to 260,000 years ago », Science, vol. 358, no 6363, , p. 652–655 (PMID 28971970, DOI 10.1126/science.aao6266, Bibcode 2017Sci...358..652S)
- Elhaik E, Tatarinova TV, Klyosov AA, Graur D, « The 'extremely ancient' chromosome that isn't: a forensic bioinformatic investigation of Albert Perry's X-degenerate portion of the Y chromosome », European Journal of Human Genetics, vol. 22, no 9, , p. 1111–16 (PMID 24448544, PMCID 4135414, DOI 10.1038/ejhg.2013.303)
- Karmin et al., « A recent bottleneck of Y chromosome diversity coincides with a global change in culture », Genome Research, vol. 25, no 4, , p. 459–66 (PMID 25770088, PMCID 4381518, DOI 10.1101/gr.186684.114) "we date the Y-chromosomal most recent common ancestor (MRCA) in Africa at 254 (95% CI 192–307) kya and detect a cluster of major non-African founder haplogroups in a narrow time interval at 47–52 kya, consistent with a rapid initial colonization model of Eurasia and Oceania after the out-of-Africa bottleneck. In contrast to demographic reconstructions based on mtDNA, we infer a second strong bottleneck in Y-chromosome lineages dating to the last 10 ky. We hypothesize that this bottleneck is caused by cultural changes affecting variance of reproductive success among males."
- (en) Martin Kuhlwilm, Ilan Gronau, Melissa J. Hubisz et Cesare de Filippo, « Ancient gene flow from early modern humans into Eastern Neanderthals », Nature, vol. 530, no 7591, , p. 429–433 (ISSN 0028-0836 et 1476-4687, DOI 10.1038/nature16544, lire en ligne, consulté le )
- Martin Kuhlwilm, Ilan Gronau, Melissa J. Hubisz et Cesare de Filippo, « Ancient gene flow from early modern humans into Eastern Neanderthals », Nature, vol. 530, no 7591, , p. 429–433 (ISSN 0028-0836, PMID 26886800, PMCID PMC4933530, DOI 10.1038/nature16544, lire en ligne, consulté le )
- Silvana Condemi, « Les gènes des derniers Néandertaliens révélés », sur Pourlascience.fr, (consulté le )
- (en) K. Prüfer, F. Racimo, N. Patterson, F. Jay, S. Sankararaman et al., « The complete genome sequence of a Neanderthal from the Altai Mountains », Nature, vol. 505, , p. 43-49 (DOI 10.1038/nature12886).
- (en) C. Posth, C. Wißing, K. Kitagawa, L. Pagani, L. van Holstein et al., « Deeply divergent archaic mitochondrial genome provides lower time boundary for African gene flow into Neanderthals », Nature Communications, vol. 8, , article no 16046 (DOI 10.1038/ncomms16046).
- (en) M. Meyer, Q. Fu, A. Aximu-Petri, I. Glocke, B. Nickel et al., « A mitochondrial genome sequence of a hominin from Sima de los Huesos », Nature, vol. 505, , p. 403-406 (DOI 10.1038/nature12788).
- (en) M. Meyer, J.-L. Arsuaga, C. de Filippo, S. Nagel, A. Aximu-Petri et al., « Nuclear DNA sequences from the Middle Pleistocene Sima de los Huesos hominins », Nature, vol. 531, , p. 504-507 (DOI 10.1038/nature17405).
- (en) F. L. Mendez, G. D. Poznik, S. Castellano et C. D. Bustamante, « The divergence of Neandertal and modern human Y chromosomes », American Journal of Human Genetics, vol. 98, , p. 728–734 (DOI 10.1016/j.ajhg.2016.02.023).
- (en) Martin Petr, Mateja Hajdinjak, Qiaomei Fu, Elena Essel, Hélène Rougier et al., « The evolutionary history of Neanderthal and Denisovan Y chromosomes », Science, vol. 369, no 6511, , p. 1653-1656 (DOI 10.1126/science.abb6460).
- (en) Mikkel Heide Schierup, « The last pieces of a puzzling early meeting », Science, vol. 369, no 6511, , p. 1565-1566 (DOI 10.1126/science.abe2766).
- (en) Alan R. Rogers, Nathan S. Harris et Alan A. Achenbach, « Neanderthal-Denisovan ancestors interbred with a distantly related hominin », Science Advances, vol. 6, no 8, (DOI 10.1126/sciadv.aay5483, lire en ligne)
- « Les super-archaïques, une population fantôme d'hominidés il y a 700 000 ans », Hominidés.com, (lire en ligne)
Voir aussi
Bibliographie
- (en) Christopher Brian Stringer, « What makes a modern human », Nature, vol. 485, no 7396, , p. 33–35 (PMID 22552077, DOI 10.1038/485033a)
- (en) Hélène Quach et al., « Genetic Adaptation and Neandertal Admixture Shaped the Immune System of Human Populations », Cell, vol. 167, no 3, , p. 643–656 (ISSN 0092-8674, PMID 27768888, DOI 10.1016/j.cell.2016.09.024, lire en ligne, consulté le )
- (en) Seán Lea, The genetic legacy of archaic hominin species, University College London, Division of Biosciences, 81 p. en ligne