Donnée biomédicale ouverte
Les données biomédicales ouvertes révèlent que la médecine et l’open data sont deux domaines qui entretiennent des relations nouvelles, complexes, en pleine évolution et parfois apparemment contradictoires. En effet, introduire l'open data dans le monde médical, vise à répondre à deux enjeux parfois a priori opposés :
- permettre une large ouverture des données de santé ;
- le faire en respectant l'éthique biomédicale et le droit de la santé, le secret de fabrication, qui sont très contraignants notamment en termes de protection de la confidentialité de certaines données, en particulier les données personnelles, surtout quand elle concerne la santé (Les données de santé sont dans ce cadre les « données susceptibles de révéler la maladie d’une personne »[1] - [2]) et du secret médical, ce qui suppose un système fiable d’anonymisation des données, assez sophistiqué pour éviter les doubles comptages et d’autres biais lors de l’utilisation de ces données[2]. Un projet de règlement européen retient une définition plus large des données de santé : « toute information relative à la santé physique ou mentale d'une personne, ou à la prestation de services de santé à cette personne »[1].
Avec l’émergence du smartphone, des tablettes (utilisés par plus de 3 milliards de personnes) et d’autres objets connectés, ainsi que d’applications médicales (ex. : surveillance du poids, de l’activité physique et sportive, de la qualité du sommeil, etc.) et d’une informatique de plus en plus ubiquitaire (17 millions de bracelets et de montres intelligentes auraient été vendus en 2014), des enjeux nouveaux apparaissent, notamment concernant la confidentialité, le stockage et la protection des données médicales personnelles au sein du et « big data ». Se pose aussi la question de leur utilisation par des tiers ou par le secteur privé (Apple, dont l'iOS 8 peut pour la première fois gérer des données personnelles de santé) a ainsi interdit la commercialisation des données de santé HealthKit[3]).
Atouts et enjeux
Dans le monde entier, l’échange et le partage dématérialisés de données de santé augmentent rapidement. Les socioéconomistes du domaine de la santé admettent qu’en complément des progrès de la prévention et en termes de dossier médical (Dossier médical personnel et carte vitale), des technologies intelligentes pourraient aider à résoudre deux grands problèmes en matière de soins de santé : les dépenses excessives dans le monde riche, et le manque d’argent des pays pauvres[4] et qu’une grande partie des problèmes de santé publique ou individuelle sont exacerbés par la pauvreté, et parfois y trouvent leur unique origine.
Or, des solutions open-source, libres et collaboratives ont prouvé qu’elles peuvent souvent permettre des sauts technologiques ou méthodologiques aboutissant à des résultats plus rapides, moins couteux et souvent plus adaptés aux besoins des utilisateurs (patients, médecins, étudiants en santé, etc. dans le cas présent). L'OCDE encourage ses membres à développer l'Open data dont pour les données de santé considérées comme une ressource qu'il est « économiquement et éthiquement logique d'utiliser autant que possible »[5].
Intérêts de l'open data pour la médecine et la santé publique
Selon la Commission open data en santé française, les enjeux sont notamment la gouvernance et la « démocratie sanitaire, l'autonomisation des patients, l’efficience de l’action publique, les progrès de la recherche et la capacité de notre pays à favoriser l’innovation et à rester compétitif en matière d’accueil d’entreprises du champ de la santé »[6]. Le Conseil national du numérique a également souligné les opportunités de l'open data en santé dans son rapport sur la Santé, bien commun de la société numérique remis au Ministre de la santé en octobre 2015[7].
Selon l’ordre national des médecins en France (lequel a consacré un débat à ce sujet lors d’un congrès de l'ordre des médecins, en octobre 2014[8]) l’accès aux données de santé anonymisées est nécessaire au pilotage des politiques de santé publique ; pour mieux répartir l’offre de soins sur les territoires, mieux dépister les effets secondaires de médicaments, améliorer le financement du système de santé, faire progresser la recherche médicale. Il est aussi nécessaire à l’évaluation, et à l’amélioration de la connaissance et de l’information du public[8]. L'open source, déjà très utilisé dans le domaine de la génomique et de l’analyse génétique[9] - [10] où les développements sont souvent trop rapides pour que les systèmes de brevetage ne soient pas des freins. Il a aussi des applications dans le domaine de l’imagerie médicale (ex. : Image J pour les applications biomédicales d’analyse d’imagerie microscopique) ou du calcul en bioinformatique[11]. Les progrès de la génétique et en particulier du séquençage ont été dopés par les démarches ouvertes qui ont permis des calculs massifs requis, faits par des équipes diverses et complémentaires parfois dispersées dans le monde[12]. Le rapport Bras considère que la donnée publique de santé (anonymisée) est « un bien public qui ne peut être approprié par aucun des acteurs du système. Il doit être administré dans l’intérêt commun par une autorité légitime en concertation avec l’ensemble des parties prenantes » (p. 7/128)[13]. Il admet que « Le recours au SI (système d'information) peut s’avérer un moyen économique pour établir certains résultats qui ne peuvent aujourd’hui être approchés qu’en organisant des études en vie réelle, lourdes et coûteuses » (p. 44/128)[13].
La médecine de catastrophe est chargée, souvent via des ONG de répondre aux tsunamis, tremblements de terre, désastres de guerre ou pandémies, ou désastres socio-écologiques liés aux effets du dérèglement climatique, de sécheresses et famines). Elle doit être très réactive et souvent agir avec peu de moyens dans des régions fonctionnant en « mode dégradé ». Dans ces contextes, elles peuvent déjà bénéficier de cartographies SIG, d'images satellitales et d'autres banques de données ouvertes[14], notamment grâce à un ensemble d’applications ouvertes spécialement dédiées au secteur de l'humanitaire (HFOSS - Humanitarian free and open source software) dont le développement s'appuie sur deux communautés, la communauté de l’humanitaire et celle de l’Open-source[15] dans un contexte principalement géré par des ONG, agences gouvernementales et groupes de civils ou militaires. Elle peut utiliser des sources et matériels médicaux mais aussi des données et supports ouverts dont notamment OpenStreetMap pour la cartographie collaborative et participative grâce notamment au réseau (lancé en 2009 par les Drs Patrick Meier et Jen Ziemke ; cofondateurs) des « cartographes de crise » (Crisis mappers[16]).
La communauté des développeurs pourrait jouer un rôle clé dans le développement d’une informatique médicale ouverte[17] plus ouverte[18].
Larges retombées pour la société
Au-delà du secteur médical et des professionnels de santé (qui n'auront en France accès au réseau d'information non anonymisée qu'à certaines conditions et après s'être identifié par leur Carte de professionnel de santé (CPS), toute la société est potentiellement concernée par les retombées directes et indirectes des approches d’innovation ouverte (quand elles sont vraiment « ouvertes »[19]) s’appliquent déjà ou pourraient s’appliquer aux patients, aux étudiants[20]. C'est le cas de certains secteurs de la pharmacie et à toute la recherche biomédicale[21], via par exemple le Web 2.0 et ou sémantique ou encore la réalité virtuelle ou augmentée.
Moyens et outils
Le Conseil national du numérique propose de mettre l'accent sur l’innovation et la recherche ouvertes dans le champ de la santé[7], c'est-à-dire d'encourager la constitution de patent pools (ou communautés de brevets) pour les innovations numériques en santé, de promouvoir l’open access pour les publications de recherche financées par des acteurs publics, ou encore stimuler la recherche ouverte en mettant à la disposition des chercheurs des espaces de travail en commun (plateformes de type co-laboratoires distribués, plateformes de publication en open source)
Publications scientifiques et techniques « ouvertes »
Les publications en open access sont un des moyens d'accélérer et élargir la diffusion et l'appropriation des informations et innovations du domaine médical (et d'autres) ; elles facilitent la valorisation et l'amélioration de leur contenu. C'est une stratégie par exemple développée par BioMed Central puis également par la Public Library of Science (PLOS), avec PLOS Biology, PLOS Medicine, PLOS One, etc.
En complément de la revue par les pairs[22], c’est aussi un moyen de limiter les fraudes, comme de détecter et corriger plus rapidement les erreurs.
La transparence et la traçabilité souvent associées à l'Open data limitent le risque de biais liés à des conflits d'intérêts.
Plates-formes pédagogiques en ligne
La création et l’animation de telles plates-formes en ligne[23], éventuellement sous forme de e-learning center, et accompagnées de MOOC[24] - [25], etc. peut s'appuyer sur les publications ouvertes (ou prépublication[26]) et bases de données ouvertes.
De telles plates-formes, si elles comprennent des outils collaboratifs et participatifs (dont Wikipédia, Wikibooks et Wikiversité peuvent faire partie) peuvent faciliter un coworking rapide et asynchrone[27].
Logiciels médicaux libres
Ils sont de plus en plus nombreux dans le domaine biomédical[28] - [29], et certains sont en développement pour un accès amélioré aux données médicales[30], pour la télémédecine et la télésanté, la gestion locale et quotidienne d’actes médicaux, dont en hôpital[31], et en lien avec les programmes nationaux de santé, par exemple aux Philippines[32], ou au service de la recherche en passant par le biomonitoring d’animaux de laboratoires[33], etc. Des systèmes d'information géographique « libres » ont été utilisés pour lutter contre le VIH[34] et sont depuis utilisés pour d’autres maladies.
Matériel médical libre
Le matériel libre est souvent moins cher et peut être rapidement amélioré par les communautés d'utilisateurs et de patients. De nombreux projets ont émergé dans les années 2000-2010, dont à titre d'exemple :
- Open Prosthetics Project : Plusieurs projets de conception et/ou production de prothèses libres ont montré leur efficacité et permis de créer des prothèses à des prix plus abordables pour les populations pauvres[35] - [36] ;
- Open Myoelectric Signal Processor : Interface matériel pour détecter et utiliser les signaux électriques du corps et commander des prothèses mobiles[37].
- Whirlwind Wheelchair International : ce projet crée des fauteuils roulants bien moins chers selon des plans libres faciles à améliorer ou adapter à des besoins particuliers (design mis sous licence Domaine public)[38].
- Open ECG project : Ce projet vise à développer un matériel open source et des solutions logicielles pour l'électrocardiographie[39].
- OpenPCR : projet visant à créer une machine à dupliquer l'ADN[40]
- Open EEG (Électro-encéphalographie)[41].
- Pandemic Ventilator Project : En cas de pandémie grippale, les moyens d'assistance respiratoire seront rapidement insuffisant notamment dans les pays pauvres, d'où l'idée de produire et diffuser sous licence libre de tels matériels[42].
- Generic Infusion Pump (GIP) ; ce projet vise à concevoir des modèles et prototypes génériques de pompe à perfusion (externe ou implantable)[43] pour injecter à des patients des analgésiques ou d'autres médicaments, développé avec le soutien de NSF CNS-0610297 ("Applying Formal Methods to Improve the Quality of Software in Medical Devices") et de NSF CNS-0834524 (Robust Composition and Interoperability of CPS Components) ;
- Open-Source Medical Devices : initiative de l'université du Wisconsin-Madison, visant à produire des matériels médicaux open source et notamment un scanner corporel médical à haute résolution, combiné à une machine de radiothérapie[44] - [45] - [46] ;
- Robot chirurgical Raven : ce robot à bras multiples open source est en cours de développement à l'université de Washington, à Seattle[47].
- Programme d’interopérabilité Plug-and-Play : ce programme (Medical Device "Plug-and-Play" Interoperability Program working on "safe interoperabilityTM" to improve patient safety) lancé par le Dr. Julian Goldman part du constat que les nombreux appareils médicaux installés dans l'environnement clinique n'ont pas été conçus pour être interconnectés ni communiquer entre eux. Ce projet vise donc à développer l'interopérabilité dans le monde du matériel médical (libre ou non), pour notamment améliorer la sécurité d'utilisation de ces matériels par exemple pour le diagnostic, la délivrance de traitements, la détection d'anomalies, la recherche médicale, etc., tout en facilitant le travail des équipes de soignants[48]. Il développe des bases de données, des outils, des tests et des applications partageables. Il est soutenu par une initiative financée à hauteur de 10 millions de dollars par les instituts nationaux de la santé des États-Unis avec l’aide de la FDA) afin de mettre en œuvre des standards ouverts[48]
- Plate-forme unique de matériels médicaux coordonnés : Ce projet (Medical Device Coordination Framework), en lien avec le précédent, part également du principe que les matériels médicaux ont été conçus, approuvés et distribués comme des monolithes individuels, alors qu'ils gagneraient à pouvoir communiquer entre eux. L’université d’État du Kansas se propose ici de créer une plate-forme matérielle open source très adaptable, grâce à un logiciel tiers (Middleware) qui crée et gère un réseau d'échange d'informations entre les différentes applications informatiques de la plate-forme, selon une architecture flexible publier-souscrire. Cette plate-forme pourra accueillir des capteurs, des matériels, des logiciels et des écrans. Ils seront interchangeables et capables de fonctionner selon des boucles et scénarios personnalisables et capables de communiquer entre eux (pour effectuer ensemble des taches automatisées par exemple) et avec le dossier médical du patient[49] (et en France les données de son Parcours de soins coordonnés...
« Médicaments libres » et « pharmacie virtuelle »
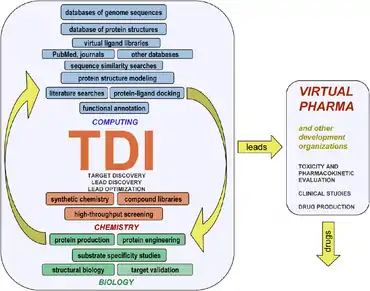
Au début du XXIe siècle, seuls 1 % des médicaments récemment mis au point et commercialisés concernent des maladies tropicales[51] et le modèle économique intégré des grandes entreprises pharmaceutiques est de plus en plus considéré comme insoutenable. Ceci engage les gouvernements, les institutions académiques mais aussi de grandes et petites entreprises de l’Industrie pharmaceutique à envisager de changer de modèle, par exemple en optant pour les modèles d’«innovation ouverte» qui ont fait leurs preuves dans d'autres domaines[52].
Une étude publiée en 2004 dans PlosOne[50] conclut que du point de vue scientifique, rien se semble en effet s’opposer à la découverte de nouveaux médicaments pour ces maladies que l’industrie pharmaceutique ne veut pas travailler, et d’autres maladies dites « négligées », grâce aux méthodes de l'Open source et de certaines sciences participatives. Les auteurs estiment que s’il existe des risques, ils semblent acceptables[50].
Ils ajoutent cependant que les projets doivent rester motivants pour les bénévoles et personnes qui y contribuent et que pour perdurer, ils exigent une certaine « masse critique » de participants (plusieurs dizaines de membres actifs) ainsi que des compétences minimales en chimie et biologie pour augmenter les chances de succès.
Développer un médicament exige en outre une ressource financière minimale (de plusieurs centaines de milliers de dollars par an), mais celle-ci peuvent en grande partie être apportée sous la forme de dons en nature ; de bases de données, d’accès au laboratoire, et de temps d’accès à une plateforme informatique[50].
L’approche « Virtual Pharma » évoquée par cette étude serait soutenue par des gouvernements et des organisations philanthropiques, dont par exemple ;
- l'Institut One World Health (www.iowh.org), une société pharmaceutique à but non lucratif principalement financée par des fonds privés et la Fondation Gates[53]
- le projet « Drugs for Neglected Diseases Initiative » dit Drugs for Neglected Diseases Initiative[54], (www.dndi.org), initié par le secteur public et des ONG afin de mobiliser des ressources pour la R & D sur de nouveaux médicaments pour les « maladies négligées », dont la leishmaniose, la maladie du sommeil, la maladie de Chagas et le paludisme ;
- le « Fred Hutchinson Cancer Research Centre » de Seattle, qui abrite l’ONG Sage Bionetworks, qui a été pionnière en matière de partage de données médicales en système open source.
- le réseau « Opensource Drug Discovery »[55] qui vise à trouver des médicaments opensource et libres, en mode collaboratif, notamment pour de nouveaux antituberculeux. Les découvertes résultant de ces recherches ne seront pas brevetées, mais au contraire mises à la disposition de tous, dans la « pharma virtuelle », ce qui facilitera le développement et la production dans les pays touchés eux-mêmes, voire une production in situ dans les régions touchées.
Ces projets de pharmacies virtuelles sont cependant confrontés à trois problèmes importants
- il est difficile de dimensionner à l’avance les moyens nécessaires en R & D (ce problème existe aussi dans les filières commerciales) ;
- ces projets nécessitent idéalement un accès aux recherches faites en amont, or elles ont été particulièrement ténues dans l'exploitation des connaissances génomiques relatives aux « maladies délaissées » jugées non rentables pour l'Industrie pharmaceutique ;
- le secteur des maladies tropicales est en outre chroniquement mal équipé et ou sous-financé. Les projets de pharmacie virtuelle doivent donc avoir de solides assises financières.
Les licences retenues les projets open sont généralement de type Creative Commons ou « domaine public », mais il semble possible en fin de processus d’utiliser des licences commerciales pour la production de ces médicaments hors des pays pauvres, de manière que la « rente » ainsi obtenue réalimente le système afin d’améliorer ou de trouver d’autres médicaments pour les maladies encore délaissées.
En mai 2010, deux grandes compagnies pharmaceutiques ont pour la première fois déposé dans une base de données ouverte les formules de centaines de molécules qui pourraient être des candidats médicaments contre la malaria[56].
Certains auteurs invitent cependant à vérifier que quand les grandes compagnies pharmaceutiques s’associent à des démarches collaboratives, elles le fassent de manière éthique et équilibrée : Ladrum et al. en 2011 invitaient à vérifier qu’elles ne fassent pas que prendre dans ce type de situation, mais qu’elles donnent aussi[57].
Défis scientifiques et techniques
Pour lutter contre les grandes épidémies (ex. : grippe pandémique) ou des virus hautement pathogènes tels qu’Ebola en 2014, selon B Muniios, « Nous devons unir nos forces et intensifier nos efforts de recherche à un niveau jamais atteint auparavant. Nous devons mobiliser les ressources intellectuelles et créatives de la communauté scientifique mondiale, et les appliquer à la maladie »[58]. Selon lui une R&D « Open source » pourrait revigorer la recherche sur les médicaments[59] grâce aux méthodes nouvelles de partage et de travail collaboratif[60], en s'appuyant par exemple sur la plate-forme InnoCentive (en) qui a plus de 300 000 « solveurs » en veille et qui via ses partenariats avec le groupe Nature Publishing et le journal Scientific American peut rapidement toucher plus de 13 millions de scientifiques dans le monde. Depuis 2001, cette plate-forme a résolu plus de 1 500 problèmes complexes jusqu'alors jugés décourageants, pour 40 millions de dollars – soit moins de 30 000 $ par problème de moyenne.
Un besoin de financement complémentaire peut être trouvé par crowdsourcing.
Les défis à relever pour la médecine du XXIe siècle incluent notamment :
- l'invention et la production en nombre de tests de diagnostic rapide et d’autodiagnostic simples, efficaces et peu coûteux. Ils doivent détecter mieux et plus rapidement les malades et les épidémies (ex. : grippe[61], virus Ebola[58]…) ;
- Identifier des principes et médicaments actifs (même partiellement) contre des maladies aujourd’hui sans médicaments ou à traitements lourds, longs et couteux (ex : pour le virus Ebola, parmi au moins 2000 médicaments potentiels ; ou déjà approuvé par la FDA pour d'autres usages[58]) ;
- la mise au point et la diffusion de méthodes mieux adaptées à la gestion des crises médicales et épiémiologiques dans des pays pauvres, ou pauvres en médecin et en infrastructures médicales[58], en particulier pour la médecine essentielle (qui serait alors moins coûteuse et plus efficace[62]).
Il faut pour cela :- - inventer pour les soignants des équipements de protection individuelle (EPI) plus efficaces (plus sûrs, moins encombrant, lavables et réutilisable et adaptés aux pays chauds ou froids. Pour les pays chauds, ces tenues doivent intégrer un système (passif et simple) de refroidissement ou de climatisation et être faite d'un matériau respirant[58] ;
- - inventer des combinaisons de protection réutilisables pour l'entourage du malade à la maison[58] ou utilisables durant le transport vers un centre de soin ;
- - inventer ou adapter des centres de soins à bas coût, incluant des unités d'isolement, du matériel de stérilisation, des incinérateurs et systèmes efficaces d'élimination des déchets de soins, facilement et rapidement déployables où ils sont nécessaires, y compris en zones rurale ou forestière[58] ;
- - inventer des moyens de faciliter la vérification de la crédibilité de l'information médicale trouvée sur l'Internet[63] - [64]
- - certifier la sûreté des logiciels associés (ex. : le programme Open Source Hardening Project conduit par l’université Stanford et la société Coverity, soutenu par le département américain de la Sécurité intérieure (DHS) et le gouvernement américain (pour environ 300 000 dollars), a publié en 2008 une liste de 11 logiciels open source (Amanda, NTP, OpenPAM, OpenVPN, Overdose, Perl, PHP, Postfix, Python, Samba et TCL) dont le code a été jugé presque exempts de défauts de sécurité[65]. En 2012, les grands programmes Open-source contenaient plutôt légèrement moins de défauts de code logiciel que les programmes propriétaires, et à taille identique présentaient une qualité des logiciels apparemment équivalente[66].
- - inventer un moyen de prendre en compte les aspects juridiques et éthiques de la protection des données issues du dossier médical informatisé, tout en facilitant la veille sanitaire, la prévention, la pharmacovigilance, la pharmaco-épidémiologie, l’évaluation post-AMM et donc pour l'épidémiologie en général[67], de manière notamment à rendre la donnée médicale plus accessible aux statisticiens et écoépidémiologistes et communautés de malades[68] tout en la conservant « privée »[69], éventuellement au profit d’innovations inattendues (sérendipité)[70] - [71].
Pour qu'ils puissent vraiment accélérer les processus de R&D et d'applications et de permettre à la communauté mondiale de les améliorer de manière continue[58], ces méthodes et matériels doivent en outre être conçus avec un souci constant d’interopérabilité[72] et dans un esprit d'accès libre, ouvert (open data, matériel libre), solidaire[73] et transparent.
Pharmacie ouverte
Plusieurs projets ou propositions ont émergé sur ce thème depuis quelques années, avec l’idée notamment portée par B Munos[58] que les maladies tropicales ou intéressant peu l’industrie pharmaceutique car peu « solvables » (et donc non rentable selon les règles de l'industrie) pourraient en bénéficier[74], ce qui a notamment conduit au projet « Tropical Disease Initiative »[75] et à la création d’un consortium pour la découverte en open-source de médicaments pour la malaria (Open Source Drug Discovery for Malaria Consortium)[76]. Munos estime que dans le domaine de la pharmacie « Open Source Pharma » (une initiative lancée par le réseau des Open Society Foundations et soutenue par la fondation Rockefeller) pourrait être mobilisée[58].
Génomique ouverte
L’expression anglophone open-source genomics décrit la partie de la génomique qui exploite avec les bioinformaticiens les progrès rapides du séquençage et en mode de travail plus ou moins collaborative des milliards de données ainsi extraites des génomes de différentes espèces, dont certaines présentent un intérêt médical évident (c’est ainsi qu’on a par exemple décrypté le génome de variants pathogènes de la bactérie E. coli produisant des Shigatoxines[77]). L'Open data est largement pratiquée dans le domaine de la génomique (avec par exemple le GBrowse de Lincoln Stein, une interface WEB open-source dédiée au genome project database ou OBiBa dédié aux biobanques ou encore Generic Model Organism Database, une boîte à outils de composants logiciels open-source permettant de visualiser, annoter, gérer et stocker des données biologiques) , mais le brevetage du vivant est également pratiqué, permettant à certains de s'enrichir en profitant d'outils gratuitement mis à disposition par d'autres.
Débats et controverses
L'open source est généralement plébiscité dès lors que les données personnelles sont protégées, et il est devenu l'une des dimensions de la médecine dite « médecine 2.0 »[78]. Il est cependant au moins pour partie controversé des années 2000 à 2010.
Une nouvelle médecine connectée incluait déjà en 2008 notamment des applications créées par des groupes privés tels que Google Health, Microsoft (Microsoft HealthVault, où le patient décide qui peut voir, utiliser, ajouter et partager les informations, et quelles applications relatives à la santé y ont accès) ou des groupements d'entreprises privées (ex. : Dossia et son système Health Management System™)[79] et il est généralement admis que l'open data peut aussi être un des moyens d'améliorer certaines gestion de crise médicale (notamment dans les pays pauvres), si la vie privée est réellement protégée. Il peut contribue à développer et déployer la médecine préventive, la médecine pré-hospitalière[80] et hospitalière, la pharmacie traditionnelle et moderne[81] ainsi que l'écoépidémiologie, l’épidémiologie participative[82], et notamment concernant certaines maladies tropicales[83] prévalentes dans des pays qui n'en ont pas les moyens financiers, avec toutefois un défi à résoudre concernant la protection des données personnelles.
Dans un contexte où le big data augmente de manière exponentielle, certains, comme Guigue & Richard en 2014 envisagent déjà « une médecine 3.0 », capable d’exploiter le Big data[84]. Une utilisation intelligente de ces données serait selon R Walberg une source d’économie et pourrait sauver de nombreuses vies[85]. Selon P Groves & al. (de McKinsey & Company) en 2013, c’est aussi un moyen de doper et d’accélérer l’innovation en santé[86].
Des entreprises privées comme Celtipharm voudraient avoir directement accès au flux de données butes (et non au SNIIRAM où les données sont mises à disposition après validation qu’après 2 mois) pour améliorer leur veille syndromique et de « data mining ».
D’autres souhaitent l’accès aux données anonymisées mais ne veulent pas que ces données essentiellement fournies par le secteur public soient appropriables par le secteur privé sans contreparties (qui peuvent être précisées par les licences).
En France
Les termes du débat semblent se préciser dans les années 2010 :
Peu après la publication d’une tribune du journal Le Monde du 16 janvier 2013, intitulée « Plus grave que le débat sur la pilule, l'affaire des données de santé publique », dans laquelle Jean de Kervasdoué (professeur au CNAM), et Didier Sicard (président d'honneur du Comité consultatif national d'éthique) citaient plusieurs effets délétères de la rétention des données publiques de santé (dont par l'État et l'Assurance maladie), une pétition-manifeste intitulée « Initiative transparence santé », a été lancée en janvier 2014, à la fois défendue par des associations de patients et des complémentaires, plaidant pour un accès de toute la société civile aux données publiques de santé (anonymisées), et en toute transparence.
Un débat public avait été annoncé sur ce thème (pour 2013), et un rapport dit « Rapport Bras »[13] était en préparation.
Fin 2013, une commission « Open data en santé », installée et missionnée par la ministre Marisol Touraine, a travaillé de novembre 2013 à mai 2014 pour proposer « une vision commune » (appropriable par l’ensemble des acteurs selon Henri Verdier, directeur d’Etalab, le service rattaché à Matignon chargé de l’ouverture des données publiques et de la promotion de l’open data). Le rapport produit par cette commission invite les producteurs de données à prévoir l’ouverture de ces données dès leur fabrication, conformément à une obligation européenne d’ouverture des données publiques « non sensibles » d’ici au 18 juillet 2015, cette obligation étant déjà effective depuis plus de 10 ans, en application de la Convention d'Aarhus, pour certaines données concernant la santé environnementale.
Il invite aussi les établissements publics producteurs de données de santé à établir un échéancier d’ouverture de ces données (une fois anonymisées) avec l’appui d’un correspondant open data[1].
Parmi les principales bases de données françaises concernées, figurent celles tenues par l'Agence technique de l'information sur l'hospitalisation (Atih) et le Système national d'information interrégimes de l'assurance maladie (SNIIRAM, 450 téraoctets notamment alimentés annuellement par environ 1,2 milliard de feuilles de soins et autrefois très peu accessible en raison d’un arrêté du 20 juin 2005, qui a été cassé par une décision contraire du Conseil d'État rendue le 19 juillet 2013) et le Programme de médicalisation des systèmes d'information (PMSI). Au-delà de ces grandes bases, selon Etalab, ce sont 240 bases de données qu’il faut anonymiser, jusqu’alors sous la responsabilité de la Caisse nationale d’assurance maladie (Cnam).
« Gratuité des données » : Ce principe a été validé en 2013[87], mais pose question en ce qu'il fait reposer les couts d’anonymisation sur le secteur public[88].
Lancement de l'Open data : Le rapport rendu en 2014 sur le thème open data en santé encourage une diffusion prudente et responsable mais proactive et des données de santé (notamment à partir des bases du SNIIRAM et du PMSI.
Mi-décembre 2014, l'Assurance maladie a déposé sur www.data.gouv.fr une série de données de santé open data sur l’offre et la consommation de soins en France, afin d'améliorer la transparence de la prescription médicamenteuse, de la traçabilité des principes actifs et leur date de commercialisation[89]. Un Hackathon devrait être organisé en 2015 (défi et course à la meilleure application lancé aux développeurs qui pourront être assistés de chercheurs, d’informaticiens et de designers).
Un programme dénommé Territoire de soins numérique, financé à hauteur de 80 millions d'euros (Investissements d'avenir) pourra profiter de l'ouverture des données. Il doit moderniser le système de soins en testant dans quelques zones pilotes « les services et les technologies les plus innovants en matière d’e-santé » (sur 18 projets proposés par les Agences régionales de santé, 5 ont été retenus en 2014).
En 2015, le parlement débattra du « projet de loi de santé » (présenté le 15 octobre 2014 en Conseil des ministres[90]) qui propose notamment la création d'un « Système national des données de santé » (SNDS) qui devrait unifier les bases de données existantes, et serait gérée par la Caisse nationale de l'assurance maladie des travailleurs salariés (CNAMTS) et qui devrait mettre en Open Data) les données anonymisées qui peuvent l'être (avec une ouverture graduelle des données, en fonction du risque de réidentification des patients)[91]
Débats sur le cadre légal (français, de 2013-2014)
Le cadre principal est en France la « loi Informatique et libertés » et l’avis obligatoire de la Commission nationale de l’informatique et des libertés (Cnil), gardienne de la confidentialité des données de santé.
Des commissions d’experts et l’Institut des données de santé (IDS) donnent, également et avant la CNIL, des avis.
Des sanctions sont prévues en cas d’infraction à la confidentialité des données.
Ces règles, à condition d’être bien appliquées sont selon le « rapport Bras » de 2013 suffisantes[13], mais le monde informatique évolue rapidement. Sous l’égide de la CNIL, une politique générale de sécurité des systèmes d’information en santé (PGSSI-S) est régulièrement mise à jour pour suivre l’évolution du droit et des techniques, et mettre à jour des référentiels de sécurité et des guides de bonnes pratiques relatives aux données médiales.
Des voix s’élèvent cependant, y compris dans le monde médical « pour réclamer le déploiement de solutions de sécurité de haut niveau et la création de nouveaux outils de régulation »[8], notamment pour le SNIIRAM), qui décrit l’offre et la consommation de soins. Le Dr Jacques Lucas (Vice-président du conseil national de l’Ordre des médecins, délégué général aux systèmes d'information en santé), rappelait en 2014 que pour certains travaux de recherche, avec un protocole dont les objectifs et la méthodologie ont été soigneusement examinés, le strict anonymat de la donnée peut parfois être discuté pour l’usage de ces données par certains acteurs (ex : Inserm, CNRS ou instituts de recherche en cancérologie), mais l’anonymisation doit être réintroduite avant le rendu des travaux de recherche, en France sous l’égide de l’Institut des données de santé. Le Conseil national du numérique a également remis son avis au gouvernement sur les conditions de l'open data en santé en octobre 2015.
Catégories de données à ouvrir en France (après anonymisation)[1]
| Typologie | Contenu |
|---|---|
| Santé publique et épidémiologie |
|
| Offre de soins |
|
| Consommation de soins |
|
| Finance & dépense médicale |
|
| Performance et opérations |
|
Freins à l’adoption de solutions opensourcées
Outre le poids des habitudes ou un souci de sécurité juridique (principalement lié à la crainte de réidentification et/ou d'un mésusage[92] éventuel des données), parmi les facteurs clés identifiés comme freins figurent :
- le poids des directives et orientations ministérielles (source par exemple identifiée au Canada par une étude conduite en 2009[93]), sans que leurs objectifs soient contestés (il se s’agit jamais d’ouvrir, sauf avec de grandes précautions, les données personnelles ou déterminants de santé (ex : facteurs socio-économiques et environnementaux) ou de risquer de créer de nouveaux facteurs de discrimination (comme cela fut le cas ou l’est encore pour vis-à-vis de séropositifs) ;
- une mauvaise informations de la part des responsables informatiques de niveau opérationnel concernant les fournisseurs de logiciels ou matériel libres (problème également identifiée au Canada en 2009)[93] ;
- Une solution open-source n’a d’intérêt pour l’innovation médicale que si elle est introduite dans un esprit d’ouverture et de collaborativité, et non comme simple moyen de réduction de coûts ;
- les difficultés de traiter des données médicales confidentielles et leurs bases de données[94] avec des approches et outils ouverts conçus pour l’ouverture et la transparence. Des progrès doivent être faits en matière d’anonymisation et de vérification de données.
Prospective
De nombreux prospectivistes s’attendent à de nouveaux bouleversements avec les développements probables des biotechnologies croisés à ceux de la bioinformatique, de la nanomédecine et la nanosanté.
Notes et références
- Ministère de la Santé (2013°, Présentation et Rapport de la commission Open data santé sur l’ouverture des données publiques de santé, remis à la ministre M Touraine le 9 juillet 2014 (PDF, 63 p)
- « Les données personnelles de santé en cinq questions » publication de l'ordre national des médecins, Paris, France, novembre 2014
- Hermann V (2014), « Apple interdit la commercialisation des données de santé HealthKit », www.nextinpact.com, consulté le 31 décembre 2014
- (en) « Clever technology can help solve two big problems in health care: overspending in the rich world and under-provisioning in the poor world » The Economist, 16 avril 2009.
- « health data constitutes a significant resource in most OECD countries and it makes economic and ethical sense to use this data as much as possible » OCDE, « Strengthening health information infrastructure for health care quality governance » OECD Health policy Studies 2013, p. 13
- Rapport de la commission « open data en santé » remis à la ministre Marisol Touraine, le 9 juillet 2014. (page 5 sur 63)
- La Documentation française, « La santé, bien commun de la société numérique - Construire le réseau du soin et du prendre soin », sur www.ladocumentationfrancaise.fr (consulté le )
- « Utilisation des données personnelles de santé : faire progresser la qualité, l'efficience et la sécurité des soins » ; source : page intitulée La confidentialité des données, enjeu majeur de la santé mobile, sur le site de l’Ordre des médecins (France), consulté 2014-12-30
- Saldanha, A. J. (2004) « Java Treeview—extensible visualization of microarray data » Bioinformatics 2004;20(17):3246-3248.
- (en) Kurtz S, Phillippy A, Delcher AL, Smoot M, Shumway M, Antonescu C. & Salzberg SL. « Versatile and open software for comparing large genomes » Genome biology 2004;5(2):R12.
- (en) Gentleman RC, Carey VJ, Bates DM, Bolstad B, Dettling M, Dudoit S, Zhang J. et al. « Bioconductor: open software development for computational biology and bioinformatics » Genome biology 2004;5(10):R80
- (en) Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Knight R. et al. « QIIME allows analysis of high-throughput community sequencing data » Nature methods 2010;7(5):335-336
- Septembre 2013 Rapport sur la gouvernance et l'utilisation des données de santé dit « rapport Bras » (PDF, 128 pages), écrit par Pierre-Louis Bras (inspecteur général des affaires sociales), avec le concours d’André Lothremis à la ministre des Affaires sociales et de la Santé en 2013
- (en) Garshnek V & Burkle FM, « Applications of telemedicine and telecommunications to disaster medicine historical and future perspectives » Journal of the American Medical Informatics Association 1999;6(1):26-37.
- (en) Chamindra de Silva (2010), « Humanitarian Free and Open Source Software », décembre 2010
- (en) The Crisis Mappers Network ; International Network of Crisis Mappers (Crisis Mappers Net)
- (en) Willison Donald J, Keshavjee Karim, Nair Kalpana, Goldsmith Charlie, Holbrook Anne M; Computerization of Medical Practices for the Enhancement of Therapeutic Effectiveness investigators. « Patients' consent preferences for research uses of information in electronic medical records: interview and survey data » BMJ 2003;326(7385):373. DOI 10.1136/bmj.326.7385.373 http://bmj.com/cgi/pmidlookup?view=long&pmid=12586673
- (en) Eysenbach G. « Consumer health informatics » BMJ 2000;320(7251):1713–6.
- Dahlander, L., & Gann, D. M. (2010). How open is innovation?. Research policy, 39(6), 699-709.
- Willinsky, J. (2003). «The nine flavours of open access scholarly publishing”. Journal of Postgraduate Medicine, 49(3), 263.
- Rai AK (2005) « Open and collaborative research: A new model for biomedicine ». In: Hahn R, editor. Innovation in frontier industries: Biotech and software. Washington : AEI-Brookings Press
- (en) (éditorial) « Peer review and fraud » Nature. 21 déc. 2006;444(7122):971–2. DOI 10.1038/444971b.444971b
- Brunet P, Cuggia M. & Le Beux P. « Collecte et baladodiffusion de ressources médico-pédagogiques à destination des étudiants en médecine de PCEM2 et de DCEM1 à l’université de Rennes 1 » in : Systèmes d’information pour l’amélioration de la qualité en santé, Paris, Springer, 2012, p. 343-352.
- (en) RF Heller, « Learning by MOOC or by crook », Med J Aust, vol. 200, no 4, , p. 192-3. (PMID 24580509, DOI 10.5694/mja14.00129, lire en ligne [html])
- (en) J Reich, « Education research. Rebooting MOOC research », Science, vol. 347, no 6217, , p. 34-5. (PMID 25554779, DOI 10.1126/science.1261627)
- (en) Eysenbach G. « Challenges and changing roles for medical journals in the cyberspace age: electronic pre-prints and e-papers » J Med Internet Res. 1999 Dec 31;1(2):E9. DOI 10.2196/jmir.1.2.e9
- Rafaël Pinilla, Application du système "wiki" à la mise en place d'une base de connaissance médicale, France, Paris Diderot - Paris 7 UFR de médecine, site Xavier-Bichat, coll. « thèse d'exercice », , 75 p. (OCLC 492102366)
- (en) McDonald CJ, Gunther S, Barnes M et al. « Open Source sofware in medical informatics - why, how and what » IJMI. 2003;69:175-184.
- (en) Meystre S & Müller H, « Open source software in the biomedical domain: Electronic health records and other useful applications » Swiss Medical Informatics 2005;55(3):1-25. pdf disponible à publications.hevs.ch
- (en) Yi Q, Hoskins RE, Hillringhouse E et al. « Integrating open-source technologies to build low-cost information systems for improved access to public health data » International Journal of Health Geographics 2008;7:29 (cc-by-sa 2.0
- (en) Bagayoko CO, Dufour JC, Chaacho S, Bouhaddou O & Fieschi M, « Open source challenges for hospital information system (HIS) in developing countries: a pilot project in Mali » BMC medical informatics and decision making 2010;10(1):22.
- (en) Tolentino H, Marcelo A, Marcelo P, Maramba I, « Linking Primary Care Systems and Public Health Vertical Programmes in the Philippines: An Open-source Experience » AMIA. 2005;311-315.
- (en) Poirrier JE, Poirrier L, Leprince P, Maquet P, « Gemvid, an open source, modular, automated activity recording system for rats using digital video » J Circadian Rhythms. 2006 Aug 25;4:10.
- (en) Vanmeulebrouk B, Rivett U, Richetts A, Loudon M, « Open source GIS for HIV/AIDS management » International Journal of Health Geographics 2008;7:53. DOI 10.1186/1476-072X-7-53
- site
- Article : Des prothèses libres pour mieux vivre (framablog)
- site
- site
- Open ECG project
- OpenPCR
- site
- site
- The Generic Infusion Pump (GIP)
- scanner corporel médical haute résolution combiné à une machine de radiothérapie
- Prajapati, S, Seaton, J, Sekhon, J, Mackie, T and Jeraj, R. 'Development of Binary Micro MLCs for an Open Source Small Animal Imaging and Therapy System with IMRT Capability', Medical Physics, 38, 3862-3862 (2011), DOI: https://dx.doi.org/10.1118/1.3613545
- Prajapati, S, Cox, B, Cadman, P, Jeraj, R and Mackie, T. 'Design of Open‐Source Binary Micro MLC for Small Animal Radiotherapy: An OSMD Initiative', Medical Physics, 40, 489-489 (2013), DOI: https://dx.doi.org/10.1118/1.4815585
- Surgical robots to provide open-source platform for medical robotics research
- Medical Device Plug-n-Play Interoperability Program (MDPnP) (Programme d’interopérabilité Plug-and-Play du matériel médical)
- La structure de coordination du matériel médical
- Maurer SM, Rai A, Sali A (2004) « Finding Cures for Tropical Diseases: Is Open Source an Answer? » PLoS Med 1(3): e56. doi:10.1371/journal.pmed.0010056, article en open-acces CC-BY-SA
- Trouiller O, Olliaro PL (1999) Drug development output from 1975 to 1996: What proportion for tropical diseases? Int J Infect Dis 3: 61–63.
- Hunter, J., & Stephens, S. (2010). Is open innovation the way forward for big pharma?. Nature Reviews Drug Discovery, 9(2), 87-88 (résumé)
- site
- site
- site
- Strauss, S. (2010). Pharma embraces open source models. Nature biotechnology (News), 28(7), 631-634
- Landrum, G. A., Lewis, R., Palmer, A., Stiefl, N., & Vulpetti, A. (2011) Making sure there's a "give" associated with the "take": producing and using open-source software in big pharma. J. Cheminformatics, 3(S-1), O3 ; Présentation orale, nov. 2010, 6th German Conference on Chemoinformatics, GCC 2010
- Munos B (2014), Beyond Heroism And Denial: How To Fortify Our Response To Ebola, Forbes (10/16/2014), propositions et défis repris par le journal Nature, parmi les défis urgents à relever dans le cadre de son programme InnoCentive
- Munos, B. (2006). «Can open-source R&D reinvigorate drug research?”. Nature Reviews Drug Discovery, 5(9), 723-729
- Eysenbach G. An ontology of quality initiatives and a model for decentralized, collaborative quality management on the (semantic) World-Wide-Web. J Med Internet Res. 2001 Dec 31;3(4):E34. doi: 10.2196/jmir.3.4.e34. http://www.jmir.org/2001/4/e34/
- Bourgeois Florence T, Simons William W, Olson Karen, Brownstein John S, Mandl Kenneth D. Evaluation of influenza prevention in the workplace using a personally controlled health record: randomized controlled trial. J Med Internet Res. 2008;10(1):e5. doi: 10.2196/jmir.984. http://www.jmir.org/2008/1/e5/v10i1e5
- Flynn, S., Hollis, A., & Palmedo, M. (2009). An economic justification for open access to essential medicine patents in developing countries. The Journal of Law, Medicine & Ethics, 37(2), 184-208
- Eysenbach Gunther. From intermediation to disintermediation and apomediation: new models for consumers to access and assess the credibility of health information in the age of Web2.0. Stud Health Technol Inform. 2007;129(Pt 1):162–6
- Eysenbach G, Diepgen T L. Towards quality management of medical information on the internet: evaluation, labelling, and filtering of information. BMJ. 1998 Nov 28;317(7171):1496–500. http://bmj.com/cgi/pmidlookup?view=long&pmid=9831581
- Aurélien Cabezon (2008) 11 projets open source certifiés comme sûrs, le 10 janvier 2008
- Cyrille Chausson (2012), Qualité logicielle : Open Source et propriétaire dans un mouchoir de poche, selon Coverity 29 févr. 2012
- Picard, S., Pellet, J., Brulet, J. F., & Trombert, B. (2006). Les aspects juridiques et éthiques de la protection des données issues du dossier médical informatisé et utilisées en épidémiologie : un point de la situation. Santé publique, 18(1), 107-117 (résumé)
- Frost Jeana H, Massagli Michael P. Social uses of personal health information within PatientsLikeMe, an online patient community: what can happen when patients have access to one another's data. J Med Internet Res. 2008;10(3):e15. doi: 10.2196/jmir.1053. http://www.jmir.org/2008/3/e15/v10i3e15
- Mandl K D, Szolovits P, Kohane I S. Public standards and patients' control: how to keep electronic medical records accessible but private. BMJ. 2001 Feb 3;322(7281):283–7. doi: 10.1136/bmj.322.7281.283. http://bmj.com/cgi/pmidlookup?view=long&pmid=11157533.
- Heverly RA. McPherson T, ed. “Digital Youth, Innovation, and the Unexpected. The John D and Catherine T MacArthur Foundation Series on Digital Media and Learning”. Cambridge, MA: MIT Press; 2008. Growing Up Digital: Control and the Pieces of a Digital Life.
- Eysenbach G. Metzger MJ, Flanagin AJ, editors. Digital Media, Youth, and Credibility. The John D and Catherine T MacArthur Foundation Series on Digital Media and Learning. Cambridge, MA: MIT Press; 2008. Credibility of health information and digital media: new perspectives and implications for youth. http://www.mitpressjournals.org/doi/pdf/10.1162/dmal.9780262562324.123
- Patel A, O'Brien R, Jones P, Quintana Y (2003), Interorability of Open Source Medical Record Systems. AMIA Symposium Proceedings965
- Benkeltoum, Nordine (2009), Les Régimes de l'open source : solidarité, innovation et modèles d'affaires, thèse de doctorat en sciences de gestion, Centre de gestion scientifique, Mines ParisTech. Texte en ligne « Copie archivée » (version du 22 juillet 2018 sur Internet Archive)
- Maurer, S. M., Rai, A., & Sali, A. (2004). Finding cures for tropical diseases: Is open source an answer?. PLoS Medicine, 1(3), e56
- the Tropical Disease Initiative ; Tropicaldisease.org 2009-07-16
- The Open Source Drug Discovery for Malaria Consortium ; Openwetware.org ; 2012-12-14
- Rohde H & al. (2011), Open-Source Genomic Analysis of Shiga-Toxin–Producing E. coli O104:H4 ; N Engl J Med N°8, pp 18–24 ; doi=10.1056/NEJMoa1107643 ;
- Eysenbach Gunther. Medicine 2.0 Congress Website. webcite http://www.medicine20congress.com/ (voir aussi : éléments de définition)
- Hughes, B., Joshi, I., & Wareham, J. (2008). Health 2.0 and Medicine 2.0: tensions and controversies in the field. Journal of Medical Internet Research, 10(3)
- Zellner, P., Béchet, A., & Belle, L. (2005). Res@ mu - Une Solution Open Source pour la médecine pré hospitalière. First He.
- Levison, L., & Fraser, H. S. (2008). Requirements for an Open-Source Pharmacy Dispensing and Stores Management Software Application for Developing Countries. In Making the eHealth Counection Conference, Bellagio, Italy, July l3-Aug (Vol. 8)
- Freifeld, C. C., Chunara, R., Mekaru, S. R., Chan, E. H., Kass-Hout, T., Iacucci, A. A., & Brownstein, J. S. (2010). Participatory epidemiology: use of mobile phones for community-based health reporting. PLoS medicine, 7(12), e1000376.
- Maurer, S. M., Rai, A., & Sali, A. (2004). Finding cures for tropical diseases: is open source an answer?. PLoS Medicine, 1(3), e56.
- Guigue, L., & Richard, C. (2014). Le Big data en Santé préfigure-t-il la «médecine 3.0»?. HEGEL [ISSN 2115-452X], 2014, 3 Rem : les auteurs sont membres du groupe de travail open data en santé/big data en santé du SYNTEC
- Walberg R. Value of big data in health care is measured not just in dollars, but in lives. Financial Post, 5 février 2014 financialpost.com/2014/02/05/value-of-big-data-in-health-care-is-measured-not-just-in-dollars-but-in-lives/?__lsa=c350-1536 lien « Copie archivée » (version du 22 juillet 2018 sur Internet Archive)
- Groves P, Kayyali B, Knott D. The ‘big-data’ revolution in healthcare: Accelerating value and innovation. McKinsey & Company, janvier 2013 http://www.mckinsey.com/insights/health_systems/~/media/7764A72F70184C8EA88D805092D72D58.ashx
- Décisions 24, 25 et 26 du CIMAP du 18 décembre 2013
- Source: http://www.ticsante.com/Open-data--le-debat-sur-l-ouverture-des-donnees-de-sante-prevu-en-septembre-NS_1547.html#ixzz3NONrtmnx
- Page des données Open data apportées par la Caisse nationale de l'assurance maladie des travailleurs salariés sur le portail Data.gouv.fr
- Communiqué ministériel :Marisol Touraine présente le projet de loi de santé 15 octobre 2014
- Open Data : le projet de loi du gouvernement concernant les données de santé
- par mésusage, le rapport Bras « entend la diffusion d’informations erronées, biaisées et non le risque de réidentification »
- Paré G, Wybo MD, Delannoy C (2009), “ Barriers to Open Source Software Adoption in Quebec's Health Care Organization”. J Med Syst 33(1):1-7 (résumé)
- Hofman, V., Selva, E., Chabannon, C., Bonnetaud, C., Gaziello, M. C., Bordone, O., ... & Hofman, P. (2010). Les biobanques en France: enjeux et contraintes. Revue francophone des laboratoires, 2010(418), 67-72.
Voir aussi
Articles connexes
- Logiciel libre, Matériel libre (= open hardware)
- Donnée ouverte, licence Ouverte
- Open Source Initiative
- Shared Source
- Open Knowledge Foundation
- Big data
- CubicWeb
- Isidore
- OpenDataFrance
- Open Food Facts
- Médecine personnalisée
- Anonymat sur Internet
- Vie privée et informatique
- Secret statistique
- Donnée de santé
Liens externes
Rapports officiels (France)
- Rapport sur la gouvernance et l'utilisation des données de santé dit « Rapport Bras » (PDF, 128 pages), écrit par Pierre-Louis Bras (inspecteur général des affaires sociales), avec le concours d’André Lothremis à la ministre des Affaires sociales et de la Santé en 2013 ; daté septembre 2013
- Rapport de la Commission open data en santé, réalisé en lien avec la mission Etalab, remis à la ministre Marisol Touraine, le 9 juillet 2014.
Bibliographie
- Allard, J., Cotin, S., Faure, F., Bensoussan, P. J., Poyer, F., Duriez, C., ... & Grisoni, L. (2007). Sofa-an opensource framework for medical simulation. In Medicine Meets Virtual Reality, MMVR 15.
- Boulos, M. N., Maramba, I., & Wheeler, S. (2006). Wikis, blogs and podcasts: a new generation of Web-based tools for virtual collaborative clinical practice and education. BMC medical education, 6(1), 41.
- Bursac, Z., Gauss, C. H., Williams, D. K., & Hosmer, D. W. (2008). Source Code for Biology and Medicine. Source code for biology and medicine, 3, 17.
- Cook, P. A., Bai, Y., Nedjati-Gilani, S. K. K. S., Seunarine, K. K., Hall, M. G., Parker, G. J., & Alexander, D. C. (2006, May). Camino: open-source diffusion-MRI reconstruction and processing. In 14th scientific meeting of the international society for magnetic resonance in medicine (Vol. 2759) ([Cook, P. A., Bai, Y., Nedjati-Gilani, S. K. K. S., Seunarine, K. K., Hall, M. G., Parker, G. J., & Alexander, D. C. (mai 2006). Camino: open-source diffusion-MRI reconstruction and processing. In 14th scientific meeting of the international society for magnetic resonance in medicine (Vol. 2759).
- Gitter, D. M. (2006). Resolving the open source paradox in biotechnology: A proposal for a revised open source policy for publicly funded genomic databases. Hous. L. Rev., 43, 1475 (http://heinonlinebackup.com/hol-cgi-bin/get_pdf.cgi?handle=hein.journals/hulr43§ion=48 résumé])
- Craig, R., Cortens, J. P., & Beavis, R. C. (2004). Open source system for analyzing, validating, and storing protein identification data. Journal of proteome research, 3(6), 1234-1242 (http://pubs.acs.org/doi/abs/10.1021/pr049882h résumé]).
- Falkman G, Gustafsson M, Jontell M, Torgersson O. A Semantic Web-Based System for Supporting Collaboration of Distributed Medical Communities of Practice. J Med Internet Res. 2008;10(3):e25. doi: 10.2196/jmir.1059.
- Hahn, R. W. (Ed.). (2010). Government policy toward open source software. Brookings Institution Press.
- Hood, L., & Flores, M. (2012). “A personal view on systems medicine and the emergence of proactive P4 medicine: predictive, preventive, personalized and participatory”. New biotechnology, 29(6), 613-624.
- Hope, J. (2009). Biobazaar: the open source revolution and biotechnology. Harvard University Press
- Hughes, B., & Wareham, J. (2010). Knowledge arbitrage in global pharma: a synthetic view of absorptive capacity and open innovation. R&d Management, 40(3), 324-343.
- National Institute for Health Care Management, Research and Educational Foundation (2002) Changing patterns of pharmaceutical innovation
- Rai AK (2005) « Open and collaborative research: A new model for biomedicine ». In: Hahn R, editor. Innovation in frontier industries: Biotech and software. Washington : AEI-Brookings Press
- Steinbeck, C., Hoppe, C., Kuhn, S., Floris, M., Guha, R., & Willighagen, E. L. (2006). Recent developments of the chemistry development kit (CDK)-an open-source java library for chemo-and bioinformatics. Current pharmaceutical design, 12(17), 2111-2120 (résumé).
- Swan, M. (2009). Emerging patient-driven health care models: an examination of health social networks, consumer personalized medicine and quantified self-tracking. International journal of environmental research and public health, 6(2), 492-525.
- Viceconti, M., Zannoni, C., Testi, D., Petrone, M., Perticoni, S., Quadrani, P., ... & Clapworthy, G. (2007). The multimod application framework: a rapid application development tool for computer aided medicine). Computer methods and programs in biomedicine, 85(2), 138-151