Sulfurimonas
Sulfurimonas est un genre de bactéries de la classe des Epsilonproteobacteria, connu pour réduire les nitrates, oxyder à la fois le soufre et l'hydrogène et contenir des hydrogénases du groupe IV[2] - [3] - [4]. Ce genre comprend quatre espèces : Sulfurimonas autorophica, Sulfurimonas denitrificans, Sulfurimonas gotlandica et Sulfurimonas paralvinellae. Le nom du genre est dérivé de « soufre » en latin et « monas » du grec, signifiant ensemble une « tige oxydant le soufre »[5]. La taille des bactéries varie environ de 1,5 à 2,5 μm de longueur et 0,5 à 1,0 μm de largeur[6] - [7] - [4].

| Règne | Bacteria |
|---|---|
| Embranchement | Proteobacteria |
| Classe | Epsilonproteobacteria |
| Ordre | Campylobacterales |
| Famille | Helicobacteraceae |
Espèces de rang inférieur
- Sulfurimonas autotrophica
- Sulfurimonas denitrificans
- Sulfurimonas gotlandica
- Sulfurimonas paralvinellae
- (Sulfurimonas Hongkongensi)
Les membres du genre Sulfurimonas se trouvent dans une variété d'environnements différents qui comprennent des évents marins profonds, des sédiments marins et des habitats terrestres[3]. Leur capacité à survivre dans des conditions extrêmes est attribuée à plusieurs copies d'une enzyme[3].
L'analyse phylogénétique suggère que les membres du genre Sulfurimonas ont une capacité de dispersion limitée et que sa spéciation a été affectée par l'isolement géographique plutôt que par la composition hydrothermale. Les courants océaniques profonds affectent la dispersion de Sulfurimonas spp., influençant sa spéciation[8]. Comme indiqué dans le rapport MLSA[9] des évents hydrothermaux d'eau profonde en 2017, l'Epsilonproteobacteria Sulfurimonas a une capacité de dispersion plus élevée par rapport aux thermophiles d'évents hydrothermaux d'eau profonde, indiquant une spéciation allopatrique[8].
Caractéristiques
| Espèce | Taille | Morphologie[10] | Mouvement | Dépendance à la température[6] | Conditions de l'habitat | Conditions optimales | Temps de doublement dans des conditions optimales (heures) |
|---|---|---|---|---|---|---|---|
| Sulfurimonas autotrophica | 1,5-2,5 x 0,5-1,0 m[5] | Tige | Un seul flagelle polaire[5] | mésophile | Température : 10 - 40 °C[5] pH : 5 - 9[5] | Température : 23 - 26 °C[10] PH= 6,5[6] | 1,4[10] |
| Sulfurimonas denitrificans | Longueur variable avec largeur ~0.3 μm[6] | Rod court ou Spirilla-like | N / A | mésophile | Température : 10-30 °C[10] ; pH : 7[11] | Température : 22 °C[10], pH : 7[6] | 12[10] |
| Sulfurimonas gotlandica | 0,66 ± 0,083 x 62,1 ± 0,54 µm[12] | Tige incurvée ou Spirilla-like | Un flagelle polaire ou deux flagelles aux pôles opposés[12] | Psychrotolérant | Température : 4 – 20 °C[6], pH : 6,7–8,0[6] | Température : 15 °C[6], | 13[6] |
| Sulfurimonas paralvinellae | 1,50-2,50 m × 0,6-0,8 m[10] | Tige | flagelle en mouvement
1,5 à 2,5 m de long et 0,6 à 0,8 m de large[10] |
mésophile | Température : 4-35 °C[10], pH : 5,4-8,6 | Température : 30 °C[10], pH : 6,1[6] | 13-16[10] |
Histoire de la reconnaissance
« Auto » et « trophicos » sont dérivés des mots grecs « auto » signifiant soi et « trophicos » faisant référence à l'allaitement, à l'entretien ou à l'alimentation, ce qui indique son autotrophie[4]. L'abondance et la distribution des sous-groupes au sein des epsilonproteobacteria et du genre Sulfurimonas ont été détectés dans la colonne d'eau en utilisant un certain nombre de techniques, y compris le clonage de l'ARNr 16S, le dépôt catalysé de marqueurs et hybridation fluorescente in situ (CARD-FISH), et des mesures de PCR quantitative[13]. Des échantillons d'eau ont été prélevés à différentes profondeurs et les concentrations de nutriments, d'oxygène et de soufre mesurées immédiatement après l'échantillonnage. L'échantillon a été mesuré pour le taux de fixation du carbone, et l'ADN extrait et les séquences spécifiques amplifiées par PCR[13].
L'épithète « dénitrificans » dans le nom Sulfurimonas denitrificans (S. denitrificans) fait référence à sa capacité à réduire les nitrates en gaz diazote, un processus connu sous le nom de dénitrification. En 2006, Sulfurimonas denitrificans était la dernière espèce à être classée dans le genre Sulfurimonas, car en 2000 elle avait été classée à tort dans le genre Thiomicrospira[14].
Les études sur Sulfurimonas gotlandica (S. gotlandica) ont principalement été menées dans la mer Baltique, en utilisant la microscopie électronique à transmission et la microscopie à fluorescence avec de l'acide phosphotungstique et une coloration DAPI comme formes de visualisation[6] - [15] - [14].
Sulfurimonas paralvinellae a d'abord été récolté à partir d'un nid de vers polychètes des grands fonds, en particulier de la famille des Alvinellidés. Des membres du genre Paralvinellae ont été trouvés sur un monticule de sulfure d' un évent hydrothermal en eau profonde dans le champ nord d'Iheya au milieu de la dépression d'Okinawa[10]. La souche a été initialement séparée du nid par une technique de dilution à extinction. Elle a été référencée sous le nom de GO25 T et présentait des caractéristiques physiologiques et phylogénétiques similaires à celles de Sulfurimonas autotrophica. Il a été déterminé plus tard que cette espèce diffère de Sulfurimonas autotrophica par un métabolisme énergétique distinct[3] - [10].
Métabolisme
En général, les bactéries ont de nombreuses voies métaboliques, et les variations de cette caractéristique du genre Sulfurimonas, permet de les classer en taxons[16]. Les membres du genre Sulfurimonas vivent dans un large éventail d'environnements et jouent un rôle vital dans les processus chimiotrophes, en fonction de l'environnement[3]. Il a été démontré que les isolats des quatre espèces de ce genre croissent avec une grande variété d'accepteurs et de donneurs d'électrons, leur permettant de se développer dans une grande variété d'environnements[3]. Ces atouts sont sa capacité chimiolithotrophique, son métabolisme capable d'accepter des relations changeantes avec les anions et les cations, et des sources de carbone inorganique, sa tolérance à l'oxygène et sa capacité à changer avec l'environnement[3] - [17]. Avec des environnements différents, quatre types de métabolisme énergétique sont observés ; y compris le métabolisme du soufre, de l'hydrogène, de l'azote et du carbone[14].
Métabolisme du soufre
En tant qu'Epsilonproteobacterium oxydant le soufre, des études ont montré que Sulfurimonas spp. utilise une grande variété de donneurs d'électrons pour la croissance, notamment le sulfure, le soufre, le thiosulfate et le sulfite[3]. Cependant, comme indiqué ci-dessous, seule Sulfurimonas gotlandica peut profiter de toutes les sources d'électrons mentionnés (#tableau 2). Le soufre oxygénase réductase (SOR) catalyse le soufre, créant du sulfite, du thiosulfate et du sulfure[18]. Les gènes SOR se trouvent chez S. gotlandica et S. autotrophica, mais sont absents chez S. denitrificans[14]. On émet l'hypothèse que S. denitrificans a perdu ses gènes SOR en raison de l'habitat à faible teneur en sulfure dans la mer des Wadden néerlandaise[14].
Sulfurimonas paralvinellae est capable d'utiliser à la fois de l'hydrogène moléculaire et du soufre réduit pour le métabolisme, elle est la deuxième epsilonprotéobactérie des grands fonds découverte à le faire[10]. Sulfurimonas paralvinellae est également capable d'utiliser l'extrait de levure comme source de soufre[3]. On observe que l'hydrogène moléculaire donne un taux de croissance plus élevé et est favorisé par Sulfurimonas paralvinellae par rapport au soufre réduit libre, même si ce dernier est présent dans l'environnement en excès. Cela peut peut-être s'expliquer par la plus petite quantité d'hydrogène moléculaire nécessaire pour maintenir la croissance dans une cellule bactérienne par rapport au soufre réduit[10].
| Sulfurimonas autorophica | Sulfurimonas denitrificans | Sulfurimonas gotlandica | Sulfurimonas paralvinellae | |
|---|---|---|---|---|
| Sulfure | ✓ | ✓ | ✓ | X |
| Soufre | ✓ | ✓ | ✓ | ✓ |
| Thiosulfate | ✓ | ✓ | ✓ | ✓ |
| Sulfite | X | X | ✓ | X |
| Hydrogène | X | ✓ | ✓ | ✓ |
À l'exception de S. paralvinellae, toutes les espèces de Sulfurimonas peuvent utiliser le sulfure comme donneur d'électrons, en oxydant le sulfure en sulfate[3]. La voie d'oxydation du sulfure utilisée est appelée sulfure-quinone réductase (SQR) et est codée par des gènes impliqués dans la voie responsable de la catalyse de l'oxydation du sulfure[3] - [19]. Il existe six types connus de protéines SQR dans tous les règnes[3].
La plupart des isolats d'Epsilonproteobacteria ont des SQR classés comme types II, IV et VI, n'ont jamais de type I et ont parfois des types III et V[3]. Sulfurimonas est le seul genre qui a des SQR de type III et V[3]. Le type III ne se trouve que chez S. denitrificans et S. gotlandica[3]. Le SQR de type IV est hautement conservé et se trouve dans les quatre espèces de Sulfurimonas et est donc considéré comme le SQR le plus important pour la survie des cellules de ce genre[3]. Selon une étude portant sur l'expression hétérologue d'homologues SQR chez S.denitrificans, il possède trois SQR fonctionnels : les types II, III et IV[19]. Une autre étude a également révélé que le type VI des SQR ne se trouvait pas chez S. denitrificans, mais qu'il l'était chez les trois autres espèces[14]. Les chercheurs ont découvert que le SQR de type VI fonctionnait dans des environnements à haute teneur en sulfure[14] et ont émis l'hypothèse que S. denitrificans ne nécessite pas de SQR de type VI car les individus auraient une faible probabilité de rencontrer du sulfure libre[14]. D'autre part, S. autotrophica est la seule des quatre espèces à contenir des SQR de type V et a été associée à des organismes vivant dans des mines acides[5].
Les rôles exacts des SQR fonctionnels restent inconnus[19]. Cependant, en général, ils sont cruciaux pour l'oxydation, l'assimilation et la signalisation des sulfures, la production d'énergie et la tolérance aux métaux lourds[19]. Par conséquent, les membres du genre Sulfurimonas contribuent de manière significative au cycle global du soufre, car tous les membres de l'espèce oxydent le soufre en sulfate[5].
Métabolisme de l'hydrogène
Toutes les espèces de Sulfurimonas sauf une peuvent utiliser l'hydrogène comme source d'énergie pour se développer[14]. S. autotrophica est le seul élément de Sulfurimonas spp. à ne pas utiliser d'hydrogène, et il a été démontré qu'il se développe avec l'hydrogène dans des conditions aérobies et anaérobies[6]. En revanche, l'hydrogène est parfois utilisé comme donneur d'électrons par des bactéries appartenant à S. denitrificans, S. gotlandica et S. paralvinellae, au lieu de composés à base de soufre[6] - [20].
Pour catalyser le métabolisme de l'hydrogène, les bactéries appartenant au genre Sulfurimonas utilisent la [NiFe]-hydrogénase. La réaction étant H2 ↔2H+ + 2e−[21]. Il existe de nombreuses hydrogénases [NiFe] différentes classées en différents groupes (groupes I à IV) et se retrouvant dans les quatre espèces de Sulfurimonas[14]. Étant donné que S. autotrophica contient des hydrogénases, une étude a conclu que dans des conditions environnementales spécifiques, il peut être capable de consommer de l'hydrogène[5].
Une autre étude montre que S. denitrificans se développe plus efficacement avec l'hydrogène qu'avec le thiosulfate[14]. Les trois espèces de Sulfurimonas expriment une hydrogénase active d'absorption d'hydrogène et peuvent croître sur l'hydrogène[14]. L'endroit où vivent les bactéries (c'est-à-dire l'eau de mer, les sédiments ou les sources hydrothermales) affectera le niveau d'oxygène dans l'environnement et donc le type de métabolisme qu'elles utilisent[14].
Métabolisme de l'azote
Pour autant que nous le sachions, le métabolisme de l'azote se produit chez tous les membres appartenant aux espèces Sulfurimonas, à l' exception de S. autotrophica[14]. Il a été démontré que celle-ci ne pouvait pas croître à une concentration de 5 mmol/L (mM) de nitrate de sodium dans des conditions de laboratoire[4]. La dénitrification par Sulfurimonas spp. joue un rôle crucial dans le cycle de l'azote[14]. Le renouvellement des nitrates pour S. denitrificans se fait de 20 mM de trois jours (avec le thiosulfate et l'hydrogène) à six jours (thiosulfate et sans hydrogène)[14]. Le renouvellement des nitrates chez les deux autres espèces, S. gotlandica et S. paralvinellae, est assez différent[14]. S. gotlandica n'a utilisé que 1 mM de nitrate en 9 jours (avec du thiosulfate) et S. paralvinellae a utilisé 10 mM de nitrate en 4 jours (avec de l'hydrogène et du soufre)[7] - [10]. De plus, S. gotlandica et S. denitrificans peuvent utiliser du nitrite au lieu du nitrate comme accepteur d'électrons[14]. La liste des accepteurs d'électrons que les espèces Sulfurimonas peuvent utiliser est résumée dans le tableau 3.
| Sulfurimonas autorophica | Sulfurimonas denitrificans | Sulfurimonas gotlandica | Sulfurimonas paralvinellae | |
|---|---|---|---|---|
| Nitrate | X | ✓ | ✓ | ✓ |
| Nitrite | X | ✓ | ✓ | X |
| Oxygène | ✓ | ✓ | ✓ | ✓ |
Pour catalyser la réaction du métabolisme de l'azote, toutes les espèces de Sulfurimonas ont une sous-unité de nitrate réductase périplasmique (Nap), catalytique (NapA). Ces NapAs epsiolonprotéobactériens ont une affinité élevée pour les nitrates et peuvent représenter une adaptation des membres de ce genre aux faibles concentrations environnementales de nitrates dans les évents en eau profonde.
Les isolats de S. gotlandica et S. denitrificans sont bien adaptés aux fluctuations des concentrations d'oxygène et d'hydrogène sulfuré dans l'environnement car ils peuvent tous deux utiliser du nitrate, du nitrite ou de l'oxygène comme accepteurs d'électrons. Leur capacité à utiliser le nitrate ou le nitrite comme accepteur d'électrons, au lieu de l'oxygène, étend leur niche écologique au-delà de l'anoxie à des zones beaucoup plus profondes de la colonne d'eau.
Spéciation
Les espèces qui constituent le genre Sulfurimonas se différencient par leurs différences physiologiques et génotypiques distinctes[6]. De nombreux isolats ont été partiellement ou complètement séquencés. Par exemple, le génome d'un isolat de S. autotrophica compte environ 2 153 198 paires de bases[5].
Les membres de ce genre bactérien habitent les eaux sulfurées, les zones rédox pélagiques et les évents en eau profonde, où les composés contenant du soufre sont abondants[2] - [3] - [4]. Les espèces du genre Sulfurimonas utilisent une variété de protéines qui catalysent des composés spécifiques trouvés dans ces zones, ce qui met en évidence les points communs et les différences dans leurs génomes d'ADN[2] - [3] - [4] - [6] - [8].
Les similitudes dans les séquences du gène de l'ARNr 16S entre Sulfurimonas gotlandica, Sulfurimonas paralvinellae, Sulfurimonas autotrophica et Sulfurimonas dentrificans sont supérieures à 90 %[6], avec S. gotlandica partage une similitude de 93,7 à 94,2 % avec les autres espèces[6]. De même, S. paralvinellae et S. autotrophica (souche OK10) ont une similarité de séquence de 96,3 % dans leurs séquences de gènes d'ARNr 16S[5] - [10], tandis que S. dentrificans et S. autotrophica (OK10) ont une similarité de séquence de 93,5 %[5].
La teneur en G + C de certains génomes de l'espèce est similaire. S. gotlandica contient une teneur en ADN G + C de 33,6 % molaire (A + T, 66,4 mol %)[6], tandis que S. paralvinellae a 37,6 % en moles de teneur en ADN G + C (63,4 % molaire A + T)[10].
L'analyse phylogénétique a montré que Sulfurimonas présente une capacité de dispersion limitée dans la mesure où la spéciation est plus affectée par la situation géographique que par la composition hydrothermale[8]. De plus, les courants océaniques profonds peuvent affecter la spéciation[8].
Le tableau suivant résume les informations fournies ci-dessus :
| Teneur en ADN G + C (mol %) | Teneur en ADN A + T (% mol) | Similitude des gènes de l'ARNr 16s (%) | |
|---|---|---|---|
| Sulfurimonas autotrophica | N / A | N / A | 96,3 % de similarité avec S. paralvinellae.
93,7-94,2 % de similarité avec S. gotlandica. 93,5 % de similarité avec S. dentrificans. |
| Sulfurimonas dentrificateurs | N / A | N / A | 93,5 % de similarité avec S. autrotrophica (OK10).
93,7-94,2 % de similarité avec S. gotlandica. |
| Sulfurimonas gotlandica | 33,6 % molaire | 66,4 % molaire | 93,7 - 94,2 % de similarité avec toutes les espèces. |
| Sulfurimonas paralvinellae | 37,6 % molaire | 63,4 % molaire | 96,3 % de similarité avec S. autrotrophica (OK10).
93,7-94,2 % de similarité avec S. gotlandica. |
Habitat
Les Sulfurimonas se trouvent couramment dans les habitats (sulfidogéniques), tels que les sédiments marins, les cheminées hydrothermales des grands fonds, les redoxclines pélagiques et les champs pétrolifères[22]. Les habitats où ils se trouvent se reflètent dans leur contenu génétique ; certains membres ont moins de gènes tandis que d'autres ont plus de gènes liés aux environnements où ils se produisent[3]. Certains de ces gènes permettent l'utilisation de différents donneurs et accepteurs d'électrons, leur permettant d'habiter une gamme particulière d'environnements[3]. Dans les sources hydrothermales des grands fonds, l'oxydation des sulfures est la source d'énergie chimique la plus importante pour Sulfurimonas spp[10]. Par coïncidence, des concentrations élevées de sulfure d'hydrogène au niveau des évents en eau profonde sont produites par des interactions entre l'eau de mer et la roche à haute température[10]. Il est à noter que les micro-organismes vivant dans l'océan profond et sombre oxydent les composés soufrés pour la chimiolithoautotrophie ; ce processus se produit par médiation microbienne[10]. Par exemple, les sulfures quinones réductases (SQR), trouvées dans tous les isolats de Sulfurimonas spp. aide à l'oxydation des composés contenant du soufre et du thiosulfate[4] - [3] - [19]. De plus, les hydrogénases et d'autres enzymes permettent à ce genre particulier de coloniser des environnements « disparates »[3]. En raison de la dépendance vis-à-vis des composés soufrés en haute mer, cela pourrait créer une concurrence entre les espèces.
Des biofilms se trouvent à environ 100 m de profondeur d'eau à côté de cheminées hydrothermales actives[12]. L'analyse phylogénétique a montré que ces tapis bactériens étaient constitués de bactéries du genre Sulfurimonas[12]. Des biofilms similaires se trouvent couramment sur les monts sous-marins créés par les volcans sous-marins et par le volcanisme intense des cheminées hydrothermales[22]. La ventilation hydrothermale favorise la croissance de tapis bactériens que Sulfurimonas colonise, comme sur les monts sous-marins Lo'ihi, Axial, Vailulu'u Suiyo, et les arcs des Mariannes et de Kermadec[22].
Interactions biotiques
Prédation
Sulfurimonas (sous-groupe GD17) domine la dénitrification chimiotrophe dans les redoxclines pélagiques de la mer Baltique[6] - [15] - [14]. En utilisant des méthodes telles que les tests de prédateurs et les cultures d'amendement bactérien, il a été constaté que la population de Sulfurimonas (sous-groupe GD17) avait un temps de doublement de 1 à 1,5 par jour, ce qui est bien plus que leur temps de doublement moyen dans les conditions optimales indiquées dans le tableau 1[23]. Cependant, le broutage peut consommer la population au cours d'une journée[23]. Cinq brouteurs actifs que l'on trouve généralement dans les ciliés redoxclines (Oligohymenophorea, Prostomatea) et les groupes de flagellés marins (MAST-4, Chrysophyta, Cercozoa) ont été trouvés grâce à l'utilisation de l'ARN-SIP[23]. Dans l'écosystème d'infiltration froide, les crabes lithodidés (Paralomis sp.), qui sont des mangeurs de filtres, se nourrissent de tapis bactériens thiotropes, constitués de nombreux types de bactéries différentes[23].
Il a été constaté qu'en l'absence d'autres prédateurs possibles, les bactéries appartenant au genre Sulfurimonas se développent dans une relation unimodale, suggérant qu'elles augmentent la diversité et la productivité bactériennes. Cela signifie que sans prédateurs, ces bactéries peuvent se différencier et croître de façon exponentielle[23].
Symbiose
Sulfurimonas est un genre que l'on observe couramment en symbiose avec d'autres organismes, principalement des vers annelés marins. Sulfurimonas paralvinellae est associé à des colonies de polychètes d'eau profonde situées à proximité de sources hydrothermales. Les nids de ces vers sont recouverts de particules de soufre réduit produites par une combinaison de processus microbiologiques et chimiques. Ces particules de soufre servent de source d'énergie facilement disponible pour les bactéries appartenant au genre Sulfurimonas[10].
La bioturbation par les vers arénicoles dans des environnements d'eau peu profonde en l'absence d'oxygène améliore le métabolisme des bactéries du phylotype Epsilonproteobacterium, qui montre plus de 95 % de similitude avec Sulfurimonas denitrificans. Les activités des vers telles que l'enfouissement et le pompage péristaltique font que l'oxygène et le dioxyde de carbone dissous dans l'eau pénètrent dans les sédiments. Cette couche enrichie descend et se mélange aux sédiments sous-jacents. L'oxygène extrait de l'eau pénètre dans cette couche anaérobie et crée du sulfate métabolisable par les bactéries. Le sulfure de l'eau peut également être détoxifié par ces vers soit par une chaîne respiratoire ramifiée, soit par une régulation redox de l'oxydation du sulfure. Le produit final de ces réactions est le thiosulfate, qui devient une autre source d'énergie pour les bactéries chimiotrophes colonisant le sédiment[24].
Compétition
Les bactéries appartenant au genre Sulfurimonas sont en compétition avec d'autres bactéries sulfato-oxydantes (SOB) pour les ressources nutritionnelles, et ont fait l'objet d'études intensives en raison de leur importance dans l'industrie pétrolière[24] - [25]. Les communautés SOB constituent des membres physiologiquement divers d'Epsilonproteobacteria, qui contient les genres Sulfurimonas, Chlorobia et Chloroflexi[25]. Tous ces genres se trouvent dans des réservoirs pétroliers et les Sulfurimonas y sont présents en abondance[24] - [25]. Les membres de ce genre occupaient environ 26 % des réservoirs, qui diffèrent tous par la température et les proportions relatives des autres SOB, ce qui indique en outre que ce genre est capable de croître dans une large gamme de températures[3] - [15] - [24] - [25]. Malgré cela, Sulfurimonas est en concurrence avec Thioclava, Sulfuricurvum et Thiohalomonas, qui sont en corrélation avec respectivement 15,4 %, 12,0 % et 17,0 %[25]. La majorité des bactéries présentes dans ces réservoirs sont des bactéries non cultivées, qui n'ont pas été étudiées en profondeur[24] - [25]. Les bactéries des genres susmentionnés tirent leur énergie de l'oxydation de composés soufrés réduits (c'est-à-dire sulfure et thiosulfate), ce qui suggère une concurrence directe entre elles pour les composés contenant du soufre[3] - [4] - [6].
Chaîne alimentaire
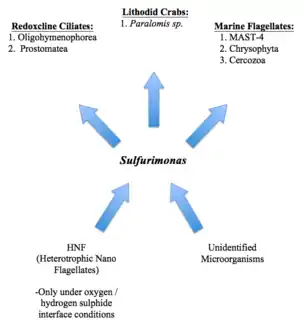
Les membres du genre bactérien Sulfurimonas sont connus pour affecter l'abondance relative des espèces qui les entourent[26]. Dans le cas de la souche GD1 de S. gotlandica, il a été démontré que les populations de nanoflagellés hétérotrophes (HNF) diminuaient tandis que les colonies de ciliés et de dinoflagellés restaient relativement constantes dans des conditions riches en oxygène/sulfure d'hydrogène[26]. À l'inverse, la tendance inverse a été observée dans des conditions suboxiques, en ce sens que le HNF et les ciliés ont augmenté en nombre, tandis que les populations de dinoflagellés sont restées constants[26]. Ces changements ont été attribués à la fois à la présence d'oxygène/sulfure d'hydrogène dans l'environnement et à la prédation potentielle de HNF par S. gotlandica[26].
Dans une autre étude, on a découvert que cinq brouteurs actifs résident dans des conditions redoxclines, à savoir les ciliés redoxclines, les groupes de flagellés marins et certaines espèces de l'écosystème à suintements froids[23] - [27]. En fait, ces organismes sont capables de consommer toute la population de Sulfurimonas dans cette zone particulière en une journée[23].
Pertinence environnementale
Pour se développer, les espèces Sulfurimonas consomment à la fois des espèces soufrées réduites et des espèces azotées oxydées. Par conséquent, les membres de Sulfurimonas affectent directement le cycle biogéochimique de ces éléments dans les environnements où ils existent et sont numériquement abondants. Par exemple, Sulfurimonas gotlandica peut représenter jusqu'à 25 % de la communauté microbienne dans le redoxcline de la mer Baltique sur la base des dénombrements cellulaires CARD-FISH[15] et Sulfurimonas spp. représentaient un nombre important de relevés d'Epsilonproteobacteries dans une étude de la diversité microbienne au niveau des évents d'écoulement diffus en haute mer à l'aide de séquences 16S amplifiées.
De plus amples recherches
Sulfurimonas hongkongensis est une espèce nouvellement découverte au sein du genre Sulfurimonas. Il a été trouvé à Hong Kong, près des sédiments côtiers du canal d'approche Kai Tak reliant le port de Victoria. Il produit de l'énergie dans des conditions anoxiques. Son donneur d'électrons est le thiosulfate, le sulfure ou l'hydrogène, et son accepteur d'électrons est le nitrate. Sa morphologie est en forme de tige, et il se développe de 15 à 35 °C (optimal à 30 °C), au pH de 6,5 à 8,5 (optimal entre 7,0 et 7,5) et 10 à 60 g L−1de NaCl (optimal à 30 g L−1). Son génome se compose de 34,9 % de GC, de 2 290 gènes codant des protéines et de 42 gènes d'ARN (3 gènes d'ARNr). Ses principaux acides gras cellulaires étaient C14:0 (4,8 %), C16:0 (32,8 %), 2-OH C16:0 (9,5 %), C16:1 (14,6 %), C18:0 (16,9 %) et C18:1 (19,2 %). La composition de ces acides gras est similaire à celles trouvées dans les isolats de Sulfurimonas paralvinellae et Sulfurimonas autotrophica, mais il existe un acide gras unique, représenté par 2-OH C16:0 qui définit qu'il s'agit d'une espèce différente des autres membres de la genre Sulfurimonas.
Références
- (en) F. Inagaki, K. Takai, H. Kobayashi, K. H. Nealson et K. Horikoshi, « Sulfurimonas autotrophica gen. nov., sp. nov., a novel sulfur-oxidizing -proteobacterium isolated from hydrothermal sediments in the Mid-Okinawa Trough », INTERNATIONAL JOURNAL OF SYSTEMATIC AND EVOLUTIONARY MICROBIOLOGY, vol. 53, no 6, , p. 1801–1805 (ISSN 1466-5026 et 1466-5034, DOI 10.1099/ijs.0.02682-0, lire en ligne, consulté le ).
- (en) Zena Cardman, « Active prokaryotic communities along a thermally and geochemically variable transect in Guaymas Basin hydrothermal sediments », ProQuest Dissertations Publishing, .
- (en) Yuchen Han et Mirjam Perner, « The globally widespread genus Sulfurimonas: versatile energy metabolisms and adaptations to redox clines », Frontiers in Microbiology, vol. 6, , p. 989 (PMID 26441918, PMCID 4584964, DOI 10.3389/fmicb.2015.00989).
- (en) Fumio Inagaki, Ken Takai, Hideki Kobayashi et Kenneth H. Nealson, « Sulfurimonas autotrophica gen. nov., sp. nov., a novel sulfur-oxidizing ε-proteobacterium isolated from hydrothermal sediments in the Mid-Okinawa Trough », International Journal of Systematic and Evolutionary Microbiology, vol. 53, no 6, , p. 1801–1805 (PMID 14657107, DOI 10.1099/ijs.0.02682-0).
- (en) Johannes Sikorski, Christine Munk, Alla Lapidus et Olivier Duplex Ngatchou Djao, « Complete genome sequence of Sulfurimonas autotrophica type strain (OK10T) », Standards in Genomic Sciences, vol. 3, no 2, , p. 194–202 (ISSN 1944-3277, PMID 21304749, PMCID 3035374, DOI 10.4056/sigs.1173118).
- (en) Matthias Labrenz, Jana Grote, Kerstin Mammitzsch et Henricus T. S. Boschker, « Sulfurimonas gotlandica sp. nov., a chemoautotrophic and psychrotolerant epsilonproteobacterium isolated from a pelagic redoxcline, and an emended description of the genus Sulfurimonas », International Journal of Systematic and Evolutionary Microbiology, vol. 63, no 11, , p. 4141–4148 (PMID 23749282, PMCID 3836495, DOI 10.1099/ijs.0.048827-0).
- (en) Kerstin Mammitzsch, Günter Jost et Klaus Jürgens, « Impact of dissolved inorganic carbon concentrations and pH on growth of the chemolithoautotrophic epsilonproteobacterium Sulfurimonas gotlandica GD1T », MicrobiologyOpen, vol. 3, no 1, , p. 80–88 (ISSN 2045-8827, PMID 24376054, PMCID 3937731, DOI 10.1002/mbo3.153).
- (en) Sayaka Mino, Satoshi Nakagawa, Hiroko Makita et Tomohiro Toki, « Endemicity of the cosmopolitan mesophilic chemolithoautotroph Sulfurimonas at deep-sea hydrothermal vents », The ISME Journal, vol. 11, no 4, , p. 909–919 (PMID 28045457, PMCID 5364360, DOI 10.1038/ismej.2016.178, résumé).
- La parenté des isolats peut également être analysée avec MultiLocus Sequence Analysis (MLSA) Multilocus sequence typing (en) qui n'utilise pas les allèles attribués, mais concatène à la place les séquences des fragments de gènes des gènes de ménage et utilise cette séquence concaténée pour déterminer les relations phylogénétiques. Contrairement à MLST, cette analyse attribue une similitude plus élevée entre les séquences ne différant que d'un seul nucléotide et une similitude plus faible entre les séquences avec de multiples différences de nucléotides. En conséquence, cette analyse est plus adaptée aux organismes à évolution clonale et moins adaptée aux organismes dans lesquels des événements de recombinaison se produisent très souvent. Il peut également être utilisé pour déterminer les relations phylogénétiques entre des espèces étroitement apparentées. Les termes MLST et MLSA sont très souvent considérés comme interchangeables, ce qui n'est cependant pas correct car chaque méthode d'analyse a ses particularités et ses utilisations.
- (en) Ken Takai, Masae Suzuki, Satoshi Nakagawa et Masayuki Miyazaki, « Sulfurimonas paralvinellae sp. nov., a novel mesophilic, hydrogen- and sulfur-oxidizing chemolithoautotroph within the Epsilonproteobacteria isolated from a deep-sea hydrothermal vent polychaete nest, reclassification of Thiomicrospira denitrificans as Sulfurimonas denitrificans comb. nov. and emended description of the genus Sulfurimonas », International Journal of Systematic and Evolutionary Microbiology, vol. 56, no 8, , p. 1725–1733 (PMID 16901999, DOI 10.1099/ijs.0.64255-0).
- (en) HOLGER W JANNASCH, CARL O. WIRSEN, DOUGLAS C. NELSON et LESLEY A. ROBERTSON, « Thiomicrospira crunogena sp. nov., a Colorless, Sulfur-Oxidizing Bacterium from a Deep-sea Hydrothermal Vent », International Journal of Systematic Bacteriology, vol. 35, no 4, , p. 422–424 (DOI 10.1099/00207713-35-4-422).
- (en) Brennan Phillips, Matthew Dunbabin, Brad Henning et Corey Howell, « Exploring the "Sharkcano": Biogeochemical Observations of the Kavachi Submarine Volcano (Solomon Islands) », Oceanography, vol. 29, no 4, , p. 160–169 (DOI 10.5670/oceanog.2016.85, lire en ligne).
- (en) Jana Grote, Matthias Labrenz, Birgit Pfeiffer et Günter Jost, « Quantitative distributions of Epsilonproteobacteria and a Sulfurimonas subgroup in pelagic redoxclines of the central Baltic Sea », Applied and Environmental Microbiology, vol. 73, no 22, , p. 7155–7161 (ISSN 0099-2240, PMID 17921285, PMCID 2168200, DOI 10.1128/AEM.00466-07, Bibcode 2007ApEnM..73.7155G).
- (en) Yuchen Han et Mirjam Perner, « The Role of Hydrogen for Sulfurimonas denitrificans' Metabolism », PLOS ONE, vol. 9, no 8, , e106218 (PMID 25170905, PMCID 4149538, DOI 10.1371/journal.pone.0106218, Bibcode 2014PLoSO...9j6218H).
- (en) Jana Grote, Thomas Schott, Christian G. Bruckner et Frank Oliver Glöckner, « Genome and physiology of a model Epsilonproteobacterium responsible for sulfide detoxification in marine oxygen depletion zones », Proceedings of the National Academy of Sciences, vol. 109, no 2, , p. 506–510 (ISSN 0027-8424, PMID 22203982, PMCID 3258601, DOI 10.1073/pnas.1111262109, Bibcode 2012PNAS..109..506G).
- (en) Jianping Xu, « INVITED REVIEW: Microbial ecology in the age of genomics and metagenomics: concepts, tools, and recent advances », Molecular Ecology, vol. 15, no 7, , p. 1713–1731 (ISSN 1365-294X, PMID 16689892, DOI 10.1111/j.1365-294X.2006.02882.x).
- (en) Barbara J. Campbell, Annette Summers Engel, Megan L. Porter et Ken Takai, « The versatile epsilon-proteobacteria: key players in sulphidic habitats », Nature Reviews. Microbiology, vol. 4, no 6, , p. 458–468 (ISSN 1740-1526, PMID 16652138, DOI 10.1038/nrmicro1414).
- (en) Claudia Janosch, « Sulfur Oxygenase Reductase (Sor) in the Moderately Thermoacidophilic Leaching Bacteria: Studies in Sulfobacillus thermosulfidooxidans and Acidithiobacillus caldus », Microorganisms, vol. 3, no 4, , p. 707–724 (PMID 27682113, PMCID 5023260, DOI 10.3390/microorganisms3040707).
- (en) Yuchen Han et Mirjam Perner, « Sulfide consumption in Sulfurimonas denitrificans and heterologous expression of its three SQR homologs », American Society for Microbiology, vol. 198, no 8, , p. 1260–1267 (PMID 26833414, PMCID 4859588, DOI 10.1128/JB.01021-15).
- (en) D. Gevertz, A. J. Telang, G. Voordouw et G. E. Jenneman, « Isolation and characterization of strains CVO and FWKO B, two novel nitrate-reducing, sulfide-oxidizing bacteria isolated from oil field brine », Applied and Environmental Microbiology, vol. 66, no 6, , p. 2491–2501 (ISSN 0099-2240, PMID 10831429, PMCID 110567, DOI 10.1128/aem.66.6.2491-2501.2000, Bibcode 2000ApEnM..66.2491G).
- (en) Paulette M. Vignais et Bernard Billoud, « Occurrence, classification, and biological function of hydrogenases: an overview », Chemical Reviews, vol. 107, no 10, , p. 4206–4272 (ISSN 0009-2665, PMID 17927159, DOI 10.1021/cr050196r).
- (en) David Emerson et Craig L. Moyer, Microbiology of seamounts, Rockville, USA, Oceanography, , 151 p..
- (en) Ruth Anderson, Claudia Wylezich, Sabine Glaubitz et Matthias Labrenz, « Impact of protist grazing on a key bacterial group for biogeochemical cycling in Baltic Sea pelagic oxic/anoxic interfaces », Environmental Microbiology, vol. 15, no 5, , p. 1580–1594 (ISSN 1462-2920, PMID 23368413, DOI 10.1111/1462-2920.12078).
- (en) Wriddhiman Ghosh et Bomba Dam, « Biochemistry and molecular biology of lithotrophic sulfur oxidation by taxonomically and ecologically diverse bacteria and archaea », FEMS Microbiology Reviews, vol. 33, no 6, , p. 999–1043 (ISSN 1574-6976, PMID 19645821, DOI 10.1111/j.1574-6976.2009.00187.x).
- (en) Huimei Tian, Peike Gao, Zhaohui Chen et Yanshu Li, « Compositions and Abundances of Sulfate-Reducing and Sulfur-Oxidizing Microorganisms in Water-Flooded Petroleum Reservoirs with Different Temperatures in China », Frontiers in Microbiology, vol. 8, , p. 143 (ISSN 1664-302X, PMID 28210252, PMCID 5288354, DOI 10.3389/fmicb.2017.00143).
- (en) « Changes during the predator exclusion experiments with... - Figure 3 of 6 », sur ResearchGate (consulté le ).
- (en) Helge Niemann, Peter Linke, Katrin Knittel et Enrique MacPherson, « Methane-Carbon Flow into the Benthic Food Web at Cold Seeps – A Case Study from the Costa Rica Subduction Zone », PLOS ONE, vol. 8, no 10, , e74894 (ISSN 1932-6203, PMID 24116017, PMCID 3792092, DOI 10.1371/journal.pone.0074894, Bibcode 2013PLoSO...874894N).