Hydrogénase
Les hydrogénases sont des enzymes qui catalysent de façon réversible la conversion des ions H+ (« protons ») en dihydrogène selon la réaction :
- 2H+ + 2e– = H2.
Les sites actifs de ces enzymes sont de nature organométallique et diffèrent entre eux notamment par la nature des métaux qui les composent. Il existe ainsi trois classes d'hydrogénases : les hydrogénases [NiFe], les hydrogénases à fer seul [FeFe] et les hydrogénases précédemment appelées sans-métal, mais qui contiennent en fait un fer.
Généralités
En 1887, Hoppe-Seyler découvre que des bactéries peuvent décomposer le formiate en H2 et CO2. Le nom « hydrogénase » est donné par Stevenson et Stickland en 1931 après avoir observé la production d’hydrogène par des bactéries du colon et son utilisation pour réduire des substrats. Les hydrogénases désignent maintenant une classe d’enzyme qui peut catalyser de façon réversible la conversion des protons en hydrogène :
Elles catalysent cette réaction à un potentiel très proche du potentiel thermodynamique (E°app = −413 mV dans l’eau, à 25 °C, sous 0,1 bar de H2 et pH 7). Dans ces organismes, l’hydrogène peut avoir deux fonctions.
La première fonction est énergétique : un excédent de pouvoir réducteur peut être éliminé sous forme d’hydrogène.
La seconde fonction métabolique utilise l’hydrogène comme substrat réducteur : réduction du CO2 en méthane chez Methanobacterium, en acide éthanoïque chez Acetobacterium, hydrogénation du fumarate chez Vibrio succinogenes, production de NAD(P)H dans les hydrogénases diaphorases comme chez Ralstonia eutropha, etc.
Si les hydrogénases sont connues depuis plus d’un siècle, la détermination de leurs structures et plus particulièrement celle de leurs sites actifs a suivi un long cheminement :
En 1956, la présence de fer non hémique est confirmée. Au cours des années 1970, des expériences de RPE ont montré que les hydrogénases contenait des clusters fer-soufre de type ferrédoxine HiPIP (« high potential iron-sulfur protein »). En 1980, Thauer a détecté la présence de nickel dans certaines hydrogénases donnant lieu à de nombreuses spéculations sur la nature du site actif. Il est maintenant établi qu’il existe deux principales classes d’hydrogénases, les hydrogénases [NiFe] et les hydrogénases à fer, ainsi qu’une classe apparentée appelée hydrogénases sans cluster fer-soufre ou Hmd. On connaît une centaine d’hydrogénases réparties chez une quarantaine d’organismes.
Classes
Les deux principales classes d'hydrogénases se distinguent par leurs activités ainsi que par leurs sites actifs très originaux. En effet, la présence de sites organométalliques possédant des ligands cyanure et carbonyle est un fait unique en biologie. Cela est d’autant plus remarquable que ces molécules prosthétiques sont généralement considérées comme toxiques pour les organismes vivants.
Hydrogénases à fer
Elles ne sont présentes que chez les eubactéries et les eucaryotes. Les structures 3D des Fe-hydrogénases fonctionnelles ont été résolues presque simultanément chez deux bactéries anaérobies : D. desulfuricans[1] et C. pasteurianum[2]. Les structures de ces enzymes sont assez différentes mais elles possèdent toutes deux des clusters [Fe-S] capables de drainer des électrons entre la surface et le site actif profondément enfoui dans la protéine. Le site actif appelé cluster-H est composé d'un cluster [4Fe-4S] lié via une cystéine pontante à un cluster dinucléaire à fer (cf. Fig. 2). Les atomes de fer de ce cluster dinucléaire possèdent des ligands cyanures et carbonyles et sont reliés par un ligand prosthétique pontant de type [1,3-propanedithiolate]. L’analyse des liaisons hydrogène entre ce ligand et la cavité ainsi que l’intuition du cristallographe peuvent laisser penser que l’atome central serait un atome d’azote (le ligand pontant serait alors la dithiométhylamine). La nature de l'atome central est encore controversée; des résultats récents suggèrent qu'il pourrait s'agir d'un oxygène (le ligand pontant serait le dithiométhyléther)[3].
Des hydrogénases à Fe fonctionnellement productrices d'H2 ne sont actuellement trouvées que chez les organismes de métabolisme anaérobie. Par contre, les recherches systématiques dans les banques de données ont mis en évidence la présence de gènes codant des protéines homologues chez les eucaryotes de métabolisme aérobie (champignons, plantes et animaux)[4]. La fonction de ces protéines chez les eucaryotes aérobies n'est pas encore complètement élucidée, mais on peut exclure une activité de type hydrogénase, aucune production d'H2 n'ayant été observée chez les eucaryotes aérobies. Ce sont des protéines liant des clusters Fe/S, en tout cas chez la levure et les végétaux[5] - [6]. Chez Saccharomyces cerevisiae, la protéine ScNar1p serait impliquée dans la maturation des protéines cytosoliques et nucléaires à Cluster Fe/S[5]. Chez l'homme, il existe deux gènes codant des Hydrogénases like. Le premier gène code une protéine nommée IOP1, impliquée dans la régulation de la dégradation de HIF1[7] et la maturation des protéines à cluster Fs/S chez l'homme[8]. Le deuxième gène code une protéine nommée IOP2, anciennement NARF qui reconnait l'extrémité farnésylée et non maturée de la prélamine A[9]. Chez les plantes, un seul gène est présent et l'inactivation de ce gène chez Arabidopsis thaliana montre que ce gène est impliqué dans le développement. La lignée mutante montre un phénotype nain qui est reversé lorsque la lignée pousse à 5 % O2[6]. Cette relation avec l'O2 a aussi été montrée chez Caenorhabditis elegans et la levure où la croissance en milieu hypoxique modérée reverse les phénotypes mutants observées.
Hydrogénases [NiFe] et [NiFeSe]
Les hydrogénases [NiFe] sont les plus nombreuses des hydrogénases. Elles sont présentes chez de nombreux micro-organismes comme les bactéries acétogènes, fixatrices d’azote, sulfato-réductrices des cyanobactéries (algues bleues) mais aussi chez des archées. Il existe en revanche peu d’hydrogénases [NiFeSe]. On peut classer ces deux types d’hydrogénases en quatre grands groupes d’après leurs fonctions cellulaires. La comparaison des alignements de séquences et plus particulièrement de deux régions conservées (autour des cystéines du site actif et près des zones N- et C-terminales) permet de retrouver ces quatre classes. Le groupe 1 se compose des hydrogénases [NiFe] associées aux membranes et qui se servent de l’hydrogène comme source d’énergie. Le deuxième groupe est composé d’hydrogénases cytoplasmiques hétérodimériques et sont absentes chez les archées. Le troisième groupe est composé d’hydrogénases cytoplasmiques hétéromultimériques pouvant lier un cofacteur (F420, NAD ou NADP) et fonctionnent de façon réversible. Le quatrième et dernier groupe regroupe les hydrogénases associées aux membranes qui produisent de l’hydrogène. Elles sont entre 50 et 100 fois moins actives que les hydrogénases à fer en production et oxydation de l’hydrogène (jusqu’à 1 000 cycles catalytiques par seconde).
Les hydrogénases [NiFe] sont caractérisées par un site actif hétérobimétallique contenant un atome de fer et un atome de nickel. Des structures cristallographiques de ces enzymes sont connues pour cinq organismes et sont de deux types: les hydrogénases [NiFeSe] et les hydrogénases [NiFe] à ligands carbonyles. Les hydrogénases [NiFeSe] sont présentes chez les bactéries sulfato-réductrices et peuvent être péri- ou cyto-plasmique.
Hydrogénases [NiFe]
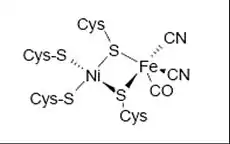
Ce sont les premières hydrogénases dont les structures cristallographiques ont été connues. Ce sont aussi les plus étudiées. L'hydrogénase [NiFe] de Desulvibrio (D.) gigas[10], est une protéine périplasmique hétérodimère, composée de deux sous-unités de 60 et 28 kDa. Elle possède trois clusters [Fe-S]: un cluster [3Fe-4S]1+/0 et deux clusters [4Fe-4S]2+/1+. Ils sont répartis de façon presque linéaire dans la petite sous-unité sur une longueur de 12 Å. Le cluster le plus proche du site actif se situe à 13 Å du site actif et est appelé cluster proximal ou cluster-p. Le cluster le plus éloigné du site actif (cluster distal ou cluster-d) se situe presque à la surface de la protéine. Ces clusters forment donc un « chemin » pour les électrons, entre un donneur extraprotéique et le site actif. Les hydrogénases [NiFe] posséderaient un atome de magnésium dans le domaine C-terminal. D’autre part, un canal hydrophobe[11] permet la circulation d’hydrogène de l’extérieur jusqu’au site actif et plus précisément près d’un site terminal vacant du nickel. Le site actif comprend un atome de nickel coordiné par quatre cystéinates formant un tétraèdre fortement distordu (cf. Fig. 4 et Fig. 6). Deux de ces cystéinates coordinent aussi un atome de fer. Cet atome de fer possède également deux ligands cyanures et un ligand carbonyle. Les ligands cyanures sont en interaction hydrogène avec les chaînes latérales de résidus de la cavité. Un site de coordination sur le fer est donc vacant et pour compléter la sphère de coordination du fer, un autre ligand peut être présent. De récents résultats cristallographiques suggèrent la présence d’un ligand oxygéné hydroxyde ou hydroperoxyde, pontant entre le nickel et le fer lorsque la protéine est oxydée. D’autres résultats cristallographiques et électrochimiques suggèrent que ce ligand pourrait aussi être un sulfure.
Hydrogénases [NiFeSe]
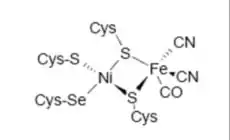
Les hydrogénases [NiFeSe] forment une sous-classe des hydrogénases [NiFe]. Elles contiennent un atome de sélénium par protéine. L'hydrogénase périplasmique de D.baculatum a été cristallisée. Il s'agit d'un hétérodimère comprenant deux sous unités de 26 et 49 kDa. Trois clusters [4Fe-4S] sont situés dans la petite sous unité, alors que la grosse sous unité ne contient « que » le site actif. Il ressemble beaucoup à celui des hydrogénases [NiFe]. L’originalité principale de cette enzyme est le remplacement d'une cystéine terminale par une sélénocystéine ligand du site actif. Les sélénocystéinates pourraient plus facilement se protoner que les cystéinates. Cela expliquerait les différences de réactivité. Dans le site actif de l'hydrogénase cristallisée, la distance entre le nickel et le fer est de 2,5 Å. Les hydrogénases [NiFe] posséderaient un atome de magnésium dans le domaine C-terminal. Pour les hydrogénases [NiFeSe], cet atome serait remplacé par un atome de fer.
Hydrogénases sans cluster fer soufre
Ce ne sont pas à proprement parler des hydrogénases car elles n'utilisent pas de protons pour produire de l'hydrogène. Cependant, les nombreuses similitudes avec les hydrogénases lui confèrent cette dénomination. Longtemps appelées « hydrogénases sans métal » (metal free hydrogenases), elles furent renommées en 2000 « hydrogénases sans cluster [Fe-S] » à la suite de la découverte d'un motif à fer au sein du cofacteur de cette enzyme. Ce fer est non-hémique et possède deux ligands carbonyle[12]. Le cofacteur s’inactive à la lumière en perdant le motif fer carbonyle qui lui est lié[13].
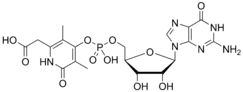
Elles sont aussi connues sous le nom de « Hmd » pour H-2-forming methylentetrahydromethanopterin dehydrogenase. Seules certaines archées méthanogènes en possèdent. Ces hydrogénases différent largement des deux autres car elles ne contiennent pas de clusters [Fe-S] et ne possèdent pas la même réactivité. En effet, elles catalysent réversiblement la réaction de réduction du N5,N10-méthényltétrahydrométhanopterine en N5,N10-méthénylénetétrahydrométhanopterine en présence d'hydrogène. Cela correspond à un transfert d'hydrure sur le substrat et à la libération d'un proton en solution. Ces hydrogénases ne sont pas capables de catalyser la réduction du méthylviologène par H2, ni de catalyser l'échange des protons de H2 avec le deutérium de l'eau lourde. C'est pour cela que ce ne sont pas des hydrogénases à proprement parler. Cependant, leur capacité à utiliser l'hydrogène leur valent l'appellation « hydrogénase ».
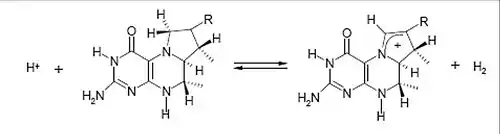
L’activité de ces enzymes dépend fortement du pH. Un pH basique favorise la formation de méthényl-H4MPT+, alors qu’un pH acide et la présence d’hydrogène favorise la formation de méthylène-H4MPT. Par exemple, la réaction de déshydrogénation peut se faire avec une vitesse maximale de 2 700 μmol/min par mg de protéine pour Methanopyrus kandleri à pH inférieur à 4,5 à plus de 90 °C.
Sources
- (en) M. W. W. Adams et E. I. Stiefel, « Biological Hydrogen Production: Not So Elementary », Science, vol. 282, no 5395, , p. 1842-1843 (ISSN 0036-8075, DOI 10.1126/science.282.5395.1842)
- (en) L. Florin, A. Tsokoglou et T. Happe, « A Novel Type of Iron Hydrogenase in the Green Alga Scenedesmus obliquus is Linked to the Photosynthetic Electron Transport Chain », J. Biol. Chem., vol. 276, , p. 6125–6132 (ISSN 0021-9258, DOI 10.1074/jbc.M008470200)
- (en) M. Frey, « Hydrogenases: Hydrogen-Activating Enzymes », ChemBioChem, vol. 3, nos 2-3, , p. 153–160 (ISSN 1439-4227, DOI 10.1002/1439-7633(20020301)3:2/3<153::AID-CBIC153>3.0.CO;2-B)
- (en) E. J. Lyon, S. Shima, R. Boecher, R. K. Thauer, F.-W. Grevels, E. Bill, W. Roseboom et S. P. J. Albracht, « Carbon Monoxide as an Intrinsic Ligand to Iron in the Active Site of the Iron−Sulfur-Cluster-Free Hydrogenase H2-Forming Methylenetetrahydromethanopterin Dehydrogenase As Revealed by Infrared Spectroscopy », J. Am. Chem. Soc., vol. 126, no 43, , p. 14239–14248 (ISSN 0002-7863, DOI 10.1021/ja046818s)
- (en) Y. Nicolet, B. J. Lemon, J. C. Fontecilla-Camps et J. W. Peters, « A novel FeS cluster in Fe-only hydrogenases », Trends Biochem. Sci., vol. 25, no 3, , p. 138-143 (ISSN 0968-0004, DOI 10.1016/S0968-0004(99)01536-4)
- (en) P. M. Vignais, B. Billoud et J. Meyer, « Classification and phylogeny of hydrogenases », FEMS Microbiol. Rev., vol. 25, no 4, , p. 455–501 (ISSN 1574-6976, DOI 10.1111/j.1574-6976.2001.tb00587.x)
Notes et références
- (en) Y. Nicolet, C. Piras, P. Legrand, E. C. Hatchikian et J. C. Fontecilla-Camps, « Desulfovibrio desulfuricans iron hydrogenase: the structure shows unusual coordination to an active site Fe binuclear center », Structure, vol. 7, no 1, , p. 13-23 (ISSN 0969-2126, DOI 10.1016/S0969-2126(99)80005-7)
- (en) J. W. Peters, W. N. Lanzilotta, B. J. Lemon et L. C. Seefeldt, « X-ray Crystal Structure of the Fe-Only Hydrogenase (CpI) from Clostridium pasteurianum to 1.8 Angstrom Resolution », Science, vol. 282, no 5395, , p. 1853-1858 (ISSN 0036-8075, DOI 10.1126/science.282.5395.1853)
- (en) A. S. Pandey, T. V. Harris, L. J. Giles, J. W. Peters et R. K. Szilagyi, « Dithiomethylether as a Ligand in the Hydrogenase H-Cluster », J. Am. Chem. Soc., vol. 130, no 13, , p. 4533–4540 (ISSN 0002-7863, DOI 10.1021/ja711187e)
- (en) J. H. P. Hackstein, « Eukaryotic Fe-hydrogenases – old eukaryotic heritage or adaptive acquisitions? », Biochem. Soc. Trans., vol. 33, , p. 47-50 (ISSN 0300-5127, PMID 15667261)
- (en) J. Balk, A. J. Pierik, D. J. A. Netz, U. Mühlenhoff et R. Lill, « The hydrogenase-like Nar1p is essential for maturation of cytosolic and nuclear iron–sulphur proteins », EMBO J., vol. 23, no 10, , p. 2105-2115 (ISSN 0261-4189, DOI 10.1038/sj.emboj.7600216)
- (en) C. Cavazza, L. Martin, S. Mondy, J. Gaillard, P. Ratet et J. C. Fontecilla-Camps, « The possible role of an [FeFe]-hydrogenase-like protein in the plant responses to changing atmospheric oxygen levels », J. Inorg. Biochem., vol. 102, nos 5-6, , p. 1359–1365 (ISSN 0162-0134, DOI 10.1016/j.jinorgbio.2008.01.027)
- (en) J. Huang, D. Song, A. Flores, Q. Zhao, S. M. Mooney, L. M. Shaw et F. S. Lee, « IOP1, a novel hydrogenase-like protein that modulates hypoxia-inducible factor-1a activity », Biochem. J., vol. 401, no 1, , p. 341–352 (ISSN 0264-6021, DOI 10.1042/BJ20060635)
- (en) D. Song et F. S. Lee, « A Role for IOP1 in Mammalian Cytosolic Iron-Sulfur Protein Biogenesis », J. Biol. Chem., vol. 283, no 14, , p. 9231-9238 (ISSN 0021-9258, DOI 10.1074/jbc.M708077200)
- (en) R. M. Barton et H. J. Worman, « Prenylated Prelamin A Interacts with Narf, a Novel Nuclear Protein », J. Biol. Chem., vol. 274, no 42, , p. 30008-30018 (ISSN 0021-9258, DOI 10.1074/jbc.274.42.30008)
- (en) A. Volbeda, M.-H. Charon, C. Piras, E. C. Hatchikian, M. Frey et J. C. Fontecilla-Camps, « Crystal structure of the nickel–iron hydrogenase from Desulfovibrio gigas », Nature, vol. 373, no 6515, , p. 580-587 (ISSN 0028-0836, DOI 10.1038/373580a0)
- (en) F. Leroux, S. Dementin, B. Burlat, L. Cournac, A. Volbeda, S. Champ, L. Martin, B. Guigliarelli, P. Bertrand, J. C. Fontecilla-Camps, M. Rousset et C. Léger, « Experimental approaches to kinetics of gas diffusion in hydrogenase », Proc. Natl. Acad. Sci. USA, vol. 105, no 32, , p. 11188-11193 (ISSN 0027-8424, DOI 10.1073/pnas.0803689105)
- (en) S. Shima, O. Pilak, S. Vogt, M. Schick, M. S. Stagni, W. Meyer-Klaucke, E. Warkentin, R. K. Thauer et U. Ermler, « The Crystal Structure of [Fe]-Hydrogenase Reveals the Geometry of the Active Site », Science, vol. 321, no 5888, , p. 572-575 (ISSN 0036-8075, DOI 10.1126/science.1158978).
- (en) S. Shima, E. J. Lyon, M. Sordel-Klippert, M. Kauss, J. Kahnt, R. K. Thauer, K. Steinbach, X. Xie, L. Verdier et C. Griesinger, « The Cofactor of the Iron–Sulfur Cluster Free Hydrogenase Hmd: Structure of the Light-Inactivation Product », Angew. Chem. Int. Ed., vol. 43, no 19, , p. 2547–2551 (ISSN 1433-7851, DOI 10.1002/anie.200353763).
Articles connexes
Liens externes
- 2B0J, PDB. Structure de l'apoenzyme hydrogénase sans cluster fer soufre de Methanothermococcus jannaschii
- 1HFE, PDB. Structure de l'hydrogénase à fer seul de Desulfovibrio desulfuricans
- 1C4A. Structure PDB de l'hydrogénase à fer seul de Clostridium pasteurianum
- 1UBR. Structure PDB de l'hydrogénase [NiFe] de Desulfovibrio vulgaris
- 1CC1. Structure PDB de l'hydrogènase [NiFeSe] de Desulfomicrobium baculatum