Agrobacterium radiobacter
Agrobacterium radiobacter[2], Rhizobium radiobacter[3] ou Agrobacterium tumefaciens est une bactérie à coloration de Gram négative trouvée dans les sols. C'est une bactérie permettant une symbiose créant des nodosités permettant la fixation d'azote[4], cru à tort comme pathogène des végétaux responsable d'une maladie appelée galle du collet (ou en anglais : crown-gall). A. tumefaciens a été identifiée à partir de galles des marguerites en 1907[5].
| Règne | Bacteria |
|---|---|
| Embranchement | Pseudomonadota |
| Classe | Alphaproteobacteria |
| Ordre | Hyphomicrobiales |
| Famille | Rhizobiaceae |
| Genre | Rhizobium |
(Beijerinck & van Delden, 1902) Conn 1942 emend. Hördt et al., 2020
- Rhizobium radiobacter (Beijerinck & van Delden, 1902) Young et al., 2001
Variétés de rang inférieur
- Agrobacterium radiobacter var. tumefaciens[1]
- Agrobacterium radiobacter var. radiobacter
Le mécanisme de formation des galles s'apparente à la conjugaison, il est dû à un plasmide bactérien, fragment d'ADN circulaire, appelé plasmide Ti qui rend les bactéries virulentes. Un fragment d'ADN du plasmide Ti, l'ADN-T est transféré de la bactérie vers la plante, puis intégré dans le génome végétal où il induit la formations des galles caractérisés par la multiplication désordonnée des cellules végétales. Agrobacterium tumefaciens infecte essentiellement les dicotylédones à la faveur d'une blessure.
Cette observation constitue le fondement d'une des techniques les plus utilisées en ingénierie génétique des végétaux pour produire des organismes génétiquement modifiés.
Taxonomie
La classification de cette bactérie, appartenant à un groupe présentant des particularités complexes, a été reconsidérée plusieurs fois. D'abord baptisée Bacterium tumefaciens par ses découvreurs de 1907[5], elle a été ensuite introduite dans le genre Agrobacterium, créé en 1942. À partir de 1993, des études phylogénétiques de l'ADN de différentes souches d'Agrobacterium et de Rhizobium aboutissent à la considération que les deux genres sont entrelacés et ne peuvent être distingués[6], formant un groupe d'espèces proches. En 2001, une autre étude considérait qu'Agrobacterium tumefaciens ne devait pas être considérée comme une espèce à part entière, mais comme une forme pathogène (parce que contenant certains plasmides) d'Agrobacterium radiobacter, décrite 1902 par Beijerinck et van Delden dans un contexte de symbiose racinaire permettant la fixation de l'azote, et seule espèce non phytopathogène du genre. Elle rejetait par conséquent la dénomination d'Agrobacterium tumefaciens, proposant de retenir radiobacter en tant qu'épithète d'origine, et Rhizobium pour le genre[7]. On sait aujourd'hui que cette bactérie permet également la fixation d'azote[4]. Ces conclusions taxonomiques ont été contestées en 2003[8], sur la base de différences moléculaires entre les "biovars", groupes identiques génétiquement mais présentant des caractéristiques biochimiques ou physiologiques différentes[9]. La problématique porte à la fois sur le statut des biovars, le rôle d'éléments génétiques transmissibles pour la classification, et le fait d'admettre ou non une nomenclature basée sur des propriétés particulières ("special purpose nomenclature")[10].
Des études plus récentes tendent vers la préservation du genre Agrobacterium[11]. Les progrès de l'analyse génomique ont abouti à améliorer en 2018-2019 l'analyse du génome de Agrobacterium radiobacter de 2014[12], qui aurait été réalisée sur des échantillons contaminés, et de considérer A. tumefaciens comme une sous-espèce d'Agrobacterium radiobacter. Les deux sous-espèces seraient appelées respectivement Agrobacterium radiobacter subsp. radiobacter, et Agrobacterium radiobacter subsp. tumefaciens[13].
Étymologie
"Agrobacterium" est formé de "bacterium", latin scientifique issu du grec, βακτηρία, baktêria, bâton, pour leur forme au microscope, de bâtonnet, et agro-, relatif aux champs, la bactérie vivant dans le sol. "Tumefaciens" signifie qui produit des tumeurs (de tumor, en latin, gonflement), pour désigner les galles qu'elle produit.
"Rhizobium" vient du grec ῥίζα, riza (« racine »), et de βίος, bíos (« vie »), qui renvoie à l'association symbiotique caractéristique de ce genre.
"Radiobacter" est construit à partir de radio-, lié aux rayons, et "bacter" pour bactérie (cf. ci-dessus).
Génome
Le génome d' Agrobacterium tumefaciens (souche de référence C58, séquencée en 2001) se compose de deux chromosomes, l'un linéaire de 2,8 Mb contenant une origine de réplication plasmidique et l'autre circulaire de 2 Mb portant une origine de réplication de type cori. A. tumefaciens C58 porte également deux plasmides AtC58 et TiC58 (le plasmide Ti) respectivement de 540 Kb et 214 Kb[14].
Cycle infectieux
La bactérie Agrobacterium tumefaciens infecte les végétaux (essentiellement des dicotylédones) à la faveur d'une blessure. Des composés phénoliques produits par la plante — généralement bactériostatiques ou antibiotiques — attirent au contraire Agrobacterium vers le site de la blessure. La bactérie s'attache aux cellules végétales. Sous l'action de ces composés phénoliques, Agrobacterium met en place un système de transfert d'un fragment de son ADN, vers la cellule blessée. Cet ADN, dit ADN-T (ou T-DNA en anglais), est porté par le plasmide Ti (tumor-inducing) et s'intègre au génome nucléaire de la cellule végétale. Les gènes portés par l'ADN-T s'expriment dans le végétal et conduisent:
- d’une part à la synthèse d'hormones de croissance végétales, l'auxine, et une cytokinine, dont la surproduction entraîne une multiplication anarchique des cellules végétales, d'où formation de la tumeur.

- d'autre part, à la synthèse par la cellule végétale de composés absents habituellement de ces cellules, appelés opines.
Les opines sont spécifiquement utilisées par les agrobactéries qui ont induit la formation de la tumeur. Cette spécificité est liée au fait que les gènes déterminant l'utilisation des opines sont portés par le plasmide Ti. De plus, certaines opines induisent le transfert du plasmide Ti d'une agrobactérie vers une autre par conjugaison. Les opines sont donc des médiateurs chimiques clefs de l'interaction Agrobacterium - plante, dont la présence dans la tumeur favorise la croissance des pathogènes et concourt à leur dissémination.
L'interaction Agrobacterium - plante peut donc être vue comme une manipulation génétique naturelle au cours de laquelle la bactérie détourne à son profit l'activité métabolique de la plante.
Étapes de la transfection
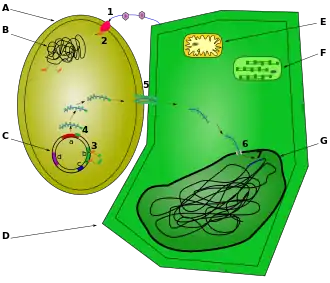
B: chromosomes d'Agrobacterium
C: Plasmide Ti : a: T-DNA , b: genes vir , c: origine de réplication , d: génes du catabolisme des opines
D: cellule végétale
E: Mitochondrie
F: Chloroplaste
G: Noyau
La bactérie perçoit les signaux phénoliques (qui sont émis par la plante) grâce à une protéine transmembranaire codée par le gène virA (1). La protéine VirA, possède une activité kinase, s'autophosphoryle et transfère le phosphate à une autre protéine, cytosolique cette fois et codée par virG (2). Cette dernière active la transcription des gènes de virulence (3).
La plante blessée émet également des signaux glucidiques, qui sont captés par une protéine codée par chvE. Celle-ci active VirA et la rend réceptive à des concentrations phénoliques faibles. L'augmentation du pH du milieu est captée par ChvI, qui active également les gènes de virulence.
Les gènes « vir » synthétisent plusieurs protéines dont une, VirD2, reconnaît les séquences double brin spécifiques des bordures droite et gauche de l'ADN-T. VirD1 ouvre les doubles brins, qui sont restreints (coupés) par VirD2; l'ADN-T est donc excisé du plasmide (4). La protéine VirD2 reste fixée sur la bordure droite de l'ADN-T, ce qui l'oriente pour sortir de la bactérie. Cette sécrétion s'effectue par un pilus protéique reliant la bactérie à la cellule végétale (5). Cet appareil de sécrétion (dit système de sécrétion de type IV) est essentiellement codé par les gènes de l'opéron virB, et par virD4 Il a été proposé que le complexe nucléoprotéique VirD2-ADN-T soit recruté par la protéine de couplage VirD4 pour traverser la membrane interne puis serait prix en charge par le complexe composé des produits des gènes virB [15]. Avant ou après son entrée dans la cellule (selon les auteurs), l'ADN-T est recouvert de plusieurs monomères de la protéine VirE2. Celle-ci est également d'origine bactérienne, codée par le gène virE2, et permet de protéger l'ADN-T monocaténaire de la dégradation dans la cellule végétale. VirD2 et VirE2 possèdent des séquences signal de localisation nucléaire dites NLS (pour Nuclear Localisation Signal) qui sont reconnues par la cellule végétale, permettant d'adresser le complexe nucléoprotéique ADN-T/VirE2/VirD2 vers le noyau de la cellule végétale (6). Ce complexe pénètre donc dans le noyau, où il s'intègre à l'ADN végétal. Les ADN polymérases du végétal l'utilisent comme matrice, et le transforment en ADN double brin (7).
Les gènes codés par l'ADN-T ne sont généralement pas exprimés dans une cellule procaryote, donc chez Agrobacterium.
Le génie génétique végétal repose en grande partie sur l'utilisation d' Agrobacterium comme vecteur naturel de gènes. La méthode est appelée "Agrobacterium tumefaciens-Mediated Transformation" (ATMT). Les biotechnologistes utilisent le plus souvent des plasmides proches du plasmide Ti, dit plasmides désarmés, car leur ADN-T ne porte plus les gènes responsables du pouvoir pathogène. Sur ces plasmides désarmés, les gènes tumoraux sont en effet remplacés par un (ou plusieurs) gène d’intérêt agronomique (par exemple le gène codant la toxine de Bacillus thuringiensis dit gène Bt, ou des gènes de résistance à l'herbicide non sélectif glyphosate) et par un ou plusieurs gènes marqueurs permettant de sélectionner les cellules transformées, puis de les multiplier sur des milieux de culture, in vitro. Des plantes entières sont ensuite régénérées par des techniques classiques de culture in vitro, faisant intervenir les hormones végétales auxine et cytokinine.
Lien avec LUCA
Une étude réalisée en 2003 montre que Agrobacterium tumefaciens présente les caractéristiques d'un acidocalcisome dans sa structure, organite que l'on pensait n'exister que chez les archées et les eucaryotes. Cette découverte est la première à montrer une caractéristique commune à tous les domaines du vivant.
Plantes infectées
Parmi les genres de plantes qui peuvent être touchées par Agrobacterium tumefaciens, toutes des dicotylédones, on peut mentionner[16]:
- des feuillus : les érables (Acer), le marronnier (Aesculus), les aulnes (Alnus), des bouleaux (Betula pendula, B. pubescens), le cornouiller sanguin (Cornus sanguinea) ; l'aubépine (Crataegus), le cognassier (Cydonia oblonga), le bois-gentil (Daphne mezereum), les fusains (Euonymus), les forsythias (Forsythia), les frênes (Fraxinus), le chêne pubescent (Quercus pubescens), le robinier (Robinia pseudoacacia), des saules (Salix), les ormes (Ulmus) ;
- des conifères : le cyprès de Lawson (Chamaecyparis lawsoniana), le pin d'Alep (Pinus halepensis) ;
- des plantes herbacées : les asters (Aster, Symphyotrichum novi-belgii), la bette (Beta vulgaris), des brassicacées (Brassica napus, B. oleracea), des chrysanthèmes (Argyranthemum frutescens, Chrysanthemum indicum), le dahlia commun (Dahlia pinnata), des daturas (Datura innoxia,D. stramonium), la carotte (Daucus carota), les glaïeuls (Gladiolus), des Rubus.
Notes et références
- PMC 6369824
- « Tomate - Agrobacterium radiobacter », sur inra.fr (consulté le ).
- J M Young, L D Kuykendall, A Kerr et E Martínez-Romero, « A revision of Rhizobium Frank 1889, with an emended description of the genus, and the inclusion of all species of Agrobacterium Conn 1942 and Allorhizobium undicola de Lajudie et al. 1998 as new combinations: Rhizobium radiobacter, R. rhizogenes, R. rubi, R. undicola and R. vitis. », International Journal of Systematic and Evolutionary Microbiology, vol. 51, , p. 89-103 (DOI 10.1099/00207713-51-1-89, lire en ligne, consulté le )
- (en) Lalita Kanvinde et G. R. K. Sastry, « Agrobacterium tumefaciens Is a Diazotrophic Bacterium », Appl Environ Microbiol, vol. 56, no 7, (ISSN 2087–2092[à vérifier : ISSN invalide], PMID 16348237, PMCID PMC184564, DOI 10.1128/aem.56.7.2087-2092.1990, lire en ligne)
- (en) Smith EF, Townsend CO, « A PLANT-TUMOR OF BACTERIAL ORIGIN », Science, vol. 25, no 643, , p. 671–3 (PMID 17746161, DOI 10.1126/science.25.643.671)
- HIROYUKI SAWADA, HIROYUKI IEKI, HIROSHI OYAIZU et SATOSHI MATSUMOTO, « Proposal for Rejection of Agrobacterium tumefaciens and Revised Descriptions for the Genus Agrobacterium and for Agrobacterium radiobacter and Agrobacterium rhizogenes », International Journal of Systematic and Evolutionary Microbiology, vol. 43, no 4, , p. 694–702 (DOI 10.1099/00207713-43-4-694, lire en ligne, consulté le )
- (en) J M Young, L D Kuykendall, E Martínez-Romero et A Kerr, « A revision of Rhizobium Frank 1889, with an emended description of the genus, and the inclusion of all species of Agrobacterium Conn 1942 and Allorhizobium undicola de Lajudie et al. 1998 as new combinations: Rhizobium radiobacter, R. rhizogenes, R. rubi, R. undicola and R. vitis. », International Journal of Systematic and Evolutionary Microbiology, vol. 51, no 1, , p. 89–103 (DOI 10.1099/00207713-51-1-89, lire en ligne [PDF], consulté le )
- Stephen K. Farrand, Peter B. van Berkum et Philippe Oger, « Agrobacterium is a definable genus of the family Rhizobiaceae », International Journal of Systematic and Evolutionary Microbiology, vol. 53, no 5, , p. 1681–1687 (DOI 10.1099/ijs.0.02445-0, lire en ligne, consulté le )
- (en) « the definition of biovar », sur www.dictionary.com (consulté le )
- (en) J. M. Young, L. D. Kuykendall, E. Martínez-Romero et A. Kerr, « Classification and nomenclature of Agrobacterium and Rhizobium – a reply to Farrand et al. (2003) », International Journal of Systematic and Evolutionary Microbiology, vol. 53, no 5, , p. 1689–1695 (DOI 10.1099/ijs.0.02762-0, lire en ligne [PDF], consulté le )
- (en) Han Ming Gan et Michael A Savka, « One More Decade of Agrobacterium Taxonomy », sur ResearchGate (consulté le )
- Linshuang Zhang, Xiangyang Li, Feng Zhang et Gejiao Wang, « Genomic analysis of Agrobacterium radiobacter DSM 30147T and emended description of A. radiobacter (Beijerinck and van Delden 1902) Conn 1942 (Approved Lists 1980) emend. Sawada et al. 1993 », Standards in Genomic Sciences, vol. 9, no 3, , p. 574–584 (ISSN 1944-3277, PMID 25197445, PMCID PMCPMC4149017, DOI 10.4056/sigs.4688352, lire en ligne, consulté le )
- (en) Han Ming Gan, Melvin V.L. Lee et Michael A. Savka, « Improved genome of Agrobacterium radiobacter type strain provides new taxonomic insight into Agrobacterium genomospecies 4 », PeerJ 7:e6366, (DOI 10.7717/peerj.6366, lire en ligne)
- (en) Goodner B, Hinkle G, Gattung S, et al., « Genome sequence of the plant pathogen and biotechnology agent Agrobacterium tumefaciens C58 », Science, vol. 294, no 5550, , p. 2323–8 (PMID 11743194, DOI 10.1126/science.1066803)
- « Science Magazine: Sign In » (consulté le )
- (en-US) « Agrobacterium tumefaciens – Plant Parasites of Europe » (consulté le )
Voir aussi
- Pathologie végétale
- Nodule (botanique)
- Milieu de culture
- Agroinfiltration
- Génie génétique
- Transgenèse
- CP4 EPSPS (transgène permettant la tolérance au glyphosate)
Liens externes
- (en) Référence BioLib : Agrobacterium tumefaciens (Smith & Townsend, 1907) (consulté le )
- (fr) Référence Catalogue of Life : Agrobacterium tumefaciens (Smith & Townsend, 1907) Conn, 1942 (Nom accepté: Rhizobium radiobacter (Beijerinck & van Delden, 1902) Young & al., 2001) Non Valide (consulté le )
- (en) Référence Catalogue of Life : Rhizobium radiobacter (Beijerinck & van Delden, 1902) Young et al., 2001 (consulté le )
- (fr+en) Référence ITIS : Agrobacterium tumefaciens (Smith & Townsend, 1907) Conn, 1942 (Nom accepté: Rhizobium radiobacter (Beijerinck & van Delden, 1902) Young & al., 2001) Non valide (consulté le )
- (fr+en) Référence ITIS : Rhizobium radiobacter (Beijerinck & van Delden, 1902) Young & al., 2001 (consulté le )
- (en) Référence NCBI : Agrobacterium tumefaciens (taxons inclus) (consulté le )
- (en) Référence uBio : site déclaré ici indisponible le 7 avril 2023
