ADN polymérase
Une ADN polymérase est une enzyme faisant partie du complexe enzymatique intervenant dans la réplication de l’ADN au cours du cycle cellulaire lors de la phase S, mais aussi dans des processus de réparation et de recombinaison de l'ADN. Les ADN polymérases utilisent des désoxyribonucléosides triphosphate comme base pour la synthèse d'un brin d'ADN, en utilisant un autre brin d'ADN comme matrice. Ce processus réplicatif utilise la complémentarité des bases nucléiques pour guider la synthèse du nouveau brin à partir du brin matrice. Il existe plusieurs familles de polymérases, qui diffèrent selon leur séquence en acides aminés et leurs propriétés catalytiques.
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
Mécanisme
Démarrage
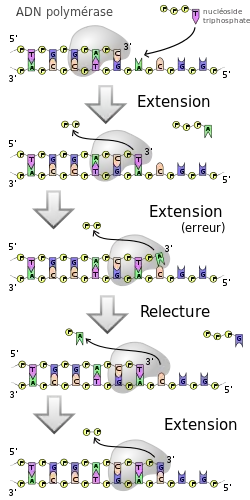
Toutes les ADN polymérases synthétisent l’ADN dans le sens 5’ → 3’, chez tous les organismes vivants, et aucune n’est capable de commencer la synthèse d'un brin de novo. Elles ne peuvent qu'ajouter des nucléotides à une extrémité hydroxyle libre, en général le 3’-OH du brin en cours de synthèse. Pour cette raison, l'ADN polymérase a besoin d’une amorce (ou primer), sur laquelle ajouter de nouveaux désoxyribonucléotides.
L’amorce peut être formée d’ADN ou d’ARN. Dans le cas de la réplication, l'amorce constituée d'ARN est synthétisée par une autre enzyme, appelée primase, au sein du complexe de réplication appelé réplisome. Celui-ci contient également une enzyme, l'hélicase, qui est nécessaire pour séparer les deux brins de l’ADN et ainsi permettre l’accès des ADN polymérases et la réplication. Dans le cas des processus de réparation et de recombinaison, l'amorce est constituée d'un segment d'ADN résultant de la coupure par une endonucléase du brin endommagé ou recombiné, qui libère une extrémité 3’-OH libre.
Élongation et processivité
Les diverses ADN polymérases se différencient par leur capacité à allonger l'ADN sans se dissocier du brin matrice, une caractéristique qui s'appelle la processivité. Certaines ADN polymérases sont faiblement associées à leur substrat et se détachent après avoir polymérisé seulement quelques nucléotides. On parle alors d'ADN polymérases distributives, ce qui est une caractéristique de beaucoup de polymérases impliquées dans les processus de réparation. Les ADN polymérases réplicatives, au contraire, sont très fortement liées au brin matrice et ne se dissocient pas. Elles peuvent polymériser plusieurs centaines de milliers de nucléotides en une seule fois. On parle alors d'ADN polymérases processives. Un cofacteur protéique, la pince (ou clamp), appelé PCNA chez les eucaryotes et les archées, et complexe β chez les bactéries, se lie aux polymérases réplicatives et augmente considérablement leur processivité.
L'activité des ADN polymérases nécessite la présence d’ions Mg2+ comme cofacteurs qui se fixent en particulier sur les groupes phosphate des nucléotides et de l'ADN.
Fidélité et activité de relecture
Certaines ADN polymérases, qui disposent aussi d'une activité exonucléase 3’ → 5’, ont la capacité de corriger les erreurs d'incorporation dans le brin néoformé. Lorsque l'ADN polymérase fait une erreur et qu'un mésappariement est formé au niveau du site actif de l'enzyme, celle-ci peut revenir en arrière et hydrolyser le nucléotide incorrect : c'est l’activité exonucléase 3’-5’, appelée également fonction d'édition. Elle peut alors réinsérer la base correcte, et reprendre la réplication. Ce processus de relecture par l'ADN polymérase améliore la fidélité du processus réplicatif et fait baisser le taux d'erreur.
Au contraire, certaines ADN polymérases sont peu fidèles et font des erreurs d'incorporation de nucléotides avec une fréquence plus élevée. Ces ADN polymérases sont utilisées spécifiquement dans les processus de réparation de l'ADN. Leur spécificité relâchée leur permet de répliquer de l'ADN contenant des lésions, c'est-à-dire des bases altérées par des modifications chimiques, résultant par exemple de l'action de rayonnement UV, ionisant ou d'agents mutagènes. On parle d'ADN polymérases translésionnelles.
On parle également d'une fonction de correction d'épreuves ou de correction sur épreuves (« proofreading » en anglais) des ADN polymérases.
ADN polymérases chez les procaryotes
Cinq ADN polymérases ont été identifiées chez les procaryotes :
- Pol I : impliquée dans la réparation et la réplication de l’ADN. Elle possède une activité polymérase 5’ → 3’, une activité exonucléase 3’ → 5’ pour la relecture, et enfin une activité exonucléase 5’ - 3’ qui lui permet d'éliminer les amorces d'ARN des fragments d'Okazaki pendant la réplication. Son activité polymérase lui permet de synthétiser de l'ADN destiné à être immédiatement suturé par une ADN ligase.
- La Pol I est peu processive et se dissocie de la matrice après l'ajout d'une vingtaine de nucléotides.
- En biologie moléculaire, seule une partie de la protéine, appelée fragment de Klenow, est employée. Ce fragment contient l'activité polymérase 5’ → 3’ et l'activité exonucléase 3’ → 5’.
- Pol II : impliquée dans la réplication de l'ADN endommagé, elle possède une activité polymérase 5’ → 3’ et une activité exonucléase 3’ → 5’.
- La Pol II est peu processive.
- Pol III : c’est la principale polymérase, qui intervient dans l'élongation de la chaîne d'ADN lors de la réplication au niveau du brin avancé et de la synthèse des fragments d’Okazaki. Elle est formée de trois sous-unités qui en constituent l'holoenzyme :
- la sous-unité α (alpha) possède l'activité polymérase 5’ → 3’,
- la sous-unité ε (epsilon) possède l'activité exonucléase 3’ → 5’, et
- la sous-unité θ (thêta) stimule l'activité de « proofreading » de la précédente.
- Cette holoenzyme forme le noyau d'un réplisome constitué en tout de dix protéines :
- deux holoenzymes de polymérase III,
- deux unités β (bêta) agissant comme pince à ADN (clamp),
- deux unités τ (tau) permettant de dimériser l'holoenzyme de polymérase III,
- une unité γ (gamma) dite chargeur de clamp elle-même constituée de trois sous-unités γ proprement dites, une sous-unité δ (delta) et une sous-unité δ’ (delta prime), et enfin
- une unité Χ (Khi) et une unité Ψ (Psi) formant un complexe 1:1 qui se lie à l'unité τ ou à l'unité γ.
- Pendant la réplication, le complexe γ se place à l'ouverture de la fourche de réplication en contact avec l'hélicase. Il déplace les sous-unités β (appelées aussi clamp bêta) autour de chaque brin d'ADN matrice comme des pinces pour permettre à l'holoenzyme qui lui est liée de glisser sur le brin matrice. De plus, le complexe γ est lié aux deux unités τ elles-mêmes liées aux deux sous-unités α des holoenzymes.
- La Pol III est très processive.
- Pol IV : ADN polymérase de la famille Y.
- Pol V : ADN polymérase de la famille Y.
ADN polymérases chez les eucaryotes
- Pol α (alpha) : à la suite des amorces d'ARN synthétisées par la primase lors de l'initiation, Pol α synthétise de courtes séquences d'ADN d'une vingtaine de nucléotides de long. Cette polymérase ne possède pas de fonction exonucléasique 3’ → 5’. Elle est associée en un complexe avec la primase responsable de la synthèse de l'amorce d'ARN[1].
- Pol β (bêta) : cette polymérase est impliquée dans des processus de réparation de l'ADN. Elle ne possède pas de fonction exonucléasique. Elle correspond à la Pol II bactérienne.
- Pol γ (gamma) : cette polymérase de la famille A intervient dans la réplication de l'ADN mitochondrial.
- Pol δ (delta) : c'est la polymérase principale qui intervient dans la réplication de l'ADN chez les eucaryotes, avec l'ADN Pol ε, dans la synthèse du brin avancé et du brin retardé. Elle possède aussi une activité exonucléasique 3' vers 5' intervenant dans la correction des erreurs et dans des processus de réparation. La polymérase δ est hautement processive lorsqu'elle est associée au PCNA. Cette polymérase correspond à la Pol III bactérienne.
- Pol ε (epsilon) : elle possède une activité polymérase 5’ → 3’ et une activité exonucléase 3’ → 5’ et intervient dans la réplication et la réparation de l'ADN. Elle lit dans le sens 3’ → 5’ et synthétise 5’ → 3’ la zone du télomère qui ne peut être synthétisée par la Pol δ (une amorce préalable doit être posée par la primase sur l'extrémité 3’ allongée du télomère). Une ligase réalise ensuite la suture entre les deux brins.
- Pol ζ (zêta) : polymérase de la famille B.
- Pol η (êta) : polymérase de la famille Y.
- Pol θ (thêta) : polymérase de la famille A. Une récente publication indique que si elle ne fait pas une excellente transcriptase, elle est capable d'effectuer une rétrotranscription[2].
- Pol ι (iota) : polymérase de la famille Y.
- Pol κ (kappa) : polymérase de la famille Y.
- Rev1 : polymérase de la famille Y.
ADN polymérases chez les archées
- Polymérase B : toutes les ADN polymérases de type B identifiées chez les archées présentent une activité polymérase 5’ → 3’ et une activité exonucléase 3’ → 5’. Elles sont constituées d'une seule sous-unité.
- Polymérase D : ces ADN polymérases trouvées chez les Euryarchaeota sont constituées de deux sous-unités différentes. La grande sous-unité DP2 possède l'activité catalytique, mais seulement en présence de la petite sous-unité DP1.
Familles d'ADN polymérases
Sur la base d’homologie de séquence protéique et de structure, les polymérases peuvent être divisées en différentes familles : A, B, C, D, X, Y et RT.
Famille A
Les polymérases de la famille A contiennent des polymérases réplicatives et des polymérases de réparation. Les polymérases réplicatives de cette famille comprennent notamment la polymérase du bactériophage T7 et l’ADN polymérase gamma des eucaryotes. Parmi les ADN polymérases de réparation, on peut trouver l’ADN polymérase I d'E. coli, de Thermus aquaticus et de Bacillus stearothermophilus. Ces polymérases de réparation sont impliquées dans des processus de réparation par excision de base et dans la synthèse des fragments d’Okazaki du brin retardé lors de la réplication.
Famille B
Cette famille regroupe essentiellement des ADN polymérases réplicatives et comprend les ADN polymérases Pol α, Pol δ et Pol ε des eucaryotes. Une ADN polymérase de la famille B a été identifiée chez toutes les espèces d’archées examinées. La famille B contient également des ADN polymérases provenant de bactéries et de bactériophages, dont les mieux caractérisées sont celles des phages T4 et RB69. Ces enzymes sont impliquées dans la synthèse du brin avancé et du brin retardé.
Famille C
Les ADN polymérases de cette famille ont été isolées chez des bactéries. Elles possèdent aussi une activité exonucléase 5’ → 3’.
Famille D
Les ADN polymérases de la famille D restent encore peu caractérisées et ont été trouvées uniquement chez des Euryarchaeota, un embranchement (phylum) du règne des archées.
Famille X
La famille X comprend l'ADN polymérase β bien caractérisée chez les eucaryotes et d'autres ADN polymérases comme la Pol σ, la Pol λ, et la Pol μ. L'ADN polymérase β est nécessaire au processus de réparation de l'ADN appelé BER (réparation par excision de base). La Pol λ et la Pol μ interviennent dans des processus de réparation de l'ADN impliquant des cassures doubles brins.
Famille Y
Les polymérases de la famille Y sont caractérisées par leur capacité à tolérer des lésions de l’ADN lors de la réplication. Elles sont appelées polymérases de translésion (TLS pour translesion sythesis polymerases). Elles ne possèdent pas de fonction exonucléase 3’ → 5’. Leur fidélité lors de la synthèse d’ADN est faible, même en absence d’ADN endommagé.
Famille RT
La famille des transcriptases inverses comprend des ADN polymérases de rétrovirus et d’eucaryotes. Ces polymérases sont capables de synthétiser de l’ADN à partir d’une matrice d'ARN.
La polymérase thêta pourrait être capable d'effectuer une rétrotranscriptase[2].
Bibliographie
- Garg, P. et P. M. Burgers. 2005. DNA polymerases that propagate the eukaryotic DNA replication fork. Crit Rev Biochem Mol Biol 40:115-28.
- Hubscher, U., G. Maga, et S. Spadari. 2002. Eukaryotic DNA polymerases. Annu Rev Biochem 71:133-63.
- Kelman, Z. 2000. DNA Replication in the Third Domain (of Life). Current Protein and Peptide Science 1:139-154.
- Johnson, A., and M. O'Donnell. 2005. Cellular DNA replicases: components and dynamics at the replication fork. Annu Rev Biochem 74:283-315.
- Nohmi, T. 2006. Environmental Stress and Lesion-Bypass DNA Polymerases. Annu Rev Microbiol.
Voir aussi
Notes et références
- (en) Marco Muzi-Falconi, Michele Giannattasio, Marco Foiani et Paolo Plevani, « The DNA Polymerase α-Primase Complex: Multiple Functions and Interactions », The Scientific World, vol. 3, (ISSN 1537-744X, DOI 10.1100/tsw.2003.05, lire en ligne, consulté le )
- (en) Gurushankar Chandramouly, Jiemin Zhao, Shane McDevitt et Timur Rusanov, « Polθ reverse transcribes RNA and promotes RNA-templated DNA repair », Science Advances, vol. 7, no 24, , eabf1771 (ISSN 2375-2548, PMID 34117057, DOI 10.1126/sciadv.abf1771, lire en ligne, consulté le )