Lactococcus lactis
Lactococcus lactis (ou lactocoque lactique) est une bactérie à Gram positif, non mobile, non sporulante, mesurant en moyenne de 0,5 à 1,5 micromètre. Les cellules de ce micro-organisme se regroupent habituellement en paires ou en courtes chaînes et sont communément isolées dans les produits végétaux, céréales, haricots, pois, pomme de terre, etc. Avec leur microflore fongique et microbienne très diversifiée, les lait crus de ruminants sont des milieux naturellement riches en lactocoques. On considère que la surface des trayons constitue la principale source de contamination du lait[2].
Lactococcus lactis est une bactérie lactique homofermentaire, son métabolisme est hétérotrophe, anaérobie aérotolérante. Sa température optimale de croissance se situe aux environs de 30 °C (elle est dite mésophile). Elle comporte habituellement un ou plusieurs plasmides portant des gènes codant des fonctions d'adaptation à l'environnement[3].
Les deux sous-espèces lactiques L. lactis subsp. lactis et subsp. cremoris sont largement utilisées dans les levains laitiers pour la fabrication de fromages ou d'autres produits laitiers fermentés. Un biovar de la sous-espèce lactis, nommé diacetylactis, est capable de métaboliser les citrates en diacétyle et acétoïne, conduisant à la flaveur caractéristique des fromages frais.
L. lactis est une bactérie modèle pour la recherche fondamentale qui est depuis plusieurs décennies, l'objet de très nombreuses études. C'est aussi la première bactérie lactique dont le génome a été entièrement séquencé[4].
Taxonomie
Depuis les premières descriptions de Lactococcus lactis par Lister en 1873[N 1] puis par Orla-Jensen en 1919 sous deux autres noms, la classification taxonomique de cette bactérie a passablement changé. Elle fut d'abord désignée comme un streptocoque lactique car elle était en forme de coque et se regroupait en chaînettes de longueurs variables. Les études de Schleifer et coll. (1985) ont ensuite justifié la création d'un nouveau genre bactérien nommé Lactococcus regroupant la quasi-totalité des streptocoques d'un ancien groupe N. La bactérie fut donc nommée Lactococcus lactis et regroupée avec les espèces garviae, piscium, plantarum, raffinolactis. C'est aussi à cette époque qu'elle fut reconnue comme agent primaire de l'acidification du lait caillé[5].
Actuellement la taxonomie de L. lactis basée sur le phénotype comporte quatre sous-espèces[6] - [7] :
- Lactococcus lactis subsp. lactis
- L. l. lactis biovar. diacetylactis
- Lactococcus lactis subsp. cremoris
- Lactococcus lactis subsp. hordniae.
- Lactococcus lactis subsp. tructae
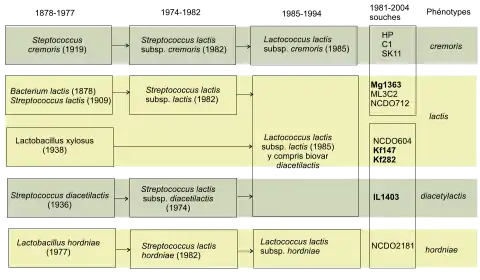
- Les sous-espèces
La sous-espèce lactis peut être distinguée par sa plus grande résistance au stress : elle est capable de croître à 40 °C ou en présence de 4 % de NaCl. Elle est de plus capable de produire de l'ammoniac à partir d'arginine. Les souches de la biovar diacetylactis sont capables de métaboliser le citrate et de le convertir en diacétyle, un composé aromatique apprécié en fromagerie pour son arôme de beurre.
Les deux sous-espèces L. lactis subsp. lactis et L. lactis subsp. cremoris sont les plus intéressantes pour la production fromagère puisqu'elles entrent dans la composition de la quasi-totalité des levains fromagers. La sous-espèce lactis est mieux adaptée à la fabrication des fromages à pâte fraiche ou molle et la sous-espèce cremoris pour ceux à pâte pressée. Cette dernière est en outre réputée faire des fromages de meilleure qualité en raison de sa contribution spécifique à la saveur. En dehors de l'environnement laitier, la sous-espèce L. lactis subsp. lactis a été isolée sur des végétaux, dans l'appareil uro-génital féminin et le système gastro-intestinal de certains poissons d'eau douce. Par contre, les tentatives d'isoler des souches sauvages de phénotype cremosis ou diacetylactis dans l'environnement ont rencontré peu de succès[8]. Les levains fromagers et en particulier ceux de phénotypes cremosis et diacetylactis ont été sélectionnés pour leur rôle dans la production de fromages et ont évolué en conséquence, si bien qu'ils ne sont plus aptes à survivre hors de l'environnement des laiteries.
Les sous-espèces hordniae et tructae n'ont pas été isolées dans les produits laitiers mais la première a été isolée d'une cicadelle (Hordnia circellata) et la seconde du mucus intestinal de la truite brune (Salmo trutta) et de la truite arc-en-ciel (Oncorhynchus mykiss) .
- La génétique
Le séquençage du génome de la souche IL1403 de Lactococcus lactis ssp. lactis a été réalisée en 2001 par A. Bolotin et als[9]. Par la suite, de nombreuses souches ont été séquencées. En 2013, étaient séquencées et disponibles dans la base de données GenBank (NCBI) les génomes des souches de L. lactis subsp. cremoris MG1363, NZ9000, SK11, A76, UC509.9 etc. et pour L. lactis subsp. lactis les souches IL1403, KF147, CV56, IO-1 etc.
Parmi toutes ces souches, L. lactis subsp. cremoris MG1363 est sans conteste la souche modèle pour l'étude de la génétique des bactéries lactiques et le développement d'applications biotechnologiques. Cette souche qui ne contient aucun plasmide permet l'introduction de gènes de lactocoques supplémentaires et de gènes provenant d'un autre organisme (gènes hétérologues) en raison de la stabilité de son génome[10]. De nombreuses fonctions essentielles pour la réussite de la fermentation sont portées par des plasmides.
Le lait est un habitat privilégié des lactocoques[11] où on les trouve à des niveaux pouvant varier de 10 à 10 000 UCF ml−1. Les multiples études génétiques des isolats de laiteries ont mis en évidence deux grands génotypes, nommés génotype cremosis et génotype lactis, (voir tableau 1, les boîtes dans l'avant dernière colonne) qui ne correspondent pas toujours aux phénotypes. Ainsi dans le génotype cremosis, on a des souches de phénotypes[6] lactis (MG1363) et cremosis (SK11) et de même avec le groupe des génotypes lactis, il y a des phénotypes lactis (KF147) mais aussi des phénotypes biovar diacetylactis (IL594). Pour illustrer ce phénomène, citons l'étude de Corroler[12] (1999) menée dans la zone d'appellation « camembert de Normandie » sur la biodiversité des lactococoques originels des laits crus. La flore des Lactococcus de six exploitations laitières a été caractérisée sur les plans phénotypique et génotypique, au cours de deux saisons. L'analyse phénotypique a montré une représentation exclusive de Lactococcus lactis subsp. lactis alors que l'analyse génotypique révélait la présence des deux sous-espèces lactis et cremosis au sein des différentes exploitations.
Métabolisme des glucides
La bactérie Lactococcus lactis est très utilisée dans la fabrication du fromage en raison de son aptitude à acidifier le milieu pour faciliter la formation du caillé mais aussi en raison de sa participation à la texture et à la saveur du produit fermenté. Le principal mécanisme détourné par l'homme est le catabolisme des sucres qui est naturellement utilisé par les lactocoques pour tirer leur énergie de l'environnement. La croissance de L. lactis dans le lait s'accompagne d'une conversion rapide du lactose en acide lactique. C'est cette acidification du milieu qui protégera ensuite le produit fermenté de la contamination par des bactéries pathogènes ou d'altération, facilitera la formation du caillé et créera les conditions optimum pour l'affinage.
Le lactose est le principal sucre du lait et son transport à l'intérieur de la bactérie, son métabolisme, la formation d'acide lactique et son expulsion extracellulaire ont été bien étudiés[6] - [13]. Le lactose est un disaccharide formé d'un galactose lié à un glucose.
- Le transport des sucres à l'intérieur de la bactérie
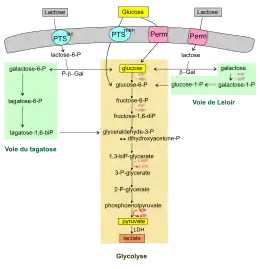
Chez L. lactis le transport membranaire des sucres à l'intérieur est assuré principalement par deux procédés : le système phosphotransférase phosphoénolpyruvate dépendant ou PTS qui couple le transport et la phosphorylation du glucide et le système perméase qui couple la translocation du sucre à celle d'un proton, suivi d'une phosphorylation ultérieure dans la cellule[4].
Le système phosphotransférase spécifique du lactose (PTSlac) a une forte affinité pour ce sucre et réalise un transport très efficace sur le plan énergétique puisqu'une molécule de lactose est transportée et phosphorylée au prix d'un seul équivalent ATP. Le lactose est ensuite hydrolysé par la P-β-galactosidase en glucose et galactose-6-P[6] qui vont rejoindre la glycolyse suivant des voies différentes. Alors que le glucose emprunte directement la voie de la glycolyse[N 2], le galactose-6-P doit passer par la voie du tagatose avant d'entrer dans la glycolyse. L'opéron engagé dans cette fermentation homolactique[N 3] rapide sont habituellement situés sur un plasmide (lac-plasmide).
Les souches de L. lactis qui fermentent lentement le lactose produisent généralement divers produits finaux comme des acétates, formiates, éthanol et lactate. Le métabolisme lent du lactose est lié à un système perméase-β-galactosidase (Perm) qui ne se rencontre que chez peu de souches laitières de L. lactis comme IL1403, et chez deux souches non laitières[6] (NCDO2054 et KF147 isolée dans les germes de haricots mungo). Après son transfert via le système perméase, le lactose est hydrolysé en glucose et galactose (par la β-galactosidase) qui rejoignent la voie de glycolyse par des voies différentes. Le glucose emprunte directement la voie de la glycolyse alors que le galactose est dirigé vers la voie de Leloir avant de rejoindre la glycolyse[13] (voir figure 1). Finalement, il semble que le système perméase-β-galactosidase ne joue qu'un rôle mineur dans l'assimilation du lactose par les souches de L. lactis.
Les lactocoques cultivés dans un milieu riche en lactose comme le lait, absorbent le lactose mais en présence d'autres sucres, ils sont aussi aptes à les absorber et les métaboliser. Ainsi, le système de transport principal du glucose de L. lactis est le mannose-PTS (PTSman) qui peut aussi transporter le mannose, la glucosamine et le fructose[14].
Outre le lactose et le glucose, les systèmes PTS peuvent transporter aussi des β-glucosides tels que l'amygdaline, l'arbutine, le cellobiose, l'esculine et la salicine. À part l'amygdaline, ces molécules sont constituées d'un aglycone relié par une liaison β-glucoside à au moins un sucre. Le cellobiose est l'unité structurale du polymère le plus abondant sur terre, la cellulose. Après translocation par le système PTS, le P-glucoside est hydrolysé en glucose et glucose-6-P ou les divers aglycones.
- La glycolyse
La glycolyse est la voie principale du métabolisme du glucose. Cette voie est rejointe directement ou en cours de route par les produits résultant du clivage du lactose (en glucose et galactose), via la voie de Leloir ou du la voie du tagatose.
La glycolyse est un mécanisme de régénération d'énergie sous forme d'ATP qui ne nécessite pas d'oxygène. Une mole de glucose produit deux moles de pyruvate et deux moles d'ATP.
Pour les bactéries lactiques homofermentaires comme le Lactococcus lactis, le pyruvate est ensuite transformé en acide lactique (lactate), produit essentiel de la fermentation lactique anaérobie, grâce à une lactate déshydrogénase (LDH).
Métabolisme du citrate
Les lactococoques lactiques comme certaines bactéries lactiques sont capables de métaboliser l'acide citrique (ou citrate) en condition d'anaérobiose pour produire des composés responsables des arômes des produits laitiers fermentés. L'acide citrique présent dans le lait est considéré comme le principal précurseur de la formation des composés aromatiques appréciés comme l'acétate, l'acétoïne (odeur de beurre), le diacétyle (odeur de fromage) et 2,3-butanediol (arôme beurré). Les 1 750 mg de citrate contenu dans chaque litre de lait se trouve dans la phase liquide et partent en partie avec l'égouttage du petit-lait.
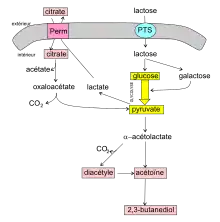
Le citrate est transporté à l'intérieur de la cellule bactérienne par le biais d'une perméase protonique, codée par le gène citP inclus dans l'opéron citQRP d'un plasmide[15]. La citrate perméase a une activité optimale à un pH compris entre 4,45 et 5,2. Le temps durant lequel le produit fermenté reste dans cette zone de pH est directement corrélé avec la quantité de diacétyle produit. Des expériences in vitro ont montré que le rôle de citP est de capter le citrate et en même temps d'excréter le lactate produit par la glycolyse. Plus il y a d'acide lactique et plus la dégradation du citrate est prononcée. En fin de fermentation lactique, le lactate s'est accumulé dans le milieu à des concentrations telles que la croissance est inhibée. Les bactéries capables de fermenter le citrate ont donc un moyen de résister aux effets inhibiteurs du lactate[16].
Une fois à l'intérieur de la bactérie, le citrate est scindé en acétate et oxaloacétate qui est lui-même décarboxylé en pyruvate[4] et dioxyde de carbone CO2. Les deux enzymes qui contrôlent ces réactions sont codées par des gènes chromosomiques (resp. sitE et sitF) constitutives des souches de L. lactis subsp. lactis biovar diacetylactis.
L'α-acétolactate est formé à partir de deux molécules de pyruvate. Cette molécule instable est décarboxylée en acétoïne (ou en diacétyle par voie chimique). Enfin, l'acétoïne est réduit en 2,3-butanediol.
Le 2,3-butanediol est responsable de l'arôme beurré, de l'odeur de crème et de l'arôme de noisette. Par contre, l'acétoïne qui a été décrit comme porteur de l'arôme de crème et d'amande, semble avoir peu d'intérêt olfactif direct[17]. Une étude d'une collection de 36 souches domestiquées ou environnementales de L. lactis quant à la production de diacétyle ou d'acétoïne, a montré que les souches domestiquées appartenant au biovar diacetylactis étaient celles capables de produire de grandes quantités de diacétyle/acétoïne durant le début de leur croissance. Chez ces souches, la consommation de citrate a été identifiée comme le déterminant majeur de la production d'arômes[18].
Le système protéolytique
Pour leur croissance, les Lactococcus lactis ont besoin d'acides aminés. La plupart des souches requièrent de l'isoleucine, leucine, histidine, méthionine et valine. Mais la concentration de ces derniers est très faible dans le lait[19] et ne peut assurer qu'une part très limitée de la croissance. Un moyen pour obtenir suffisamment d'acides aminés est de recourir à la décomposition des protéines par la voie enzymatique. Ainsi, L. lactis utilise la caséine, la protéine la plus abondante du lait, comme source principale d'acides aminés nécessaires à sa croissance.
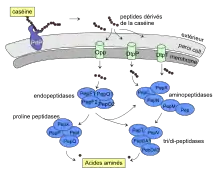
La machinerie protéolytique du lactocoque est composée :
- d'une protéase extracellulaire ancrée à la paroi (PrtP),
- de trois systèmes de transport de peptides
- d'un grand nombre de peptidases intracellulaires.
La protéase de paroi PrtP, fixée à l'extérieur de la cellule, est une enzyme à sérine qui hydrolyse les caséines en oligopeptides. Ceux-ci sont ensuite transportés à l'intérieur de la cellule par trois transporteurs. Les peptides sont alors hydrolysés par plusieurs peptidases[20] en acides aminés. En 2008, il y avait 41 peptidases connues ou potentielles chez L. lactis enregistrées dans les bases de données comme NCBI. Les peptides sont cassés de diverses manières par ces différentes peptidases.
Les lactocoques jouent un rôle essentiel dans la transformation du lait en fromage. Nous avons vu que par la production d'acide lactique, ils contribuent à déstructurer les micelles de caséines et à provoquer leur coagulation. Une fois le caillé égoutté, celui-ci subit une phase d'affinage qui correspond à une digestion enzymatique. La protéolyse est le phénomène dominant de l'affinage. Et à nouveau les lactocoques peuvent jouer un rôle important en raison de leur système protéolytique. Les peptidases intracellulaires peuvent être libérées dans le caillé à la suite de la lyse cellulaire. Elles deviennent alors les principales responsables de la formation de petits peptides et d'acides aminés libres dans le fromage[15].
Écosystèmes du lait et des fromages
Les lactocoques lactiques jouent un rôle essentiel dans la production de laits fermentés et de fromages. Ils peuvent être présents en tant que :
- bactéries lactiques sauvages, éléments naturels dans la flore du lait, apportées par les trayons, le matériel de traite et de stockage du lait et l'atelier de fabrication ou
- ensemencées volontairement dans le lait.
- Le lait cru
L'amélioration de l'hygiène dans les pratiques de la traite et du stockage du lait a permis de réduire considérablement la flore microbienne indésirable des laits crus (actuellement en France environ 10 000 germes totaux ml−1). Plus de 200 espèces microbiennes ont été décrites dans les laits[21]. Parmi celles-ci, certaines sont dangereuses pour la santé du consommateur[N 4] et d'autres sont utiles à l'acidification des caillés et au développement des caractéristiques organoleptiques des fromages. Lactococcus lactis fait partie bien sûr de cette flore utile, dite d'intérêt technologique.
À l'intérieur de la partie supérieure de la mamelle, le lait est stérile. Pour comprendre la source des milliers de micro-organismes qui colonisent le lait, Anne-Cécile Normand[2] - [22] a mené une étude du cheminement des micro-organismes depuis l'étable jusqu'au lait. Elle a pu établir que la majorité des espèces bactériennes recensées dans le lait avait pour origine l'environnement de la salle de traite (air, nourriture utilisée pendant la traite, trayons).

D'après une étude microbiologique du lait de vaches, faite sur 27 troupeaux dans la région de production du camembert de Normandie, durant les six premiers mois de l'année (Desmasures et als[23], 1997), il ressort que les lactocoques (760 cfu ml−1) sont les plus nombreux après les Pseudomonas (2 000 cfu ml−1), suivi par les Micrococcaceae (720 cfu ml−1). Les bactéries lactiques étaient généralement bien représentées dans tous les échantillons, avec un rapport lactocoques/germes totaux d'environ 10 %, soit un ratio bien moins élevé que dans les laits contaminés.
Cette étude pionnière, de la flore globale du lait cru destiné à la fabrication de fromage, éclaire l'apport des micro-organismes du lait aux caractéristiques organoleptiques des fromages. Les études qui ont suivi ont confirmé ces résultats[24]. Les micro-organismes habituellement trouvés dans les laits crus de vache sont des staphylocoques et des bactéries corynéformes (100 - 1 000 UFC ml−1), les bactéries à Gram négatif (Entérobactéries et Pseudomonas sp.) (100 - 1 000 UFC ml−1), puis viennent cinq groupes à concentration moyenne semblable (10 - 100 UFC ml−1) : Lactocoques, Lactobacilles, Leuconostoques, Entérocoques, et les Levures. La suppression de la microflore par microfiltration ou pasteurisation du lait, entraine une perte de goût et une diminution de la richesse du goût du fromage. Sur le cantal, les écarts de profils aromatiques entre les fromages au lait cru et ceux au lait pasteurisé, ont pu être corrélés à des différences de niveaux de composés aromatiques mesurés dans la pâte[25]. Sur les trois paramètres étudiés jouant un rôle sur l'arôme du cantal, on a découvert que la pasteurisation avait un effet négatif plus important que l'alimentation des vaches ou la durée d'affinage. Les « olfactogrammes » des fromages au lait cru ont été globalement plus riches en intensité et en nombre de pics, avec notamment une proportion élevée d’acide butyrique.
Une étude de Verdier-Metz et als[26] 2004, a montré l'intérêt de préserver des laits à des niveaux compris entre 15 000 et 30 000 germes totaux ml−1 parce qu'ils présentent un indice de diversité génétique plus élevé que les laits ultra-propres à moins de 5 000 germes totaux ml−1. Les laits ultra-propres (dits pauci-microbiens) ont une acidification naturelle plus lente que les laits plus chargés.
D'après une étude des flores bactériennes du lait de chèvre, menée auprès de 38 fermes d'élevage caprin, productrices de Pélardon et Rocamadour AOP[11] dans les régions du Languedoc-Roussillon et Midi-Pyrénées, il ressort que le groupe dominant de micro-organismes (cultivé sur agar Elliker) est constitué de bactéries lactiques (173 colonies sur 177) avec principalement L. lactis et des entérocoques (161 colonies sur 173). La sous-espèce L. lactis subsp. cremoris semble spécifiques de seulement deux propriétés. D'autres études[24] donnent en moyenne 100 - 1 000 lactocoques ml−1 pour le lait de chèvre et 10 000 lactocoques ml−1 pour le lait de brebis.
- Le fromage
La microflore du lait est la première à intervenir dans la fabrication du fromage. C'est pourquoi on retrouve dans les fromages les Lactococcus, Lactobacillus, Enterococcus, Leuconostoc, Pediococcus et Streptococcus. La fonction de ces bactéries est de dégrader le lactose en acide lactique et de former les composés d'arôme du fromage.

Du fait de ces propriétés, les Lactococcus lactis sont utilisés dans la plupart des levains laitiers mésophiles[15] souvent associés à Leuconostoc mesenteroides subsp. cremoris. Ces levains mésophiles sont en général employés[27] pour la fabrication des fromages dont la température du caillé ne dépasse pas 40 °C soit principalement les fromages à pâte fraiche et à pâte molle. Ainsi, pour faire du fromage blanc avec un lait ayant subi des traitements thermiques ou chimiques, il faut ensemencer avec des souches Lactococcus lactis. Avec les fromages à pâte molle comme le camembert AOC de Normandie qui est produit avec du lait de vache n'ayant subi aucun traitement thermique[28], les lactocoques sont déjà présents dans le lait. Durant la phase de maturation primaire, ces lactocoques se développent significativement, contrairement aux lactobacilles et aux levures. Ils sont ensuite consolidés par l'ajout massif de levains lactocoques. Finalement, la flore lactique du camembert est dominée par la sous-espèce Lactococcus lactis subsp. lactis au plan phénotipique[N 5].
Les lactocoques lactiques sont aussi couramment rencontrés[29] dans les fromages à pâte pressée non cuite (salers, saint-nectaire, castelmagno) ou semi cuite (fontina) et les fromages à pâte persillée (stilton, gorgonzola, cabrales).
Il a été observé que lors de la fabrication d'un fromage saint-paulin utilisant un levain de lactocoques, la conversion d'acides aminés en composés d'arômes s'obtenait grâce à une aminotransférase aromatique (AraT) de L. lactis subsp. cremosis NCDO763[30]. Les aminotransférases (ou transaminases) catalysent une réaction entre un acide aminé et le acide α-cétonique correspondant. L'activité de la transaminase AraT conduit à la production[31] d'isovalérate (arôme de fromage affiné), de 2-méthylbutanal (arôme fruité, malté), de benzaldéhyde (arôme d'amande amère) et de composés soufrés comme le méthanethiol (arôme de chou). Une autre transaminase BcaT intervenant dans la dégradation des acides aminés branchés, a été purifiée dans la même souche de cremosis[32]. Elle conduit à la production d'isobutyrate, d'isovalérate, d'aldéhydes et d'alcools[27].
La technologie principale pour la fabrication de fromages de chèvre fermiers ou de brebis sous AOP[11] (comme rocamadour, pélardon, picodon, selles-sur-cher, sainte-maure-de-touraine, crottin de Chavignol…) consiste à associer présure et ensemencement par des bactéries lactiques pour provoquer la coagulation. La déstabilisation des caséines est initiée par l'ajout de présure mais à une dose insuffisante pour obtenir un caillé ferme. C'est ensuite l'acidification produite par l'ajout de bactéries comme Lactococcus lactis, associées aux bactéries indigènes du lait cru qui permet la formation du coagulum. L'ensemencement par les lactocoques se fait soit en utilisant des ferments lactiques industriels soit en utilisant le lactosérum prélevé au moment du moulage de la fabrication précédente. Le lactosérum, composé d'un ensemble de souches spécifiques d'une exploitation, contient majoritairement des L. lactis[11] et contribue à la spécificité des fromages fermiers.
Voir aussi
Notes
- 1873 d'après The Taxonomicon et 1878 pour Pot B, Ludwig W, Kersters K, Schleifer KH, Taxonomy of lactic acid bacteria In Bacteriocins of Lactic Acid Bacteria, Edited by de Vuyst L, Vandamme EJ. New York: Blackie Academic & Professional; 1994:13-89
- ou voie d'Embden-Meyerhof-Parnas
- une homofermentation qui convertit plus de 90 % du pyruvate en lactate est dite homolactique
- comme Campylobacter jejuni, certains Escherichia coli, Listeria monocytogenes, Staphylococcus aureus, Salmonella, Yersinia enterocolitica…
- les lactocoques atteignent 109 ufc g−1 au cours de l'affinage (Desmasures, 1995)
Références
- Taxonomicon
- Anne-Cécile Normand, Étude des flux microbiens dans les étables de production laitière de Franche-Comté et de Bavière. Intérêts dans les domaines de l'asthme et de l'allergie., Thèse : Université de Franche-Comté, Besançon,
- Susan Mills, « Plasmids of lactococci - genetic accessories or genetic necessities? », FEMS microbiology reviews, vol. 30, no 2, , p. 243-273 (ISSN 0168-6445, DOI 10.1111/j.1574-6976.2005.00011.x)
- (sous la dir.) Djamel DRIDER, Hervé Prévost, Bactéries lactiques, Physiologie, métabolisme, génomique et applications industrielles, ECONOMICA,
- Sandine W.E., « New nomenclature of the non-rod-shaped lactic acid bacteria. », Biochimie, vol. 70,
- Tamara Aleksandrzak-Piekarczyk, Lactic Acid Bacteria : R & D for Food, Health and Livestock Purposes, InTech, , 672 p. (ISBN 978-953-51-0955-6, lire en ligne), « Lactose and β-Glucosides Metabolism and Its Regulation in Lactococcus lactis: A Review »
- Johan E T van Hylckama Vlieg, « Natural diversity and adaptive responses of Lactococcus lactis », Current opinion in biotechnology, vol. 17, no 2, , p. 183-190 (ISSN 0958-1669, DOI 10.1016/j.copbio.2006.02.007)
- William J Kelly, « Chromosomal diversity in Lactococcus lactis and the origin of dairy starter cultures », Genome biology and evolution, vol. 2, , p. 729-744 (ISSN 1759-6653, DOI 10.1093/gbe/evq056)
- Alexander Bolotin, « The Complete Genome Sequence of the Lactic Acid Bacterium Lactococcus lactis ssp. lactis IL1403 », Genome Research, vol. 11, no 5, , p. 731-753 (ISSN 1088-9051 et 1549-5469, DOI 10.1101/gr.169701, lire en ligne, consulté le )
- Maxim Moisan, Système de recombineering pour les bactériophages infectant Lactococcus lactis, thèse Université de Laval, (lire en ligne)
- Hélène Tormo, DIVERSITE DES FLORES MICROBIENNES DES LAITS CRUS DE CHEVRE ETFACTEURS DE VARIABILITE, thèse : Université de Toulouse,
- Corroler David, Biodiversité des lactocoques sauvages au sein de la zone d’appellation d’origine « Camembert de Normandie » : implication au cours de la transformation fromagère., thèse: Université de Caen Basse-Normandie,
- Sandy Raynaud, Régulation métabolique et transcriptionnelle de l'autoacidification chez Lactococcus lactis, Thèse de doctorat, Université Paul Sabatier, Toulouse, , 309 p.
- Reizer, J. et Peterkofsky A., Sugar Transport and Metabolism in Gram-positive Bacteria, Ellis Horwood Limited, Chichester., , chap. 1 (« Sugar transport in the lactic acid bacteria. »)
- Patrick F. Fox, Cheese : Chemistry, Physics and Microbiology : Vol 1, General Aspects, Academic Press, , 3e éd. (ISBN 0-12-263652-X)
- Christian Magni, « Mechanism of Citrate Metabolism in Lactococcus lactis: Resistance against Lactate Toxicity at Low pH », Journal of Bacteriology, vol. 181, no 5, , p. 1451-1457 (ISSN 0021-9193, lire en ligne, consulté le )
- Biogenese des aromes dans les fromages
- Delphine Passerini, « New insights into Lactococcus lactis diacetyl- and acetoin-producing strains isolated from diverse origins », International journal of food microbiology, vol. 160, no 3, , p. 329-336 (ISSN 1879-3460, DOI 10.1016/j.ijfoodmicro.2012.10.023)
- Sandra Helinck, « The effects of adding lactococcal proteinase on the growth rate of Lactococcus lactis in milk depend on the type of enzyme. », Applied and Environmental Microbiology, vol. 63, no 6, , p. 2124-2130 (ISSN 0099-2240, lire en ligne)
- Liliana López Kleine, DETERMINATION DU RÔLE DE CERTAINES PEPTIDASES BACTERIENNES PAR INFERENCE A PARTIR DE DONNEES HETEROGENES ET INCOMPLETES, thèse : L’Institut des Sciences et Industries du Vivant et de l’Environnement (Agro Paris Tech),
- MONTEL M.C., BOUTON Y., PARGUEL P., « Ecosystèmes des laits et des fromages au lait cru – enjeux pour leur maîtrise », Renc. Rech. Ruminants, no 19, (lire en ligne)
- Bouton Y, Guyot P, Vacheyrou M, Normand AC, Piarroux R, Beuvier E., « Étude des flux bactériens dans les étables de production laitière de Franche- Comté. », 15e colloque du Club des Bactéries Lactiques, 13-15 novembre,
- N Desmasures, « Microbiological composition of raw milk from selected farms in the Camembert region of Normandy », Journal of applied microbiology, vol. 83, no 1, , p. 53-58 (ISSN 1364-5072)
- (coordonné par) Cécile Laithier, Microflore du lait cru, Vers une meilleure connaissance des écosystèmes microbiens du lait et de leurs facteurs de variation, CNAOL, (lire en ligne)
- CORNU A., RABIAU N., PRADEL P., VERDIER-METZ I. , KONDJOYAN N., TOURNAYRE P., BERDAGUE J.L., MARTIN B., « Contribution de la pasteurisation, de la durée d’affinage et de l’alimentation des vaches à la formation des composés d’arôme de fromages de type Cantal », Renc. Rech. Ruminants, vol. 14, (lire en ligne)
- Verdier- Metz I., Michel V., Delbès C., Montel MC, « Analysis by SSCP method of the microbial diversity of milk flora », 6th International Meeting of Mountain Cheese. Ragusa, vol. 1 & 2 juin,
- Orianne Cholet, Étude de l'écosystème fromager par une approche biochimique et moléculaire, Thèse : Institut National Agronomique Paris-Grignon,
- Claude Béranger, Les Fermentations au service des produits de terroir, Paris, Institut National de la Recherche Agronomique, , 296 p. (ISBN 2-7380-1203-5 et 9782738012036)
- Emilie Retureau, Reconstitution de communautés microbiennes complexes pour l’inhibition de Listeria monocytogenes à la surface de fromages à pâte pressée non cuite, Thèse : Université Blaise Pascal, Clermont-Ferrand,
- L Rijnen, « Inactivation of lactococcal aromatic aminotransferase prevents the formation of floral aroma compounds from aromatic amino acids in semi-hard cheese », International Dairy Journal, vol. 9, no 12, , p. 877-885 (ISSN 0958-6946, DOI 10.1016/S0958-6946(00)00009-1, lire en ligne, consulté le )
- Rijnen, L., Yvon, M., van Kranenburg, R., Courtin, P., Verheul, A., Chambellon, E. & Smit, G, « Lactococcal aminotransferases AraT and BcaT are key enzymes for the formation of aroma compounds from amino acids in cheese », International Dairy Journal, vol. 13,
- Yvon, M., Chambellon, E., Bolotin, A. & Roudot-Algaron, F., « Characterization and role of the branched-chain aminotransferase (Bca T) isolated from Lactococcus lactis subsp. cremoris NCDO763. », Applied and Environmental Microbiology, vol. 66,
Bibliographie
- Thi-Ly Tran (2013) Unraveling the muco-adhesion of Lactococcus lactis : development of biophysical approaches. Biochemistry, Molecular Biology. Thèse de doctorat, INSA de Toulouse, soutenue le .<NNT :2013ISAT0029>.<tel-01127035> (en), résumé en français p. 11