Cycle de réplication du SARS-CoV-2
Le cycle de réplication du SARS-CoV-2 se traduit par (1) l’entrée du virus à l’intérieur de la cellule, (2) le détournement de la machinerie cellulaire par le virus, (3) la réplication du virus par la cellule et/ou la réalisation de taches bien précises par la cellule répondant aux besoins du virus.
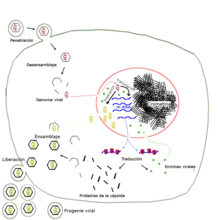
Cycle de réplication
Récepteurs
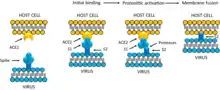
Pour infecter une cellule, le SARS-CoV-2 utilise (1) des récepteurs cellulaires lui permettant de se lier à la cellule, (2) des cofacteurs lui permettant de mieux s’agripper à la cellule, et (3) des protéases lui permettant de cliver sa protéine S et d’activer ainsi la fusion avec la cellule.
Deux cofacteurs sont suggérés pour le SARS-CoV-2, il s'agit de :
- l'héparane sulfate, un polysaccharide, connu pour être le cofacteur du HCoV-NL63 qui a également ACE2 comme récepteur cellulaire[1] ;
- la neuropiline 1 (NRP1), une protéine transmembranaire, qui a un rôle important dans l'angiogenèse, la progression tumorale ou encore dans le transport axonal[2]. Dans des autopsies, le SRAS-CoV-2 a été détecté dans des cellules qui exprimaient NRP1 mais où ACE2 était à peine détectable. Il s’agissait de cellules olfactives des voies nasales, du bulbe olfactif et des voies olfactives du cerveau. NRP1 est connue pour être le cofacteur des virus EBV et HTLV-1. Dans le cas du SARS-CoV-2, il est suggéré que NRP1 pourrait être soit un cofacteur, soit un récepteur cellulaire à part entière comme ACE2[3] - [4] - [5].
Principaux
Via sa protéine S, le SARS-CoV-2 peut se lier à plusieurs récepteurs pour infecter des cellules :
- ACE2, l'enzyme de conversion de l'angiotensine 2, a été le premier récepteur cellulaire identifié pour le SARS-CoV-2, dans la mesure où ce récepteur était le plus connu du SARS-CoV-1[6] - [7].
- L-SIGN (en), appelée également CD209L, est une lectine de type-C, c’est-à-dire une protéine dépendante du calcium qui se lie spécifiquement et de façon réversible à certains glucides. Comme récepteur transmembranaire de cellules, L-SIGN est exprimé principalement dans le foie, la rate, les ganglions lymphatiques et dans une moindre mesure dans le tissu pulmonaire humain sur les cellules alvéolaires de type II. L-SIGN peut interagir avec le récepteur ICAM3 (en) exprimé par certains globules blancs. L-SIGN avait déjà été identifié comme récepteur pour le HCoV-E229 et le SARS-CoV-1, il a aussi été identifié comme récepteur du SARS-CoV-2[8] - [9]. L-SIGN est également un récepteur pour d’autres virus : la grippe, ebola, l'hépatite C, HIV, cytomegalovirus, le virus du Nil occidental, le virus de l'encéphalite japonaise ou encore le virus Marburg[10] - [11] ;
- DC-SIGN, appelée également CD209, est une lectine de type-C présentant 77 % d’homologie avec L-SIGN[12] qui interagit elle aussi avec le SARS-CoV-2[8] - [9]. DC-SIGN est surtout exprimée par les cellules dendritiques, dans les tissus lymphoïdes, les ganglions lymphatiques et la rate[10]. La reconnaissance du récepteur DC-SIGN par le SARS-CoV-2 lui donne la possibilité d'infecter ou de se lier aux cellules dendritiques. Il avait déjà été suggéré que la protéine S du SARS-CoV-1 se liait à DC-SIGN, mais sans pouvoir les infecter[13]. D’autres virus ont aussi cette faculté : la grippe A, le HIV, ebola, cytomegalovirus, l’hépatite C, la dengue, la rougeole, le virus de l’encéphalite japonaise, le virus de Lassa, le virus respiratoire syncytial, le virus de la fièvre de la vallée du Rift, le virus Uukuniemi ou encore le virus du Nil occidental[10].
Complémentaires
D'autres récepteurs pour infecter des cellules sont proposés :
- les récepteurs Fc sont des protéines transmembranaires présentes à la surface des globules blancs. Les récepteurs Fc se lient aux anticorps qui ont opsonisé (c'est-à-dire « enrobé ») les pathogènes. Le pathogène peut être absorbé par le globule blanc par phagocytose ou endocytose. Lorsque les anticorps deviennent non neutralisants, des virus tels que les coronavirus utilisent les récepteurs Fc pour infecter les globules blancs, par un mécanisme connu sous le nom de facilitation dépendante des anticorps[14]. Chez le coronavirus du chat, un des rares coronavirus capable de se reproduire activement dans un globule blanc (les macrophages), ce phénomène induit une péritonite infectieuse. D’autres virus qui développent une résistance aux anticorps, détournent les récepteurs Fc avec plus ou moins de gravité : la grippe, les flavivirus (la dengue, le virus de la fièvre jaune, le virus Zika…), le VIH, la rougeole, le virus respiratoire syncytial…[15] - [16] ;
- CD147 ? Appelé également la basigine, le récepteur CD147 est une hypothétique voie d’infection des cellules par le SARS-CoV-2[17]. CD147 est une protéine transmembranaire de la superfamille des immunoglobulines[18]. CD147 est un récepteur à la surface des globules rouges qui peut être détourné par Plasmodium falciparum, le parasite qui cause le paludisme humain[19] - [20]. Le récepteur CD147 est aussi exprimé par les lymphocytes[17]. L'infection de cellules via le récepteur CD147 avait déjà été proposée avec le SARS-CoV-1[21]. Il semble que la protéine S ne soit pas la clé permettant au SARS-CoV-2 de se lier au récepteur CD147[22]. Avec le SARS-CoV-1, l'interaction proposée entre CD147 et le virion était via la protéine N, de manière indirecte : la protéine N se fixerait à une protéine intermédiaire, la cyclophiline A (CyPA), qui elle se lierait à CD147[23] ;
- Mac-1 (en) et LFA-1 ? La protéine ORF7a du SARS-CoV-2 pourrait être davantage une protéine structurale qu’une protéine accessoire. Au même titre que les protéines S, M, E et N, la protéine ORF7a est partie intégrante des virions. ORF7a est capable de se lier aux globules blancs, notamment aux monocytes/macrophages via le récepteur Mac-1 (en)[Note 1] et aux lymphocytes via le récepteur LFA-1[Note 2]. Son affinité avec les monocytes CD14 + est très forte, tandis qu’elle est moindre avec les lymphocytes B et T. L’interaction d’ORF7a avec les monocytes engendre la sécrétion de cytokines pro-inflammatoires : IL-6, IL-1b, IL-8 et TNF-a[24] - [25].
Protéine S : liaison et fusion
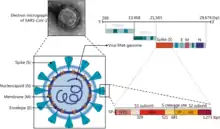

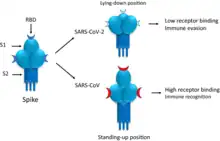
La protéine S (dites protéine spike ou protéine spicule) du SARS-CoV-2 forme des péplomères. Elle est constituée de deux sous-unités fonctionnelles : la sous-unité S1 permet la liaison du virus au récepteur de la cellule hôte et la sous-unité S2 assure la fusion de l’enveloppe virale avec la membrane cellulaire[26].
La sous-unité S1 contient un CTD et un NTD :
- le C-terminal domain (CTD) se décompose en 3 parties :
- le receptor binding domain (RBD), appelé également CTD1, qui se lie au récepteur ACE2.
- le subdomain 1 (SD1), appelé également CTD2.
- le subdomain 2 (SD2), appelé également CTD3[27] - [28] - [29].
Le SD2 héberge un C-end terminal rule (CendR), qui une fois libéré après le clivage de S1 et S2, peut se lier à la neuropiline 1[2]. Il est suggéré que la neuropiline 1 facilite le transport axonal du SARS-CoV-2[2]. En d'autres termes, la neuropiline 1 permettrait au SARS-CoV-2 de voyager via les neurones et faciliterait l'infection de nombreux organes. Les SD1 et du SD2 pourraient également aider le RBD à alterner entre un état visible et masqué (‘up’ and 'down' state conformations)[30]. Certains anticorps ciblant le SD2, en particulier ceux enrobant la séquence d'acides aminés LYQDVNC localisée entre les codons 611 et 617 du SD2 de la protéine S, seraient susceptibles de faciliter l'infection des globules blancs par le SARS-CoV-2[14].
- le N-terminal domain (NTD) se lie à L-SIGN (en) ou DC-SIGN (en). Cette affinité est significativement plus faible qu'avec ACE2[10] - [31] ;
Une fois que la sous-unité S1 est liée à un récepteur, généralement ACE2, la sous-unité S2 organise la fusion des membranes. Un peptide de fusion ou Fusion Peptide (FP) de S2 s’accroche à la membrane de la cellule cible. L'autre extrémité du S2 est le transmembrane domain (TM) et le cytoplasmic tail (CT) qui agissent également comme un point d'accroche mais sur le virion. À partir de ces deux points d'accroche (le FP et le TM-CT), le heptad repeat 1 (HR1) avec le central helix (CH) du S2 forment une superhélice trimérique parallèle sur laquelle s'enroule le heptad repeat 2 (HR2) en formant un trimère d'épingles à cheveux à six hélices, ce qui provoque la fusion des deux membranes. Enfin en faisant intervenir le connector domain (CD), le virus peut pénétrer dans la cellule cible[32]. Ce cas de figure est l’infection par fusion directe.
Outre la fusion directe, l’infection d’une cellule par le SARS-CoV-2 pourrait se faire par endocytose[33]. L'endocytose est un processus au cours duquel la membrane d'une cellule enveloppe et absorbe une particule. Autrement dit une cellule engloutit un virion dès lors qu'il est lié à un récepteur ACE2. Lors d’une infection par endocytose, la protéine S du virion absorbé se clive en S1 et S2 à l'aide d'une enzyme de la cellule qui est le plus souvent Cathepsin B (en) ou Cathepsin L (en)[34]. La chloroquine a montré in vitro qu'elle pouvait inhiber l’infection par endocytose mais pas celle par fusion directe. En s’accumulant dans les endosomes et lysosomes des cellules, la chloroquine augmente le pH dans ces organelles, altérant ainsi leur fonctionnement essentiel pour l’endocytose des coronavirus[35] - [33]. L’infection d'une cellule via l'hypothétique récepteur CD147 se ferait uniquement par endocytose[17]. L’infection via ACE2 et par fusion directe est très probablement le principal mode d’infection du SARS-CoV-2.
Pour infecter une cellule, deux clivages protéolytiques successifs sont nécessaires. Le premier clivage génère S1 et S2. Le second appelé S2' libère l'extrémité du peptide de fusion (FP). Une des originalités de la protéine S du SARS-CoV-2 est qu’elle intègre une séquence d'activation au site de clivage dit « S1 / S2 », similaire aux séquences observées dans les virus de grippe. Ainsi, à l’instar du virus de la grippe, la protéine S du SARS-CoV-2 peut être clivée à l’aide d’une protéase qui se trouve en abondance dans le plasma : la furine. Le SARS-CoV-1 en était incapable. Les scientifiques suggèrent que cette capacité à pouvoir cliver S1 et S2 par la furine est la raison pour laquelle le SARS-CoV-2 est devenu une épidémie[36]. En revanche d'après le virologue du CNRS Étienne Decroly, la protéase TMPRSS2 connue pour être capable de cliver la protéine S du SARS-CoV-1, est présentée à tort par la littérature comme capable de cliver S1/S2 ou S2' du SARS-CoV-2[33]. TMPRSS2 facilite bien l'infection de cellules par le SARS-CoV-2 mais surtout dans le cadre de la formation de syncytia[37]. Enfin le clivage de S2' peut se faire aussi bien par la furine que par Cathepsin B (en) et probablement par d'autres protéases[33].
Syncytia
Dans des organes tels que les poumons, les reins ou le foie, les cellules épithéliales sont étroitement compactées, l’espace extracellulaire est très limité. Le SARS-CoV-2 y tire un avantage. Les cellules infectées peuvent fusionner avec les cellules non infectées avoisinantes, formant des « syncytia », c’est-à-dire des cellules géantes englobant des dizaines de cellules productrices de virus[37]. Ce processus est médié par la protéine S nouvellement synthétisée qui s'accumule à la surface de la cellule infectée. Mais la formation de syncytia peut se faire aussi par « fusion from without (FFWO) », c’est-à-dire par des cellules infectées qui n’ont pas encore répliqué la protéine S[38].
Ce mode d’infection permet au SARS-CoV-2 d’infecter vite un grand nombre de cellules avec une faible quantité de protéines S. Dans l’univers des coronavirus, ce mode d’infection a été identifié chez le HCoV-229E[39] ou encore le virus de l'hépatite murine[40] qui est le pathogène le plus important et le plus étudié des souris de laboratoire. D’autres virus ont également cette faculté d’infecter via la formation de syncytia : le virus de la rougeole, le VIH et le cytomégalovirus. La rougeole est capable de former des syncytia dans le cerveau, pouvant causer une encéphalite[38].
Lorsque le SARS-CoV-2 infecte de nouvelles cellules au sein d’un syncytium, les anticorps se révèlent d’une faible efficacité[38]. En revanche, IFITM (en), une protéine transmembranaire produite en présence d'interférons, peut bloquer cette fusion. Mais son effet peut être contrecarré par TMPRSS2 qui facilite la formation de syncytia[37].
Réplication du génome viral
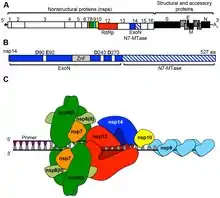
Préalablement à la multiplication du génome viral, dans la phase la plus précoce de la traduction, seul le gène ORF1ab est traduit par les ribosomes en deux polyprotéines appelées Pp1a et Pp1ab. Pp1ab est divisée en 16 protéines non structurales (nsp1 à nsp16). Pp1a est identique à Pp1ab mais est tronquée : elle ne comporte que les 11 premières protéines non structurales (nsp1 à nsp11)[41]. Chacune des protéines nsp possède une activité précise[42] - [43] :
- pour activer les protéines non structurales, Pp1a et Pp1ab ont chacune deux protéases : PLpro et 3CLpro[44]. PLpro se trouve en position nsp3 et 3CLpro en position nsp5. Après s'être auto-clivées (auto-libérées), PLpro clive nsp1 et nsp2, tandis que 3CLpro clive toutes les autres protéines non structurales[45] ;
- de nombreuses protéines non structurales s'assemblent alors pour former le Complexe Réplicase-Transcriptase (CRT) indispensable à la réplication en série du génome viral. Au sein du CRT, nsp12 est centrale. Nsp12 est une ARN polymérase ARN-dépendante nommée réplicase (RdRp), dont la première action est de synthétiser un brin d'ARN négatif, c'est-à-dire une copie à l'envers de la totalité du génome du virus, appelée antigénome[44]. À partir de cet antigénome, Nsp12 reproduit en série des brins d'ARN positifs, autrement dit des reproductions dans le bon sens du génome viral. En amont du CRT, nsp7 et nsp8 forment une pince coulissante ARN qui permet de dérouler l'ARN viral à recopier, sous l'impulsion de nsp13, une hélicase qui se trouve en aval du complexe. En aval du CRT, nsp14 avec l'aide de nsp10, relit les copies et corrige les erreurs de nsp12. Nsp16 joue un rôle important avec l’aide de nsp14 dans la formation de la coiffe des ARNs à son extrémité 5'. Nsp15 pourrait avoir un rôle de 'nettoyeur' dans la mesure où il est capable de dégrader les débris d'ARN. Enfin nsp9 est la protéine de liaison à l'ARN en sortie du complexe[46] - [47] - [48] ;
- afin de protéger le Complexe Réplicase-Transcriptase d’éventuels mécanismes de défense cellulaires comme l'autophagie, nsp3, nsp4 et nsp6 réorganisent les différentes membranes internes de la cellule infectée en un réseau interconnecté de vésicules à double membrane (double-membrane vesicle - DMV). Les ARN viraux répliqués sont conservés dans ces DMV en attendant d'être encapsulés dans de nouveaux virions[49] - [50].
Après avoir synthétisé un antigénome, et avant de répliquer en série le génome viral, nsp12 produit des brins d'ARN viraux complémentaires dit subgénomiques qui serviront à la mise en production des 9 protéines accessoires (10, 9c, 9b, 8, 7b, 7a, 6, 3b et 3a) et des quatre protéines structurales (N, M, E et S) par les ribosomes[44]. Ces fractions du génome viral produites par nsp12 sont de taille variable mais comprennent toutes l'extrémité 3' du génome du SARS-CoV-2 et terminent toutes par l'ORF10. Sauf exception, seul l'ORF placé en tête au 5'terminal, est traduit[51]. À titre d'exemple, un brin d'ARN subgénomique qui permet de synthétiser l'ORF2 codant la protéine S, incorpore tous les autres ORF suivants liés aux protéines structurales et accessoires, mais les ribosomes ne synthétisent que la protéine S.
Activité anti-interférons
Lorsque des cellules sont infectées par un pathogène, elles produisent des interférons (IFN), qui ont des propriétés antivirales. Les IFN sont des glycoprotéines de la famille des cytokines. Ce sont des molécules de signalisation qui permettent d'alerter les cellules voisines qu'une infection est en cours[52]. Les IFN sont classés en 3 types. Les IFN de types I et III (en) jouent un rôle prépondérant dans l’immunité innée antivirale dans la plupart des cellules de l’organisme. Les IFN de type I sont présents dans la quasi-totalité des cellules. Ceux de type III sont plutôt produits dans les muqueuses, lesquelles sont particulièrement exposées aux infections virales[53]. Lorsque des interférons de type I sont produits, ils induisent, dans les cellules infectées et les cellules avoisinantes, l’expression de centaines de gènes (interferon-stimulated gene (en), ISG), ce qui va permettre l’établissement d’un état antiviral[52].
Dans les cellules, les interférons sont activés par les récepteurs de reconnaissance de motifs moléculaires (pattern recognition receptors, PRR), des protéines « sentinelles » en charge de reconnaître des motifs moléculaires associés aux pathogènes (pathogen-associated molecular patterns, PAMP), c’est-à-dire de détecter la trace de pathogènes. L’activation des interférons se fait par une cascade de signalisation. Les PRR activent le plus souvent IRF3, une protéine de régulation des interférons-béta qui « dort » dans le cytoplasme des cellules. Lorsqu’elle est activée par un PRR, la protéine IRF3 entre dans le noyau de la cellule où elle stimule le gène codant l'IFN-béta[52]. La synthèse d’IFN-béta par les ribosomes est un préalable à l'activation du facteur de transcription IRF7 (en) qui active le gène des IFN-alpha[52]. Lorsque les interférons sont synthétisés, l’activité de la cellule est modifiée et est désormais consacrée en grande partie à sa défense. La réponse antivirale interféron est diversifiée dans ses cibles et a une amplitude considérable[53].
Face au SARS-CoV-2, les PRR censés activer les IRF3 sont soit des récepteurs de type Toll (toll-like receptors, TLR), soit des RIG-I-like receptors (en) (RLR)[53] :
- les TLR 7 et 8 sont spécialisés dans la reconnaissance d’ARN simple-brin viral comme celui du SARS-CoV-2. Ces TLR7 et 8 sont capables de détecter le SARS-CoV-2 s’il pénètre par endocytose. Or le SARS-CoV-2 infecte le plus souvent via une fusion directe ;
- des RLR, notamment RIG-I et MDA5, sont aussi en mesure de détecter la présence d’ARN viraux dans le cytosol de la cellule.
Phase précoce
Lorsque le PRR RIG-I détecte la présence d’un ARN viral, il enclenche une cascade de signalisation. RIG-I active la protéine TRIM25 (en) qui elle-même interagit avec la protéine MAVS (en) qui elle-même initie TBK1 et IKK-epsilon (en), conduisant à l’activation d’IRF3 et in fine à la production d'interférons béta[21]. Or lorsque le SARS-CoV-2 infecte une cellule par fusion directe, le génome viral s’introduit avec la protéine N dans la cellule. Une fois dans la cellule, la protéine N pourrait se lier à TRIM25, et bloquer ainsi la signalisation de RIG-1 et par conséquent la production d’interféron béta[54] - [35].
Dès qu'elle est synthétisée, nsp3, la plus grande des protéines non structurales du SARS-CoV-2, participe également à cette activité précoce anti-interféron par des modifications post-traductionnelles. Nsp3 est une protéine clivante, ce qui se traduit concrètement[41] :
- par la de-MARylation de PARP14 (en), une enzyme monoADP-ribosyltransférase qui se lie à l’ADN. En inhibant PARP14, nsp3 perturbe STAT1 (en) une protéine qui interagit avec l'ADN[55], ainsi que IRF3 une protéine régulatrice d’interférons[56] ;
- en agissant comme une déubiquitinase (en) (DUB), nsp3 annule les ordres des ubiquitines cellulaires, des petites protéines qui régulent le trafic et le recyclage des protéines dans la cellule en se fixant sur elles[57] ;
- plus encore, par deISGylation, nsp3 perturbe le cycle de ISG15 (en), une protéine qui se fixe également à d’autres protéines dans le but de les optimiser. En particulier, nsp3 clive les ISG15 des IRF3[57].
Par ailleurs des protéines non structurales sont en charge de prendre le contrôle de la machinerie traductionnelle de la cellule et d'éteindre la traduction « normale » qui jusque là fonctionne, empêchant ainsi la synthèse de protéines interférons par les ribosomes. Nsp1 se fixe au ribosome de la cellule hôte et bloque tous les ARN messagers de la cellule, sauf ceux liés au génome du virus[58]. Nsp2 se lie aux prohibitines, des protéines de la cellule hôte qui contribuent à la régulation environnementale de la cellule[59]. Nsp2 assure probablement d'autres fonctions importantes qui n'ont pas encore été découvertes.
Phase secondaire
D'autres protéines accessoires du SARS-CoV-2 verrouillent l'inhibition des interférons :
- La protéine ORF3b du SARS-CoV-2 est différente de celle du SARS-CoV-1. Toutes deux ont en commun qu'elles bloquent la translocation, c’est-à-dire l’entrée dans le noyau, de la protéine régulatrice d’interférons IRF3. ORF3b empêche ainsi IRF3 d’activer le gène censé transcrire des interférons, c'est-à-dire sonner l’alerte en réponse à l’infection. La protéine ORF3b du SARS-CoV-2 a une très forte activité anti-interférons alors même qu'elle est tronquée. L’ORF3b qui la code contient 4 codon-stops. Lorsque l'épidémie a éclaté à Wuhan, la protéine ORF3b n'était synthétisée que jusqu'au 1er codon stop. Au gré des mutations, les codons stop devraient progressivement être éliminés. Or plus la protéine ORF3b s'allonge, plus elle devrait gagner en efficacité. Et sans même avoir dépassé ses 4 paliers d’évolution, l’activité anti-interféron de la protéine ORF3b du SARS-CoV-2 est déjà plus importante que celle du SARS-CoV-1[60] - [61]. ORF3b est probablement la protéine du SARS-CoV-2 exerçant la plus forte activité anti-interférons ;
- la protéine ORF6 se lie au complexe des pores nucléaires de la cellule infectée. En présence d’ORF6, les ARN messagers cellulaires restent bloqués dans le noyau. Par un mécanisme assez analogue au virus de la stomatite vésiculaire, ORF6 se fixe via son C-terminal domain (CTD) au complexe protéique NUP98 (en)-RAE1 (en) impliqué dans le processus de transport de l’ARN. ORF6 bloque ainsi la translocation nucléaire induite par STAT1 (en) et STAT2. En perturbant le transport nucléo-cytoplasmique, ORF6 exerce une activité anti-interféron[62] - [63] - [64] ;
- la protéine ORF9b, également codée par le SARS-CoV-1, contribue à inhiber dans la cellule infectée la production d’interférons et bloque l’apoptose (autodestruction de la cellule) en interagissant avec la protéine membranaire externe mitochondriale TOMM70A (en). ORF9b se lie avec Tom70, par éviction de la protéine de choc thermique 90 (Hsp90), laquelle est essentielle au bon fonctionnement des interférons et de l’apoptose. Par ailleurs l’interaction entre Tom70 et ORF9b produit de l'acide lactique, peu propice à l’émission d’interférons[65] - [66] ;
- la protéine ORF9c du SARS-CoV-2 est analogue à la protéine ORF14 du SARS-CoV-1. Dans les cellules infectées, ORF9c interagit avec les protéines des organelles membranaires, c’est-à-dire les protéines du réticulum endoplasmique, des mitochondries ou encore celles de l'appareil de Golgi. ORF9c pourrait activement contribuer à l'évasion immunitaire du SARS-CoV-2. En présence de ORF9c, environ 150 protéines cellulaires antivirales censées produire des interférons ou aider à la présentation d’antigène viral, sont régulées à la baisse. A contrario, ORF9c régule à la hausse une quinzaine de protéines cellulaires favorables à sa réplication, lesquelles activent des cytokines pro-inflammatoires (IL-6 et IL-1) ou encore p38 MAPK[67] - [68] - [69].
Activité anti-antigène
Un antigène est un fragment d'un pathogène, permettant aux globules blancs de reconnaître l'agresseur et de lutter contre en mobilisant la mémoire immunitaire.
Dans les cellules infectées par le SARS-CoV-2, ORF8 pourrait bloquer le fonctionnement du CMH-I. Le CMH-I est censé en cas d’infection faciliter la présentation d’un antigène à des globules blancs : les lymphocytes T CD8. En bloquant le CMH-I, ORF8 permet au SARS-CoV-2 de retarder une réponse antivirale appropriée. D’autres virus ont cette faculté de perturber le fonctionnement du CMH-I des cellules infectées : le VIH avec sa protéine nef, ou encore l’adénovirus avec les protéines KSHV K3 et K5 et la protéine E3 / E19. La protéine ORF8 du SARS-CoV-2 a une faible homologie avec celle du SARS-CoV-1, ce qui laisse penser qu'elles accomplissent des fonctions différentes[70] - [71].
La protéine ORF3a perturbe le fonctionnement de l’autophagie, un mécanisme cellulaire qui contribue normalement à l’élimination d’agents pathogènes et à la synthèse d'un antigène. L’autophagie devient inefficace dès lors qu’ORF3a empêche la fusion des autophagosomes avec les lysosomes. Un autophagosome est une vésicule qui capture des débris ou des intrus. La dégradation des composants emprisonnés dans l’autophagosome dépend de la fusion avec un lysosome, une autre vésicule spécialisée dans la digestion des éléments intracellulaires. Cette fusion est médiée par un complexe protéinique associant SNARE STX17 (en)-SNAP29 (en)-AMP8 (en). Or le fonctionnement de la protéine SNARE STX17 dépend lui-même d’un autre complexe protéique appelé HOPS (homotypic protein sorting). Et ORF3a bloque HOPS en se liant à une de ses protéines, VPS39 (en), et en la séquestrant dans les endosomes tardives de la cellule infectée. Cette capacité d’ORF3a à perturber l’autophagie des cellules infectées est propre au SARS-CoV-2 et n’avait pas été observée chez le SARS-CoV-1[72].
Au sein de la cellule, la réplication de la protéine S contribue à activer la protéine GATA-3 au profit de l’évasion immunitaire du SARS-CoV-2. Sous l’influence de la protéine S, la protéine GATA-3 qui est un facteur de transcription de la cellule, stimule le gène codant HLA-E (en). Des molécules HLA-E sont ainsi produites et présentées au milieu extracellulaire par le CMH-I. En arborant des récepteurs HLA-E, les cellules infectées sont protégées contre une éventuelle destruction par les lymphocytes NK. En effet les récepteurs HLA-E des cellules infectées activent les récepteurs NKG2A des lymphocytes NK, ce qui a pour effet de les inhiber[73] - [74].
Dérèglement de l'homéostasie
La protéine ORF10 est propre au SARS-CoV-2 et n'est pas rencontrée chez les autres coronavirus humains. ORF10 interagit avec le complexe ubiquitine ligase Cullin 2 (en) via ZYG11B (en)[75]. La fonction de la protéine ORF10 est probablement de perturber la machinerie d'ubiquitination de la cellule infectée. Le système ubiquitine protéasome (SUP) a un rôle majeur dans l’homéostasie cellulaire, c'est-à-dire dans la régulation des protéines de la cellule et dans sa réponse innée face à une intrusion. De nombreux virus interagissent avec le complexe Cullin pour optimiser leur cycle réplicatif ou échapper à la réponse antivirale cellulaire : l'adénovirus, le virus d'Epstein-Barr, le papillomavirus humain (HPV) et le virus de l'immunodéficience bovine (en)[76]. D’après Élise Biquand et Caroline Demeret, la capacité d’un virus à détourner la machinerie d'ubiquitination de cellules favorise le franchissement de la barrière des espèces, et est un facteur crucial dans l’émergence d’une épidémie[77].
Réplication des virions
Dès qu’elles émergent des ribosomes, les protéines structurales s’assemblent entre elles dans le lumen (l’intérieur) d’un compartiment dérivé du réticulum endoplasmique, appelé « ERGIC (en) »[42]. Cette étape est appelée le bourgeonnement[42]. D’abord, une protéine N (nucléocapside) se fixe à une copie d'ARN et l’empaquète dans une protéine M (membrane) qui donne sa forme au virion[42]. Ensuite des protéines S sont incorporées[42]. La protéine M dirige la plupart des interactions protéine-protéine nécessaires à l'assemblage des virus après sa liaison à la nucléocapside[78]. La protéine E contribue à l’assemblage et à la libération du virion hors de la cellule infectée, en suivant la voie de sécrétion (appareil de Golgi, puis vésicules sécrétoires)[44]. Le virion quitte le milieu extracellulaire par exocytose, et est prêt à infecter une autre cellule.
Notes et références
Notes
- Mac-1 (en), appelé également CR3, est un récepteur exprimé principalement par un certain type de globules blancs : les monocytes, les macrophages ou les cellules dendritiques. Mac-1 fait partie du système du complément, c’est-à-dire d’une famille de protéines impliquées dans les mécanismes d'élimination des pathogènes. Mac-1 est le résultat de la liaison de CD18 avec CD11b. Mac-1 participe à la réponse immunitaire innée en reconnaissant les peptides antigènes étrangers et en les phagocytant. Mac-1 se lie facilement avec la protéine ORF7a du SARS-CoV-2 laquelle est présente sur les virions. Les globules blancs se liant à la protéine ORF7a sont potentiellement infectables par le SARS-CoV-2 ?
- LFA-1 fait également partie du système du complément. LFA-1 est le résultat de la liaison entre CD18 et CD11. LFA-1 est une protéine spécialisée dans la reconnaissance des antigènes. Le récepteur LFA-1 est exprimé par une grande variété de globules blancs : les lymphocytes B, les lymphocytes T, les lymphocytes NK, les macrophages et les neutrophiles. LFA-1 est reconnu par la protéine ORF7a du SARS-CoV-2
Références
- (en) Natalia Zamorano Cuervo, « ACE2: Evidence of role as entry receptor for SARS-CoV-2 and implications in comorbidities », Elife, (lire en ligne, consulté le ).
- (en) Bindu S. Mayi, « The role of Neuropilin-1 in COVID-19 », PLoS Pathog., (lire en ligne, consulté le )
- Léna Hespel, « La neuropiline, l’autre porte d’entrée pour le SARS-CoV-2 », sur Pourlascience.fr (consulté le )
- (en) James L. Daly, Boris Simonetti, Katja Klein et Kai-En Chen, « Neuropilin-1 is a host factor for SARS-CoV-2 infection », Science, vol. 370, no 6518, , p. 861–865 (ISSN 0036-8075 et 1095-9203, PMID 33082294, DOI 10.1126/science.abd3072, lire en ligne, consulté le )
- (en) Ludovico Cantuti-Castelvetri, Ravi Ojha, Liliana D. Pedro et Minou Djannatian, « Neuropilin-1 facilitates SARS-CoV-2 cell entry and infectivity », Science, vol. 370, no 6518, , p. 856–860 (ISSN 0036-8075 et 1095-9203, PMID 33082293, DOI 10.1126/science.abd2985, lire en ligne, consulté le )
- (en) Mariana Borsa, « Attacking the defence: SARS-CoV-2 can infect immune cells », Nature Reviews Immunology, (lire en ligne, consulté le ).
- (en) Marjorie C. Pontelli, « Infection of human lymphomononuclear cells by SARS-CoV-2 », University of Oxford - Immunology Network, (lire en ligne, consulté le ).
- (en) Michel Thépaut, Joanna Luczkowiak, Corinne Vivès et Nuria Labiod, « DC/L-SIGN recognition of spike glycoprotein promotes SARS-CoV-2 trans-infection and can be inhibited by a glycomimetic antagonist », PLOS Pathogens, vol. 17, no 5, , e1009576 (ISSN 1553-7374, PMID 34015061, PMCID PMC8136665, DOI 10.1371/journal.ppat.1009576, lire en ligne, consulté le )
- (en) Razie Amraei, Wenqing Yin, Marc A. Napoleon et Ellen L. Suder, « CD209L/L-SIGN and CD209/DC-SIGN Act as Receptors for SARS-CoV-2 », ACS Central Science, vol. 7, no 7, , p. 1156–1165 (ISSN 2374-7943 et 2374-7951, PMID 34341769, PMCID PMC8265543, DOI 10.1021/acscentsci.0c01537, lire en ligne, consulté le )
- (en) Nader Rahimi, « C-type Lectin CD209L/L-SIGN and CD209/DC-SIGN: Cell Adhesion Molecules Turned to Pathogen Recognition Receptors », Biology, (lire en ligne, consulté le ).
- Clara Delaunay, « Impact dans l'infection par le coronavirus de DC-SIGN, L-SECtin et des récepteurs apparentés : Caractérisation structurelle et fonctionnelle des récepteurs lectines de type C détournés par des virus pathogènes émergents. », École doctorale chimie et science du vivant (Grenoble), (lire en ligne, consulté le ).
- (en) Psylvia Léger, « Differential Use of the C-Type Lectins L-SIGN and DC-SIGN for Phlebovirus Endocytosis », Traffic, (lire en ligne, consulté le ).
- (en) Zhi-Yong Yang, « pH-Dependent Entry of Severe Acute Respiratory Syndrome Coronavirus Is Mediated by the Spike Glycoprotein and Enhanced by Dendritic Cell Transfer through DC-SIGN », American Society for Microbiology Journals, (lire en ligne, consulté le ).
- (en) Jiong Wang, « The potential for antibody-dependent enhancement of SARS-CoV-2 infection: Translational implications for vaccine development », J Clin Transl Sci., (lire en ligne, consulté le ).
- (en) Lynne Peeples, « News Feature: Avoiding pitfalls in the pursuit of a COVID-19 vaccine », Proceedings of the National Academy of Sciences, vol. 117, no 15, , p. 8218–8221 (ISSN 0027-8424 et 1091-6490, DOI 10.1073/pnas.2005456117, lire en ligne, consulté le ).
- (en) Paul A. Offit, « Antibody-dependent Enhancement (ADE) and Vaccines », Children's Hospital of Philadelphia, (lire en ligne, consulté le ).
- (en) Ke Wang, « CD147-spike protein is a novel route for SARS-CoV-2 infection to host cells », Nature - Signal Transduction and Targeted Therapy, (lire en ligne, consulté le ).
- (en) Ke Wang, Wei Chen, Yu-Sen Zhou et Jian-Qi Lian, « SARS-CoV-2 invades host cells via a novel route: CD147-spike protein », bioRxiv, Microbiology, (DOI 10.1101/2020.03.14.988345, lire en ligne, consulté le ).
- Cécile Crosnier, Leyla Y. Bustamante, S. Josefin Bartholdson et Amy K. Bei, « Basigin is a receptor essential for erythrocyte invasion by Plasmodium falciparum », Nature, vol. 480, no 7378, , p. 534–537 (ISSN 1476-4687, PMID 22080952, PMCID 3245779, DOI 10.1038/nature10606, lire en ligne, consulté le ).
- (en) Meng-Yao Zhang, Yang Zhang, Xiao-Dong Wu, Kun Zhang, Peng Lin, Hui-Jie Bian, Min-Min Qin, Wan Huang, Ding Wei, Zhao Zhang, Jiao Wu, Ruo Chen, Fei Feng, Bin Wang, Gang Nan, Ping Zhu et Zhi-Nan Chen, « Disrupting CD147-RAP2 interaction abrogates erythrocyte invasion by Plasmodium falciparum », Blood, vol. 131, no 10, , p. 1111–1121 (DOI 10.1182/blood-2017-08-802918, lire en ligne, consulté le ).
- (en) Keli Chen, « SARS-CoV-2 Nucleocapsid Protein Interacts with RIG-I and Represses RIG-Mediated IFN-β Production », Viruses, (lire en ligne, consulté le ).
- (en) Jarrod Shilts, « No evidence for basigin/CD147 as a direct SARS-CoV-2 spike binding receptor », Nature - Scientific Reports, (lire en ligne, consulté le ).
- (en) Zhinan Chen, « Function of HAb18G/CD147 in Invasion of Host Cells by Severe Acute Respiratory Syndrome Coronavirus », The Journal of Infectious Diseases, (lire en ligne, consulté le ).
- (en) Zubair Ahmed Nizamudeen, « Structural assessment of SARS-CoV2 accessory protein ORF7a predicts LFA-1 and Mac-1 binding potential », Bioscience Reports, (lire en ligne, consulté le ).
- (en) Ziliang Zhou, « Structural Insight Reveals SARS-CoV-2 Orf7a as an Immunomodulating Factor for Human CD14+ Monocytes », iScience, (lire en ligne [PDF], consulté le ).
- Imane Jamai Amir, « Covid-19 : virologie, épidémiologie et diagnostic biologique », Option/Bio., (lire en ligne, consulté le ).
- (en) Tomer Meirson, « Structural basis of SARS-CoV-2 spike protein induced by ACE2 », Bioinformatics., (lire en ligne, consulté le ).
- (en) Fatemeh Pourrajab, « Molecular Basis for Pathogenicity of Human Coronaviruses », Infect Drug Resist., (lire en ligne, consulté le ).
- (en) Wenfei Song, « Cryo-EM structure of the SARS coronavirus spike glycoprotein in complex with its host cell receptor ACE2 », PLoS Pathog., (lire en ligne, consulté le ).
- (en) Rory Henderson, « Controlling the SARS-CoV-2 spike glycoprotein conformation », Nature Structural & Molecular Biology, (lire en ligne, consulté le ).
- (en) Wai Tuck Soh, « The N-terminal domain of spike glycoprotein mediates SARS-CoV-2 infection by associating with L-SIGN and DC-SIGN », Biorxiv, (lire en ligne, consulté le ).
- (en) Umesh Kalathiya, « Highly Conserved Homotrimer Cavity Formed by the SARS-CoV-2 Spike Glycoprotein: A Novel Binding Site », J Clin Med., (lire en ligne, consulté le ).
- Etienne Decroly, « Rôle du site de clivage par la furine et du domaine de reconnaissance du récepteur ACE2 dans l'émergence du SARS-CoV-2 », IHU Méditerranée-Infection, (lire en ligne, consulté le ).
- Michihito Sasaki, « SARS-CoV-2 variants with mutations at the S1/S2 cleavage site are generated in vitro during propagation in TMPRSS2-deficient cells », PLoS Pathog., (lire en ligne, consulté le ).
- V. Bonny, « COVID-19 : physiopathologie d’une maladie à plusieurs visages », Rev Med Interne., (lire en ligne, consulté le ).
- Erwan Sallard, « Retrouver les origines du SARS-CoV-2 dans les phylogénies de coronavirus », Med Sci (Paris), (lire en ligne, consulté le ).
- (en) Julian Buchrieser, « Syncytia formation by SARS‐CoV‐2‐infected cells », EMBO J, (lire en ligne, consulté le ).
- (en) Samuel A. Theuerkauf, « Quantitative assays reveal cell fusion at minimal levels of SARS-CoV-2 spike protein and fusion from without », iScience, (lire en ligne, consulté le ).
- (en) Miyuki Kawase, « Protease-mediated entry via the endosome of human coronavirus 229E », J Virol., (lire en ligne, consulté le ).
- (en) Ehud Lavi, « Syncytia Formation Induced by Coronavirus Infection Is Associated with Fragmentation and Rearrangement of the Golgi Apparatus », Virology, (lire en ligne, consulté le ).
- (en) Jian Lei, Yuri Kusov et Rolf Hilgenfeld, « Nsp3 of coronaviruses: Structures and functions of a large multi-domain protein », Antiviral Research, vol. 149, , p. 58-74 (DOI 10.1016/j.antiviral.2017.11.001, lire en ligne, consulté le ).
- Matthew Borok, « Le cycle viral de SARS-CoV-2 », L'Arbre des connaissances, (lire en ligne, consulté le ).
- Dylan Juckel, « Les coronavirus, ennemis incertains », Med Sci (Paris), (lire en ligne, consulté le ).
- Jean-Claude Hervé et Naoum Salamé, « Réplication et expression de l’information génétique au cours du cycle infectieux du coronavirus », Institut français de l'éducation, (lire en ligne, consulté le ).
- « COVID-19 », Anticorps en ligne, (lire en ligne, consulté le ).
- (en) Everett Clinton Smith, « Coronaviruses as DNA Wannabes: A New Model for the Regulation of RNA Virus Replication Fidelity », PLoS Pathog, (lire en ligne, consulté le ).
- Guillaume Dubois, « Coronavirus humain OC43, neurovirulence et neuropropagation : Importance de sa protéine d’enveloppe », Institut National de la Recherche Scientifique du Québec, (lire en ligne, consulté le ).
- (en) Monica C. Pillon, « Cryo-EM structures of the SARS-CoV-2 endoribonuclease Nsp15 reveal insight into nuclease specificity and dynamics », Nature, (lire en ligne, consulté le ).
- « SARS-CoV-2 : comment se comporte-t-il une fois dans nos cellules ? », INSERM, (lire en ligne, consulté le ).
- (en) Philip V’kovski, « Coronavirus biology and replication: implications for SARS-CoV-2 », Nature, (lire en ligne, consulté le ).
- Luis Enjuanes, « Coronaviridae - Les Coronavirus », Departement of Molecular and Cell Biology, CNB, Campus Universidad Autonoma Cantoblanco, (lire en ligne, consulté le ).
- Ghizlane Maarifi, « La réponse interféron : Un grand pouvoir implique de grandes responsabilités », EDP Sciences, (lire en ligne, consulté le ).
- Erwan Sallard, « Les interférons de type I et III : des effecteurs de l’immunité innée antivirale », Planet Vie - ENS, (lire en ligne, consulté le ).
- (en) Yong Hu, « The Severe Acute Respiratory Syndrome Coronavirus Nucleocapsid Inhibits Type I Interferon Production by Interfering with TRIM25-Mediated RIG-I Ubiquitination », J Virol., (lire en ligne, consulté le ).
- (en) Jean-Michel Claverie, « A Putative Role of de-Mono-ADP-Ribosylation of STAT1 by the SARS-CoV-2 Nsp3 Protein in the Cytokine Storm Syndrome of COVID-19 », Viruses, (lire en ligne, consulté le ).
- (en) Amanda L Tauber, « The potential association between PARP14 and the SARS-CoV-2 infection (COVID-19) », Future Med Chem., (lire en ligne, consulté le ).
- (en) Donghyuk Shin, « Papain-like protease regulates SARS-CoV-2 viral spread and innate immunity », Nature, (lire en ligne, consulté le ).
- Franck Martin, « Une structure en "épingle à cheveux" contrôle la traduction du génome du virus SARS-CoV-2 », CNRS, (lire en ligne, consulté le ).
- (en) Francis K. Yoshimoto, « The Proteins of Severe Acute Respiratory Syndrome Coronavirus-2 (SARS CoV-2 or n-COV19), the Cause of COVID-19 », The Protein Journal, vol. 39, no 3, , p. 198–216 (ISSN 1875-8355, PMID 32447571, PMCID 7245191, DOI 10.1007/s10930-020-09901-4, lire en ligne, consulté le ).
- (en) Yoriyuki Konno, « SARS-CoV-2 ORF3b Is a Potent Interferon Antagonist Whose Activity Is Increased by a Naturally Occurring Elongation Variant », Cell Rep, (lire en ligne, consulté le ).
- (en) Yoriyuki Konno, « Review of SARS-CoV-2 ORF3b Is a Potent Interferon Antagonist Whose Activity Is Increased by a Naturally Occurring Elongation Variant », University of Oxford - Immunology Network, (lire en ligne, consulté le ).
- (en) Lisa Miorin, « SARS-CoV-2 Orf6 hijacks Nup98 to block STAT nuclear import and antagonize interferon signaling », PNAS, (lire en ligne, consulté le ).
- (en) Amin Addetia, « SARS-CoV-2 ORF6 disrupts nucleocytoplasmic transport through interactions with Rae1 and Nup98 », bioRxiv, (lire en ligne, consulté le ).
- (en) Yoichi Miyamoto, « SARS-CoV-2 ORF6 disturbs nucleocytoplasmic trafficking to advance the viral replication », bioRxiv, (lire en ligne, consulté le ).
- « Une équipe internationale de chercheurs a identifié des vulnérabilités communes aux coronavirus SARS-CoV-2, SARS-CoV-1 et MERS-CoV », CNRS, (lire en ligne, consulté le ).
- (en) He-wei Jiang, « SARS-CoV-2 Orf9b suppresses type I interferon responses by targeting TOM70 », Nature, (lire en ligne, consulté le ).
- (en) Fangfang Lu, « SARS-CoV-2 ORF9c: a mysterious membrane-anchored protein that regulates immune evasion? », Nature Reviews Immunology, (lire en ligne, consulté le ).
- (en) Ana Dominguez Andres, « SARS-CoV-2 ORF9c Is a Membrane-Associated Protein that Suppresses Antiviral Responses in Cells », University of Oxford - Immunology Network, (lire en ligne, consulté le ).
- (en) Nima Hemmat, « The roles of signaling pathways in SARS-CoV-2 infection; lessons learned from SARS-CoV and MERS-CoV », Archives of Virology, (lire en ligne, consulté le ).
- (en) Matthew D. Park, « Immune evasion via SARS-CoV-2 ORF8 protein? », Nature Reviews Immunology, (lire en ligne, consulté le ).
- (en) Y. Zhang, « The ORF8 Protein of SARS-CoV-2 Mediates Immune Evasion through Potently Downregulating MHC-I », University of Oxford - Immunology Network, (lire en ligne, consulté le ).
- (en) Guangyan Miao, « ORF3a of the COVID-19 virus SARS-CoV-2 blocks HOPS complex-mediated assembly of the SNARE complex required for autolysosome formation », Developmental Cell, (lire en ligne, consulté le ).
- (en) Daria Bortolotti, « SARS-CoV-2 Spike 1 Protein Controls Natural Killer Cell Activation via the HLA-E/NKG2A Pathway », Cells., (lire en ligne, consulté le ).
- Mathilde Allard, « Fonctions immunologiques de la molécule HLA-E :Implication en Cancérologie et en Transplantation », École Doctorale « Biologie Santé » Nantes – Angers, (lire en ligne, consulté le ).
- (en) David E. Gordon, « A SARS-CoV-2 protein interaction map reveals targets for drug repurposing », Nature, (lire en ligne, consulté le ).
- (en) Weijia Cai, « The structure and regulation of Cullin 2 based E3 ubiquitin ligases and their biological functions », Cell Div, (lire en ligne, consulté le ).
- Élise Biquand et Caroline Demeret, « Interactions avec le système ubiquitine : un prérequis de l’adaptation des virus influenza à l’homme ? », EDP Sciences, (lire en ligne, consulté le ).
- (en) Coronaviruses, vol. 1282, Springer, coll. « Methods in Molecular Biology », , 1–23 p. (ISBN 978-1-4939-2438-7, PMID 25720466, DOI 10.1007/978-1-4939-2438-7_1), « Coronaviruses: an overview of their replication and pathogenesis ».