Photosystème II
Le photosystème II (PSII) est une oxydoréductase qui catalyse la réaction :
- 2 H2O + 2 plastoquinone + 4 hν O2 + 2 plastoquinol.
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
Ce complexe enzymatique est le premier à intervenir dans les réactions de la photosynthèse dépendantes de la lumière. On le trouve dans la membrane des thylakoïdes chez les cyanobactéries et dans les chloroplastes des plantes et des algues. Il absorbe des photons dont l'énergie excite des électrons qui sont ensuite transférés à travers tout un ensemble de coenzymes et de cofacteurs jusqu'à une plastoquinone réduite en plastoquinol :
- Dans la photophosphorylation non cyclique, ces électrons sont remplacés par oxydation de molécules d'eau du lumen des thylakoïdes, ce qui a pour effet de libérer de l'oxygène O2 et des protons H+. Le photosystème II fournit ainsi les électrons pour toutes les réactions de photosynthèse et contribue à former un gradient de concentration de protons autour de la membrane des thylakoïdes en favorisant l'acidification du lumen de ces sous-organites. Les électrons à haut potentiel transférés à la plastoquinone sont utilisés en dernier ressort par le photosystème I pour réduire le NADP+ en NADPH.
- Dans la photophosphorylation cyclique, ces électrons sont excités dans le photosystème I puis circulent à travers la ferrédoxine, le complexe cytochrome b6f et la plastocyanine avant de retourner dans le photosystème I.
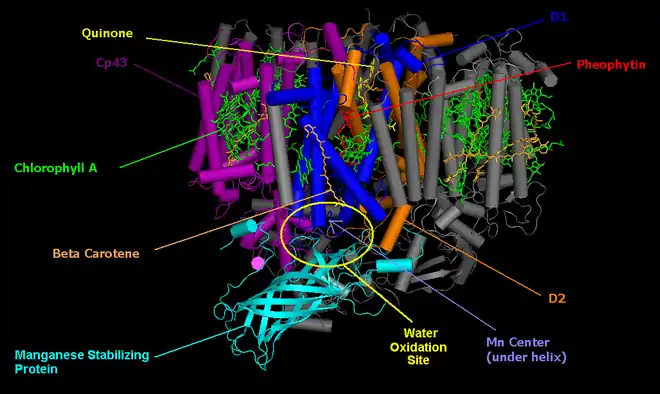
Structure
Le noyau du photosystème II consiste en un hétérodimère pseudo-symétrique de deux protéines homologues, D1 et D2[2]. Contrairement à tous les centres réactionnels de tous les autres photosystèmes qui ont une paire de molécules de chlorophylle étroitement associées, le pigment qui subit la séparation de charge photoinduite initiale dans le photosystème II est un monomère de chlorophylle. La charge positive n'étant pas partagée entre deux molécules, le pigment ionisé est très oxydant et intervient dans la dissociation de l'eau.
Le photosystème II des plantes vertes et des cyanobactéries est constitué d'une vingtaine de sous-unités, le nombre exact dépendant des espèces, ainsi que de diverses protéines de collecte lumineuse. Chaque photosystème II contient au moins 99 cofacteurs[3] :
- 35 chlorophylle a ;
- 12 β-carotène ;
- 2 phéophytine ;
- 2 plastoquinone ;
- 2 hème ;
- 1 bicarbonate HCO3− ;
- 20 lipides ;
- 1 cluster Mn4CaO5 (avec un ion chlorure Cl−) ;
- 1 cation ferreux Fe2+ non héminique ;
- peut-être également 2 cations de calcium Ca2+ par monomère.
| Sous-unités protéiques sont la fonction est connue | |
|---|---|
| Sous-unité | Fonction |
| D1 | Protéine contenant un centre réactionnel et se liant à la chlorophylle P680, à la phéophytine, au β-carotène, à la quinone et au complexe d'oxydation de l'eau |
| D2 | Protéine contenant un centre réactionnel |
| CP43 | Se lie au complexe d'oxydation de l'eau |
| PsbO | Protéine de stabilisation du manganèse, qui jouxte le complexe d'oxydation de l'eau |
| Coenzymes et cofacteurs | |
| Molécule | Fonction |
| Chlorophylle | Absorbe les photons lumineux et convertit leur énergie en énergie chimique |
| β-carotène | Absorbe les trop-pleins d'énergie de photoexcitation |
| Hème b559 | Également de la protoporphyrine IX contenant du fer |
| Phéophytine | Accepteur d'électrons initial |
| Plastoquinone | Transporteur d'électrons mobile interne à la membrane du thylakoïde |
| cluster Mn4CaO5 | Complexe d'oxydation de l'eau (Oxygen Evolving Complex) |
Plusieurs structures cristallines ont été déterminées pour le photosystème II. Les codes PDB d'accès aux structures de ces protéines sont par exemple 3ARC, 3BZ1, 3BZ2 (3BZ1 et 3BZ2 sont des structures monomériques du dimère de photosystème II), 2AXT (voir ci-dessous), 1S5L, 1W5C, 1ILX, 1FE1 et 1IZL.
Complexe d'oxydation de l'eau

Le complexe d'oxydation de l'eau, souvent appelé oxygen-evolving complex de son nom anglais, est un cluster oxométallique de quatre cations de manganèse dont le nombre d'oxydation va de +3 à +5 et un cation de calcium bivalent Ca2+. Au cours de l'oxydation de l'eau, les quatre électrons de l'eau circulent l'un après l'autre d'abord sur la chaîne latérale de la tyrosine D1-Y161 puis directement sur la chlorophylle P680.
La structure exacte de ce cluster demeure encore discutée, notamment les structures obtenues par cristallographie aux rayons X car il apparaît que les atomes de manganèse sont réduits par les rayons X intenses utilisés pour l'observation, ce qui altère la structure du complexe. Cependant, cette méthode, combinée avec d'autres techniques moins destructrices telles que la spectroscopie EXAFS et la résonance paramagnétique électronique, donne une idée assez claire de la structure de ce cluster. Une possibilité est une structure de type cubane[4].
Sa structure a été résolue en 2011 avec une résolution de 1,9 Å, ce qui a révélé la présence de cinq atomes d'oxygène servant de ponts oxo entre les cinq atomes métalliques et quatre molécules d'eau liées au cluster Mn4CaO5. Plus de 1 300 molécules d'eau ont été trouvées dans chaque photosystème II, certaines formant de grands réseaux par liaison hydrogène qui pourraient servir de canaux à protons H+, à molécules d'eau H2O ou à oxygène O2[5].

Notes et références
- (en) Bernhard Loll, Jan Kern, Wolfram Saenger, Athina Zouni et Jacek Biesiadka, « Towards complete cofactor arrangement in the 3.0 Å resolution structure of photosystem II », Nature, vol. 438, , p. 1040-1044 (lire en ligne) DOI 10.1038/nature04224
- (en) A. W. Rutherford et P. Faller, « Photosystem II: evolutionary perspectives », Philosophical Transactions of The Royal Society B Biological Sciences, vol. 358, no 1429, , p. 245-253 (PMCID 1693113, lire en ligne) DOI 10.1098/rstb.2002.1186
- (en) Albert Guskov, Jan Kern, Azat Gabdulkhakov, Matthias Broser, Athina Zouni et Wolfram Saenger, « Cyanobacterial photosystem II at 2.9-Å resolution and the role of quinones, lipids, channels and chloride », Nature Structural & Molecular Biology, vol. 16, , p. 334-342 (lire en ligne) DOI 10.1038/nsmb.1559
- (en) Kristina N. Ferreira, Tina M. Iverson, Karim Maghlaoui, James Barber et So Iwata, « Architecture of the Photosynthetic Oxygen-Evolving Center », Science, vol. 303, no 5665, , p. 1831-1838 (lire en ligne) DOI 10.1126/science.1093087
- (en) Yasufumi Umena, Keisuke Kawakami, Jian-Ren Shen et Nobuo Kamiya, « Crystal structure of oxygen-evolving photosystem II at a resolution of 1.9 Å », Nature, vol. 473, , p. 55-60 (lire en ligne) DOI 10.1038/nature09913