Ribonucléase pancréatique bovine
La ribonucléase pancréatique bovine, ou ribonucléase A, est la ribonucléase pancréatique extraite de l'espèce Bos taurus. Cette enzyme, une endonucléase qui clive l'ARN monocaténaire, est l'un des sujets d'études classiques en sciences des protéines[2]. Elle a été l'objet de deux prix Nobel de chimie : en 1972 à Christian Boehmer Anfinsen pour ses travaux sur le repliement des protéines et à Stanford Moore et à William Howard Stein pour leurs travaux sur la structure tridimensionnelle des protéines et leur mécanisme réactionnel[3] ; en 1984 à Robert Bruce Merrifield pour le développement de la synthèse chimique des protéines[4].
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
La ribonucléase pancréatique bovine clive l'ARN après les nucléotides à pyrimidine[5] selon un mécanisme en deux étables commun aux ribonucléases pancréatiques. Elle peut être inhibée par l'inhibiteur de ribonucléase, par les ions de métaux lourds, ou encore par les complexes uridine-vanadate.
Structure et propriétés
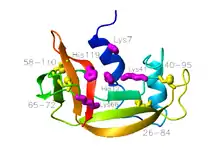
La ribonucléase A est une protéine d'assez petite taille, comprenant 124 résidus d'acides aminés pour une masse d'environ 13,7 kDa. On peut la décrire comme constituée de deux couches α et β avec une crevasse profonde se liant à l'ARN. La première couche est constituée de trois hélices α (résidus 3-14, 24-34 et 50-60) du côté N-terminal de la protéine. La deuxième couche est constituée de trois épingles à cheveux β (résidus 61-74, 79-104 et 105-124 du côté C-terminal) formant deux feuillets β. Les épingles à cheveux formées des résidus 61-74 et 105-124 forment un feuillet β à quatre brins antiparallèles qui repose sur l'hélice no 3 (formée des résidus 50-60). L'épingle à cheveux β la plus longue interagit avec un court brin β formé des résidus 42-45 pour donner un feuillet β à trois brins antiparallèles qui repose sur l'hélice no 2 (formée des résidus 24-34).
Son état natif contient quatre ponts disulfure situés entre les résidus de cystéine 26 et 84, 58 et 110, 40 et 95, et 65 et 72. Les deux premiers sont indispensables au repliement de la protéine en reliant une hélice α de la première couche à un feuillet β de la deuxième couche, formant un petit cœur hydrophobe dans leur voisine. Les deux derniers ne sont en revanche pas indispensables, et peuvent l'un ou l'autre être réduits (mais pas les deux) dans affecter l'état natif de la protéines dans les conditions physiologiques. Ces ponts disulfure relient des segments formant des boucles et sont relativement exposés au solvant. Le dernier des quatre ponts disulfure a une tendance naturelle très forte à se former, bien supérieurs à ce que laisserait supposer son entropie de boucle (en), aussi bien comme peptide que comme protéine entière. Ceci tend à suggérer que l'épingle à cheveux β constituée des résidus 61-74 a une forte tendance naturelle à se former lors du repliement général de la protéine.
C'est une protéine basique, dont le point isoélectrique vaut pI = 9,63. Ses nombreuses charges électriques positives sont cohérentes avec le fait qu'elle se lie à l'ARN, qui est un polyanion. De manière plus générale, elle est inhabituellement pauvre en résidus apolaires, particulièrement en chaînes latérales aliphatiques. Ceci pourrait expliquer qu'il lui faille pas moins de quatre ponts disulfure pour stabiliser sa structure tridimensionnelle. La pauvreté en résidus hydrophobes pourrait également être utile en réduisant la répulsion électrostatique en ses groupes fortement chargés électriquement et les régions à faible permittivité que sont les résidus apolaires.
L'hélice α N-terminale, formée des résidus 3-13, est connectée au reste de la protéine par un expaceur flexible formé des résidus 16-23. Cet espaceur peut être clivé par la subtilisine entre les résidus no 20 et no 21 sans que cette hélice ne se dissocie du reste de l'enzyme. Le complexe protéique qui en résulte est appelé ribonucléase S ; le peptide formé par les résidus 1-20 est le peptide S tandis que le reste de la protéine, formé des résidus 21-124, est la protéine S. La constante de dissociation du peptide S de la protéine S est voisine de 30 × 10−12 mol L−1 : cette forte affinité peut être mise à profit par les techniques de purification des protéines en liant le peptide S à une protéine étudiée et en faisant circuler l'ensemble à travers un dispositif de chromatographie d'affinité.
Histoire
La ribonucléase pancréatique bovine est devenue un objet d'études courant en biologie moléculaire essentiellement parce qu'elle est extrêmement stable et a pu être purifiée en grandes quantités même avec les techniques les plus anciennes de purification des protéines. Armour and Company a ainsi pu purifier un kilogramme de ribonucléase A en 1940[6], ce qui représente une grande quantité d'enzyme, surtout pour cette époque, et permettait de disposer d'échantillons de ribonucléase à bas prix. Son nom de ribonucléase A vient précisément de sa grande abondance sur le marché et de son rôle de premier plan dans l'étude des premières ribonucléases.
Les études de Christian Boehmer Anfinsen sur le repliement oxydatif de la ribonucléase pancréatique bovine ont jeté les bases de la compréhension des relations entre la séquence en acides aminés et le repliement des protéines en une structure tridimensionnelle donnée, et ont consolidé l'hypothèse thermodynamique (en) selon laquelle une protéine adopte une conformation tridimensionnelle qui minimise son énergie libre[6] - [7].
La ribonucléase A est également la première enzyme pour laquelle un mécanisme réactionnel correct a été proposé, et ce avant même que sa structure ait été élucidée[8]. C'est également la première protéine mettant en évidence l'effet de l'isomérisation non native de liaisons peptidiques précédant des résidus de proline lors du repliement[9].
La ribonucléase pancréatique bovine a par ailleurs été le premier modèle protéique étudié à l'aide de techniques telles que l'absorbance, le dichroïsme circulaire, la spectroscopie Raman, la résonance paramagnétique électronique et la résonance magnétique nucléaire, ou à faire l'objet de méthodes d'étude chimique telles que la modification chimique des chaînes latérales exposées, la reconnaissance antigénique et la protéolyse limitée de segments désordonnés. La ribonucléase S, résultant du traitement de la ribonucléase A par la subtilisine, fut la troisième protéine dont la structure tridimensionnelle a été résolue par cristallographie, en 1967[10].
Notes et références
- (en) Alexander Wlodawer, L. Anders Svensson, Lennart Sjoelin et Gary L. Gilliland, « Structure of phosphate-free ribonuclease A refined at 1.26 Å », Biochemistry, vol. 27, no 8, , p. 2705-2717 (PMID 3401445, DOI 10.1021/bi00408a010, lire en ligne)
- (en) Ronald T. Raines, « Ribonuclease A », Chemical Reviews, vol. 98, no 3, , p. 1045-1066 (PMID 11848924, DOI 10.1021/cr960427h, lire en ligne)
-
(en) « The Nobel Prize in Chemistry 1972 » (consulté le ) :
« The Nobel Prize in Chemistry 1972 was divided, one half awarded to Christian B. Anfinsen "for his work on ribonuclease, especially concerning the connection between the amino acid sequence and the biologically active conformation", the other half jointly to Stanford Moore and William H. Stein "for their contribution to the understanding of the connection between chemical structure and catalytic activity of the active centre of the ribonuclease molecule". »
-
(en) « The Nobel Prize in Chemistry 1984 » (consulté le ) :
« The Nobel Prize in Chemistry 1984 was awarded to Bruce Merrifield "for his development of methodology for chemical synthesis on a solid matrix". »
- (en) « On the structure of ribonucleic acids. II. The products of ribonuclease action », Journal of Biological Chemistry, vol. 205, no 2, , p. 767-782 (PMID 13129256, lire en ligne)
- (en) F. M. Richards, « The 1972 nobel prize for chemistry », Science, vol. 178, no 4060, , p. 492-493 (DOI 10.1126/science.178.4060.492, lire en ligne)
- (en) Garland R. Marshall, Jiawen A. Feng et Daniel J. Kuster, « Back to the future: Ribonuclease A », Peptide Science, vol. 90, no 3, , p. 259-277 (PMID 17868092, DOI 10.1002/bip.20845, lire en ligne)
- (en) Claudi M. Cuchillo, M. Victòria Nogués et Ronald T. Raines, « Bovine Pancreatic Ribonuclease: Fifty Years of the First Enzymatic Reaction Mechanism », Biochemistry, vol. 50, no 37, , p. 7835-7841 (PMID 21838247, PMCID 3172371, DOI 10.1021/bi201075b, lire en ligne)
- (en) Franz X. Schmid et Robert L. Baldwin, « Acid catalysis of the formation of the slow-folding species of RNase A: Evidence that the reaction is proline isomerization », Proceedings of the National Academy of Sciences of the United States of America, vol. 75, no 10, , p. 4764-4768 (PMID 283390, PMCID 336200, DOI 10.1073/pnas.75.10.4764, lire en ligne)
- (en) H. W. Wyckoff, Karl D. Hardman, N. M. Allewell, Tadashi Inagami, L. N. Johnson et Frederic M. Richards, « The Structure of Ribonuclease-S at 3.5 A Resolution », Journal of Biological Chemistry, vol. 242, no 17, , p. 3984-3988 (PMID 6037556, lire en ligne)