Phylogéographie
La phylogéographie est l'étude des principes et processus qui gouvernent la distribution des lignées généalogiques, spécialement celle de niveau intraspécifique[1] - [2].
Elle étudie les phénomènes génétiques et démographiques, en particulier les phénomènes de spéciation[2], ayant conduit à la distribution et à la structuration actuelle des populations, par exemple sous l'effet de la dérive des continents, des modifications de courants océaniques, des oscillations climatiques (qui induisent des glaciations et une baisse importante du niveau marin notamment), ou de catastrophes géoclimatiques (résilience post-crises d'extinctions majeures).
Depuis peu et prospectivement, elle doit prendre en compte le fait que l'Homme est à l'origine de grandes perturbations phylogéographiques, à échelle planétaire, à la suite de l'invention de l'usage du feu, de l'agriculture, et des transports planétaires (espèces introduites, espèces invasives, maladies émergentes, etc.), mais aussi à la suite de la fragmentation écopaysagère des continents, à la surpêche, ou à la suite de la destruction et artificialisation des milieux à grande échelle, ou encore à la suite du dérèglement climatique induit par des émissions massives de gaz à effet de serre, parallèlement à la destruction ou saturation de nombreux puits de carbone. La diffusion dans le milieu naturel de nombreuses populations clonées, et d'OGM ou d'espèces devenues antibiorésistantes, résistantes à certains biocides (dont pesticides agricoles ou domestiques ou vétérinaires) posent également de nouveaux problèmes.
Développement
Le terme phylogéographie a été utilisé pour la première fois par John Avise dans son travail Intraspecific Phylogeography: The Mitochondrial DNA Bridge Between Population Genetics and Systematics (Phylogéographie intraspécifique : le pont d'ADN mitochondiral entre la génétique des populations et la systématique), publié en 1987[3]. La biogéographie historique examine comment les conditions historiques, géologiques, climatiques et écologiques ont influencé la répartition actuelle des espèces. Dans le cadre de la biogéographie historique, les chercheurs évaluaient le passé des relations géographiques et évolutives des organismes. Deux développements au cours des années 60 et 70 ont été particulièrement importants pour former les bases de la phylogéographie moderne : le premier était la diffusion de la pensée cladistique, et le second était le développement de la théorie de la tectonique des plaques[4].
L'école de pensée qui en résulte est la biogéographie par vicariance, qui explique l'origine de nouvelles lignées à travers des événements géologiques comme la dérive des continents ou la formation de rivières. Lorsqu'une population est divisée par une nouvelle rivière ou une nouvelle chaîne de montagnes (événement de vicariance), deux populations sont créées. La paléogéographie, la géologie et la paléoécologie sont des domaines importants qui fournissent des informations intégrées dans les analyses phylogéographiques.
Au milieu des années 1970, les analyses génétiques des populations se tournent vers les marqueurs mitochondriaux[5]. Les progrès de la réaction en chaîne par polymérase (PCR), le processus par lequel des millions de copies d'un segment d'ADN peuvent être répliqués et séquencés, est crucial dans le développement de la phylogéographie : les informations contenues dans les séquences d'ADN mitochondrial deviennent beaucoup plus accessibles. Les méthodes de calcul progressent aussi et utilisent mieux les données (par exemple, en utilisant la théorie de la coalescence) améliorent l'inférence phylogéographique[5].
Les premiers travaux phylogéographiques ont été récemment critiqués pour leur nature narrative et leur manque de rigueur statistique. La seule méthode recevable était l'analyse des clades imbriqués d'Alan Templeton, qui utilise une clé d'inférence pour déterminer la validité d'un processus donné pour expliquer la concordance entre la distance géographique et la parenté génétique. Les travaux plus récents utilisent des approches statistiques plus forte qu'initialement[6] - [7] - [8].
Exemple
Les cycles de glaciation des derniers 2,4 millions d'années ont périodiquement restreint certaines espèces à des refuges isolés. Cette distribution restreinte peut entraîner un goulet d'étranglement dans une population, ce qui réduit la variabilité génétique. Une fois que l'inversion du changement climatique permet une migration rapide hors des zones refuge, ces espèces se propagent rapidement dans l'habitat nouvellement disponible. Un certain nombre d'études empiriques trouvent des signatures génétiques d'espèces animales et végétales qui soutiennent ce scénario de refuges et d'expansion postglaciaire[9]. Cela s'est produit sous les tropiques (où le principal effet de la glaciation est l'augmentation de l'aridité, c'est-à-dire l'expansion de la savane et la rétraction de la forêt tropicale humide) [10] - [11] mais aussi dans les régions tempérées directement influencées par les glaciers[12].
Phylogéographie et conservation
La phylogéographie peut aider à hiérarchiser les zones de haute valeur pour la conservation.
Les analyses phylogéographiques jouent un rôle important dans la définition des unités évolutives significatives (UES), une unité de conservation en dessous du niveau de l'espèce qui est souvent définie sur la distribution géographique unique et les modèles génétiques mitochondriaux[13].
Une étude récente sur les écrevisses des cavernes en péril dans les Appalaches de l'est de l'Amérique du Nord [14] démontre comment les analyses phylogénétiques ainsi que la répartition géographique peuvent aider à reconnaître les priorités de conservation. En utilisant des approches phylogéographiques, les auteurs ont découvert que dans ce que l'on pensait être une seule espèce largement distribuée, deux espèces étaient présentes, dont une espèce ancienne. Les autorités ont pu prendre des décisions pour garantir que les deux lignées soient protégés.
Une analyse des salamandres du genre Eurycea, également dans les Appalaches, révèle que la taxonomie du groupe sous-estimait la diversité des espèces[15]. Les auteurs de cette étude ont aussi constaté que les modèles de diversité phylogéographique étaient davantage associés aux profils passés d'écoulement des eaux qu'aux profils actuels, ce qui indique que les changements passés ont joué un rôle important dans la diversité des populations des salamandres.
Une compréhension approfondie de la structure phylogéographique permet ainsi de mieux prioriser les zones de conservation.
Phylogéographie comparative
Le domaine de la phylogéographie comparative cherche à expliquer les mécanismes responsables des relations phylogéniques et de distribution entre différentes espèces. Par exemple, des comparaisons entre plusieurs taxons peuvent clarifier l'histoire des régions biogéographiques[16]. Par exemple, les analyses phylogéographiques des vertébrés terrestres de la péninsule de Basse-Californie[17] et des poissons marins des deux côtés du Pacifique et du golfe de la péninsule[16] présentent des signatures génétiques qui suggèrent qu'un événement de vicariance a affecté plusieurs taxons pendant le Pléistocène ou le Pliocène.
La phylogéographie donne également une perspective historique importante sur la composition de la communauté. L'histoire explique la diversité régionale et locale de deux manières[10]. Premièrement, au niveau régional, la composition de la réserve d'espèces résulte de l'équilibre entre la spéciation et l'extinction. Deuxièmement, au niveau local, la composition des espèces est influencée par l'interaction entre l'extinction locale des populations d'espèces et la recolonisation[10]. Une approche phylogénétique comparative dans les zones tropicales humides d'Australie indique que les modèles régionaux de distribution et de diversité des espèces sont largement déterminés par les extinctions locales et les recolonisations ultérieures correspondant aux cycles climatiques.
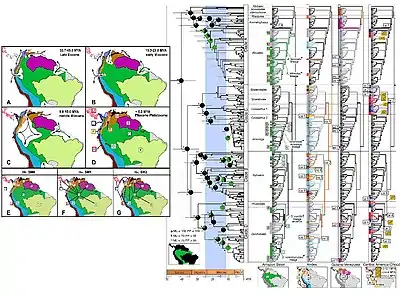
La phylogéographie intègre la biogéographie et la génétique pour étudier plus en détail l'histoire linéaire d'une espèce dans le contexte de l'histoire géoclimatique de la planète. Un exemple d'étude de grenouilles venimeuses d'Amérique du Sud (illustré en introduction) est utilisé pour démontrer comment les phylogéographes combinent la génétique et la paléogéographie pour reconstituer l'histoire écologique des organismes dans leur environnement. Plusieurs événements géoclimatiques majeurs ont influencé la distribution biogéographique des organismes dans cette zone, y compris l'isolement et la reconnexion de l'Amérique du Sud, le soulèvement des Andes, un vaste système de bassin d'inondation en Amazonie au cours du Miocène, la formation de l'Orénoque et de l'Amazone, et les cycles entre différents climats humides et secs du Pliocène au Pléistocène[18].
En utilisant ces informations paléogéographiques contextuelles (chronologie dans les cases A à D), les auteurs de cette étude[18] proposent une hypothèse nulle qui ne suppose aucune structure spatiale et deux hypothèses alternatives impliquant la dispersion et d'autres contraintes biogéographiques (les hypothèses sont présentées dans les cases E à G, nommées SMO, SM1 et SM2).
L'histoire évolutive et les relations entre les différentes espèces de grenouilles venimeuses sont reconstruites à l'aide d'arbres phylogénétiques dérivés de données moléculaires. Les arbres moléculaires sont cartographiés par rapport à l'histoire paléogéographique de la région pour une étude phylogéographique complète. L'arbre au centre de la figure a des longueurs de branches calibrées sur une horloge moléculaire : la barre de temps géologique est affichée en bas. Le même arbre phylogénétique est dupliqué quatre fois de plus pour montrer où chaque lignage est distribué et se trouve (illustré dans les cartes en-dessous)[18].
La combinaison de techniques utilisées dans cette étude illustre plus généralement comment les études phylogéographiques se déroulent et testent des modèles d'influence commune. Les données paléogéographiques enregistrent les événements historiques qui expliquent les ramifications dans les arbres moléculaires.
Cette étude constate que toutes les espèces de grenouilles toxiques d'Amazonie existantes proviennent de quatorze lignées qui se sont dispersées dans leurs zones respectives après le retrait du bassin d'inondation du Miocène[18]. Des études phylogéographiques régionales de ce type sont répétées pour différentes espèces comme moyen d'essai indépendant. Les phylogéographes trouvent des schémas largement concordants et répétés parmi les espèces de la plupart des régions de la planète, ce qui est dû à une influence commune de l'histoire paléoclimatique[19].
Phylogéographie humaine
La phylogéographie permet de comprendre l'origine et les schémas de dispersion de l'espèce Homo sapiens. Sur la base d'observations de restes squelettiques d'anciens restes humains et d'estimations de leur âge, les anthropologues proposent deux hypothèses concurrentes sur les origines humaines.
La première hypothèse est appelée le modèle Out of Africa, qui soutient que la dernière expansion hors de l'Afrique il y a environ 100 000 ans a conduit les humains modernes à remplacer tous les autres populations du genre Homo d'Eurasie résultant d'une vague précédente d'émigration hors d'Afrique. La seconde hypothèse, à origine multirégionale (ou Out of nowhere) propose que les individus de cette même expansion d'il y a 100 000 ans se sont mélangés génétiquement avec des populations humaines d'émigrations africaines plus anciennes. Une étude phylogéographique a découvert une Ève mitochondriale qui vivait en Afrique il y a 150 000. Cette étude fournit un soutien précoce au modèle Out-of-Africa[20].
Bien que cette étude ait ses lacunes, elle a reçu une attention considérable des scientifiques et du grand public. Une analyse phylogéographique plus approfondie qui utilise dix gènes différents au lieu d'un seul marqueur mitochondrial indique qu'au moins deux expansions majeures hors d'Afrique après l'extension initiale de l'aire Homo erectus ont eu lieu, et ont joué un rôle important dans la formation du pool génétique humain moderne : de nombreux échanges génétiques ont eu lieu[21]. Ces résultats ont fortement démontré le rôle central de l'Afrique dans l'évolution des humains modernes, mais ont également indiqué que le modèle multirégional avait une certaine validité. Ces études ont été largement supplantées par des études génomiques de population qui utilisent beaucoup plus de données.
L'histoire de l'évolution humaine est devenue plus claire avec le projet 1000 génomes. L'ADN de milliers d'individus dans le monde, mais aussi d'échantillons prélevés sur deux homininis non-Homo sapiens (Néandertaliens et Denisoviens) a été séquencé et analysé. Il y aurait eu plusieurs mélanges entre Néandertaliens, Denisoviens, et homininis africains archaïques. Une expansion eurasienne aurait eu lieu dans la région australasienne, précédant l'expansion hors de l'Afrique.
Phylogéographie des virus
Les virus aident à comprendre la dynamique du changement évolutif grâce à leur taux de mutation rapide et leur temps de génération rapide[22]. Une approche phylogéographique a été adoptée pour de nombreuses maladies qui menacent la santé humaine, notamment la dengue, la rage, la grippe et le VIH[22]. De même, une approche phylogéographique jouera probablement un rôle clé dans la compréhension des vecteurs et de la propagation de la grippe aviaire, démontrant la pertinence de la phylogéographie pour le grand public.
Phylogéographie des langues
Une étude phylogéographique des langues anciennes et modernes a tenter de vérifier si les langues indo-européennes provenaient d' Anatolie ou des steppes d'Asie centrale[23]. L'évolution de la langue a été modélisée en termes de gain et de perte de mots apparentés dans chaque langue au fil du temps, pour produire un cladogramme des langues apparentées. La combinaison de ces données avec les plages géographiques connues de chaque langue permet d'affirmer que les langues ont une origine anatolienne datant de 8 000 à 9 500 ans[23].
L’apport des SIG en phylogéographie
Un biais fréquent dans la littérature phylogéographique provient du fait que les aspects phylogéniques et purement géographiques qui constituent cette discipline ont longtemps été isolés les uns des autres. Ainsi, la plupart du temps, les données qui constituent la phylogénie (les données moléculaires, les traits morphologiques observés, etc.) doivent être quantitatifs et statistiquement testables, alors que la composante géographique de la géophylogénie n’est souvent que qualitative. Pour cette raison, la phylogéographie a souvent été une approche narrative et spéculative décrivant et analysant la microévolution et les principes de spéciation[2].
Un système d’information géographique (SIG) est un ensemble informatique d’outils qui collecte, stocke, transforme, analyse et qui affiche des données variées géoréférencées pour résoudre des problèmes divers[24]. La phylogéographie utilise un ensemble hétérogène de données quantitatives et qualitatives, obtenues à partir d’une grande variété de sources, et qui sont, ou peuvent être facilement géoréférencées. Les SIG facilitent l’intégration et l’interrelation des toutes ces données[2].
Un défi est donc d'appliquer le potentiel offert par les outils de l'information géographique aux principes de la phylogéographie pour créer un système d’information phylogéographique dans le but d’unifier plus efficacement phylogénie et géographie[2]. Cela est maintenant possible avec des logiciels[25]. Exemple : Geophylobuilder 1.0, qui crée des géophylogénies à partir d’une phylogénie et de données géographiques qui lui sont associées.
Le SIG apporte un environnement informatique facile d'approche permettant la récolte, l'analyse et la visualisation de données géographiques, et donc de former des systèmes intégrés d’information phylogéographiques.
Il y a actuellement un intérêt grandissant des phylogéographes pour l’analyse spatiale et géographique, et les SIG permettent de lier entre elles les données génétiques et environnementales provenant d’études diverses via un langage commun géoréférencé[2].
Geophylobuilder 1.0 pour ArcGIS
Geophylobuilder 1.0 pour ArcGIS est une extension développée par Kidd et Liu[25]. Cette extension utilise les phylogénies, dont les fichiers sont de type Newick (.nwk), et des données qui définissent l’emplacement spatial des entités échantillonnées, comme un échantillon d’ADN, par exemple. La visualisation des géophylogénies ainsi créées est possible en deux dimensions dans ArcMap ou en trois dimensions dans ArcScene.
Exemple d’application de Geophylobuilder 1.0
L’article de Webb et al.[26] décrit la phylogénie de la famille des Goodeidae sur la base de gènes mitochondriaux. Les auteurs ont tenté de décrire cette phylogénie dans un contexte géographique.
Notes et références
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Phylogeography » (voir la liste des auteurs).
- (en) John C. Avise, « The history and purview of phylogeography : a personal reflexion », Molecular Ecology, vol. 7, no 4, , p. 371-379 (DOI 10.1046/j.1365-294x.1998.00391.x)
- (en) David M. Kidd et Michael G. Ritchie, « Phylogeographic information systems : putting the geography into phylogeography », Journal of biogeography, vol. 33, no 11, , p. 1851-1865 (ISSN 0305-0270)
- Avise, J Arnold, R M Ball et E Bermingham, « Intraspecific Phylogeography: The Mitochondrial DNA Bridge Between Population Genetics and Systematics », Annual Review of Ecology and Systematics, vol. 18, , p. 489–522 (DOI 10.1146/annurev.es.18.110187.002421)
- De Queiroz, A., « The resurrection of oceanic dispersal in historical biogeography », Trends in Ecology and Evolution, vol. 20, no 2, , p. 68–73 (PMID 16701345, DOI 10.1016/j.tree.2004.11.006)
- Avise, J. C., « The history and purview of phylogeography: a personal reflection », Molecular Ecology, vol. 7, no 4, , p. 371–379 (DOI 10.1046/j.1365-294x.1998.00391.x, lire en ligne)
- Knowles, L. L. and W. P. Maddison, « Statistical phylogeography », Molecular Ecology, vol. 11, no 12, , p. 2623–2635 (PMID 12453245, DOI 10.1046/j.1365-294X.2002.01637.x)
- Templeton, A. R., E. Routman et C. A. Phillips, « Separating Population Structure from Population History: A Cladistic Analysis of the Geographical Distribution of Mitochondrial DNA Haplotypes in the Tiger Salamander, Ambystoma Tigrinum », Genetics, vol. 140, no 2, , p. 767–782 (PMID 7498753, PMCID 1206651)
- Templeton, A. R., « Nested clade analyses of phylogeographic data: testing hypotheses about gene flow and population history », Molecular Ecology, vol. 7, no 4, , p. 381–397 (PMID 9627999, DOI 10.1046/j.1365-294x.1998.00308.x)
- Cruzan, M. B et A. R. Templeton, « Paleoecology and coalescence: phylogeographic analysis of hypotheses from the fossil record », Trends in Ecology and Evolution, vol. 15, no 12, , p. 491–496 (PMID 11114435, DOI 10.1016/S0169-5347(00)01998-4)
- Schneider, C. J., M. Cunningham et C. Moritz, « Comparative phylogeography and the history of endemic vertebrates in the Wet Tropics rainforests of Australia », Molecular Ecology, vol. 7, no 4, , p. 487–498 (DOI 10.1046/j.1365-294x.1998.00334.x)
- Da Silva, M. N. F. and J. L. Patton, « Molecular phylogeography and the evolution and conservation of Amazonian mammals », Molecular Ecology, vol. 7, no 4, , p. 475–486 (PMID 9628001, DOI 10.1046/j.1365-294x.1998.00276.x)
- Taberlet, P., L. Fumagalli, A.G. Wust-Saucy et J.F. Cossons, « Comparative phylogeography and postglacial colonization routes in Europe », Molecular Ecology, vol. 7, no 4, , p. 453–464 (PMID 9628000, DOI 10.1046/j.1365-294x.1998.00289.x)
- Moritz, C., « Defining "evolutionary significant units" for conservation », Trends in Ecology and Evolution, vol. 9, no 10, , p. 373–375 (PMID 21236896, DOI 10.1016/0169-5347(94)90057-4)
- Buhay, J. E. et K. A. Crandall, « Subterranean phylogeography of freshwater crayfishes shows extensive gene flow and surprisingly large population sizes », Molecular Ecology, vol. 14, no 14, , p. 4259–4273 (PMID 16313591, DOI 10.1111/j.1365-294X.2005.02755.x)
- Kozak, K. H., A. B. Russell et A. Larson, « Gene lineages and eastern North American paleodrainage basins: phylogeography and speciation in salamanders of the Eurycea bislineata species complex », Molecular Ecology, vol. 15, no 1, , p. 191–207 (PMID 16367840, DOI 10.1111/j.1365-294X.2005.02757.x)
- Riginos, C., « Cryptic vicariance in Gulf of California fishes parallels vicariant patterns found in Baja California mammals and reptiles », Evolution, vol. 59, no 12, , p. 2678–2690 (PMID 16526514, DOI 10.1554/05-257.1)
- Riddle, B. R., D. J. Hafner, L. F. Alexander et J. R. Jaeger, « Cryptic vicariance in the historical assembly of a Baja California Peninsular Desert biota », Proceedings of the National Academy of Sciences, vol. 97, no 26, , p. 14438–14443 (PMID 11095731, PMCID 18937, DOI 10.1073/pnas.250413397, Bibcode 2000PNAS...9714438R)
- Santos, Coloma, Summers, Caldwell et al., « Amazonian Amphibian Diversity Is Primarily Derived from Late Miocene Andean Lineages », PLOS Biol, vol. 7, no 3, , e1000056 (PMID 19278298, PMCID 2653552, DOI 10.1371/journal.pbio.1000056)
- J. Avise, Phylogeography: The History and Formation of Species, President and Fellows of Harvard College, (ISBN 978-0-674-66638-2, lire en ligne)
- Cann, R.L., Stoneking, M. et A. C. Wilson, « Mitochondrial DNA and human evolution », Nature, vol. 325, no 6099, , p. 31–36 (PMID 3025745, DOI 10.1038/325031a0, Bibcode 1987Natur.325...31C)
- Templeton, A. R., « Out of Africa again and again », Nature, vol. 416, no 6876, , p. 45–51 (PMID 11882887, DOI 10.1038/416045a, Bibcode 2002Natur.416...45T, lire en ligne)
- Holmes, E. C., « The phylogeography of human viruses », Molecular Ecology, vol. 13, no 4, , p. 745–756 (PMID 15012753, DOI 10.1046/j.1365-294X.2003.02051.x)
- Bouckaert, Philippe Lemey, Michael Dunn et Simon J. Greenhill, « Mapping the Origins and Expansion of the Indo-European Language Family », Science, vol. 337, no 6097, , p. 957–960 (PMID 22923579, PMCID 4112997, DOI 10.1126/science.1219669, Bibcode 2012Sci...337..957B)
- (en) Ian Heywood, Sarah Cornelius et Steve Carver, An introduction to geographical information systems, Pearson Education Limited, , 464 p. (ISBN 978-0-13-129317-5)
- (en) David M. Kidd et Xianhua Liu, « Geophylobuilder 1.0 : an ArcGIS extension for creating geophylogenies », Molecular Ecology Ressources, vol. 8, no 1, , p. 88-91 (DOI 10.1111/j.1471-8286.2007.01925.x)
- Shane A. Webb, Jefferson A. Graves, Constantino Macias-Garcia, Anne E. Magurran, Diarmid Ó Foighil et Michael G. ritchie, « Molecular phylogeny of the livebearing Goodeidae (Cyprinodontiformes) », Molecular Phylogenetics and Evolution, vol. 30, no 3, , p. 527-544 (DOI 10.1016/S1055-7903(03)00257-4)
Voir aussi
Articles connexes
Bibliographie
- Avise, J.C. (1992) Molecular population structure and the biogeographic history of a regional fauna: a case history with lessons for conservation biology. Oikos 63: 62-76.
- Avise, J.C. (2000) Phylogeography: The history and formation of species Cambridge, Massachusetts: Harvard University Press.