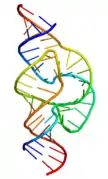
Ribozyme en tête de marteau
Un ribozyme en tête de marteau (hammerhead ribozyme en anglais) est un segment d'ARN qui catalyse le clivage et la jonction réversibles d'un site spécifique sur une molécule d'ARN. Ces ribozymes sont utilisés comme modèles pour la recherche sur les structures et les propriétés de l'ARN ainsi que dans les expériences de clivage ciblé de l'ARN, certaines en vue d'applications thérapeutiques. Ils tirent leur nom de la ressemblance des premières représentations de leur structure secondaire avec le requin marteau[2]. Ils ont d'abord été identifiés parmi des ARN satellites et des viroïdes puis se sont révélés être très largement distribués à travers de nombreuses formes de vie.
Les réactions d'auto-clivage, publiées pour la première fois en 1986[3] - [4], font partie d'un mécanisme de réplication circulaire. La séquence de ces ribozymes est suffisante pour l'auto-clivage[5] et agit en formant une structure tertiaire tridimensionnelle conservée.
Occurrence naturelle
Les premiers ribozymes en tête de marteau ont été découverts en 1986 dans certains agents infectieux de plantes à ARN tels que les viroïdes et des satellites[3] - [4]. Puis un autre a été identifié l'année suivante dans l'ADN satellite de génomes de tritons[6]. Puis d'autres exemples de ce ribozymes ont été trouvés dans le génome de différents organismes tels que des schistosomes[7], des grillons des cavernes[8], l'arabette des dames[9] et quelques mammifères tels que des rongeurs et l'ornithorynque[10]. En 2010, il est devenu évident que le ribozyme en tête de marteau est en réalité répandu dans le génome des bactéries jusqu'aux eucaryotes[11], y compris chez l'homme[12]. Des publications du même ordre ont par la suite confirmé et étendu ces observations[13] - [14] - [15], révélant la nature ubiquitaire du ribozyme en tête de marteau dans tous les règnes du vivant[16].
Dans les génomes d'eucaryotes, de nombreux ribozymes en tête de marteau semblent liés à des rétrotransposons (SINE)[11], hormis une famille étonnamment conservée trouvée dans le génome de tous les amniotes[12] ; ces derniers, appelés HH9 et HH10, se trouvent dans les introns de quelques gènes spécifiques et renvoient à une fonction biologique préservée au cours de la biosynthèse de l'ARN prémessager[17].
Structure primaire et secondaire
Ribozyme minimal
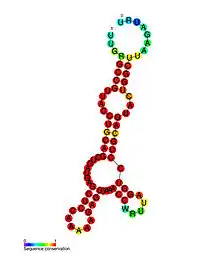
La séquence minimale requise pour la réaction d'auto-clivage comprend environ 13 nucléotides formant un « noyau », ou « cœur », conservé, la plupart de ces bases nucléiques n'étant pas impliquée dans des paires de bases canoniques de type Watson-Crick. Cette région est bordée par trois régions en tige-boucle constituées essentiellement de paires de bases canoniques de type Watson-Crick mais sans autre contrainte du point de vue de la séquence. La constante catalytique du ribozyme en tête de marteau minimal est d'environ 1 min−1 — un intervalle de valeurs de 0,1 min−1 à 10 min−1 est couramment observé selon les séquences non conservées et la longueur des trois tiges hélicoïdales — dans les conditions standard de réaction : concentration en Mg2+ d'environ 10 mmol l−1, pH de 7,5 et tepérature de 25 °C. La plupart des recherches sur les ribozymes en tête de marteau ont été réalisées sur le ribozyme minimal.
Type I, type II et type III
|
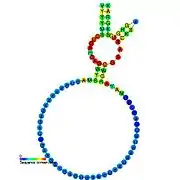
Le ribozyme en tête de marteau est structuré en trois hélices constituées de bases appariées, ces hélices étant liées entre elles par de courts segments aux séquences conservées. Ces hélices sont appelées I, II et III. Les ribozymes en tête de marteau peuvent être rangés en trois types en fonction du numéro de l'hélice dans laquelle se trouvent les extrémités 5' et 3' : on trouve le type I dans le génome de procaryotes, d'eucaryotes et d'agents infectieux à ARN de plantes, tandis que le type II n'a été décrit que chez des procaryotes[14] - [15] et le type III est trouvé essentiellement chez les plantes, les agents infectieux de plantes et les procaryotes[16] - [17].
Ribozyme complet
Le ribozyme en tête de marteau complet contient des séquences supplémentaires dans les tiges I et II qui permettent la formation de contacts intramoléculaires supplémentaires stabilisant la structure tertiaire active de la molécule et permettant d'atteindre des vitesses de clivage mille fois plus élevées que celles des séquences minimales[18] - [19].
Structure tertiaire
|
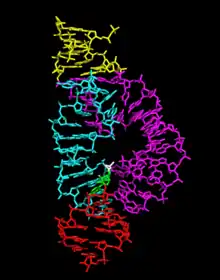
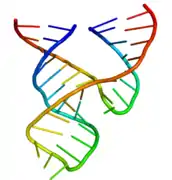
Le ribozyme en tête de marteau minimal a été intensivement étudié par les biochimistes ainsi que par cristallographie aux rayons X, spectroscopie RMN et d'autres techniques d'investigation biophysiques. La première structure tridimensionnelle détaillée d'un ribozyme minimal a été obtenue en 1994 à partir d'une diffraction par rayons X d'un ribozyme en tête de marteau lié à un analogue ADN de substrat[20], une structure entièrement en ARN étant publiée l'année suivante[21]. Cette dernière a mis en évidence trois hélices de ribonucléotides appariés, appelées I, II et III, séparées par de courtes séquences conservées : le connecteur entre les hélices I et II contient généralement la séquence CUGA tandis que celui situé entre les hélices II et III contient la séquence GAAA ; le site actif de la réaction de clivage se trouve entre les hélices I et III, où se trouve généralement un résidu de cytosine.
Cette structure cristallisée obtenue par diffraction aux rayons X s'est cependant révélée difficilement conciliable avec les contraintes géométriques déduites d'analyses par résonance magnétique nucléaire et d'expériences biochimiques destinées à étudier la cinétique et le mécanisme catalytique du ribozyme. Ainsi, les résidus invariants C3, G5, G8 et G12 du ribozyme minimal sont critiques au point que la moindre substitution d'un groupe fonctionnel exocyclique sur l'un de ces résidus conduit à une disparition quasi complète voire complète de toute activité catalytique, et ceci bien qu'ils ne semblent pas former de liaison hydrogène dans le cadre d'une paire Watson-Crick dans aucune des structures cristallisées de ribozyme en tête de marteau minimal — hormis pour le résidu G5. D'autres difficultés ont été mises en évidence, par exemple le rôle des résidus G8 et G12 dans la catalyse acide/basique du ribozyme que les structures cristallisées ne rendent pas évident, ou encore l'effet Overhauser nucléaire observé par résonance magnétique nucléaire entre les résidus U4 et U7, que la structure cristallisée ne permet pas d'expliquer car cet effet requiert une distance maximale de 6 Å difficilement compatible avec la structure obtenue par cristallographie aux rayons X.
Ces difficultés — et quelques autres, rendant difficilement conciliables les structures obtenues par cristallographie avec les contraintes géométriques déduites par d'autres approches — affectant la structure tertiaire des ribozymes en tête de marteau minimaux ont été largement résolues avec la structure cristallisée de ribozymes complets. Cette dernière montre de nombreuses interactions entre les boucles des tiges I et II, y compris à distance du site actif[1].
Site actif
La configuration tridimensionnelle du site actif du ribozyme en tête de marteau est stabilisée par de nombreuses interactions intramoléculaires du ribozyme complet, alignant l'oxygène 2' du résidu C17, qui constitue le nucléophile du site catalytique, presque parfaitement pour une substitution nucléophile bimoléculaire (SN2). Le résidu G12 est suffisamment proche de ce nucléophile pour établir une liaison hydrogène avec lui et permettre de soustraire un proton de l'oxygène 2' si le résidu G12 est lui-même déprotoné. L'hydroxyle 2' du résidu G8 établit une liaison hydrogène avec l'oxygène du groupe situé en 5' et est par conséquent susceptible de fournir un proton lorsque les charges négatives s'accumulent sur l'oxygène 5' du ribose du résidu A1.1.
Le remplacement du résidu G8 par un résidu C8 (mutation G8C) fait disparaître toute activité catalytique au ribozyme en tête de marteau, tandis qu'une double mutation C3G + G8C en conserve l'essentiel ; le remplacement du résidu G8 par un résidu désoxyG8 en réduit très sensiblement l'activité catalytique, ce qui suggère que l'hydroxyle 2' de ce résidu est bel et bien critique pour son mécanisme réactionnel.
Le rapprochement du résidu A9 et des phosphates scissiles (en) requiert une concentration élevée en charges positives, d'où l'observation que les ions métalliques bivalents tels que Mg2+ sont nécessaires à faible concentration ionique, mais peuvent être dispersés à plus forte concentration de cations monovalents. La réaction fait ainsi probablement intervenir l'extraction du proton 2' du résidu C17 puis l'attaque nucléophile du phosphate adjacent. Le ribose du résidu G8 fournit alors un proton à la rupture de la liaison entre le phosphore scissile et l'oxygène 5' du groupe partant puis est reprotoné à partir d'une molécule d'eau.
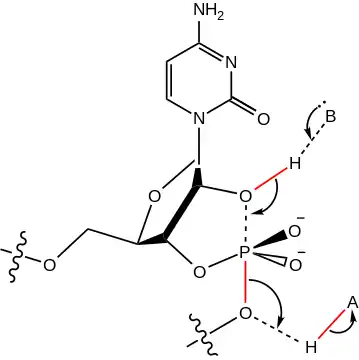 Mécanisme réactionnel proposé pour le site actif du ribozyme en tête de marteau. Une base (B) retire un proton de l'oxygène 2' tandis qu'un acide (A) fournit un proton au groupe partant 5' ce qui donne un phosphate cyclique 2',3'. Les liaisons rompues (en rouge) et formées (en pointillés) doivent être en position axiale à environ 180° de part et d'autre. Le mouvement des électrons est représenté par des flèches.
Mécanisme réactionnel proposé pour le site actif du ribozyme en tête de marteau. Une base (B) retire un proton de l'oxygène 2' tandis qu'un acide (A) fournit un proton au groupe partant 5' ce qui donne un phosphate cyclique 2',3'. Les liaisons rompues (en rouge) et formées (en pointillés) doivent être en position axiale à environ 180° de part et d'autre. Le mouvement des électrons est représenté par des flèches.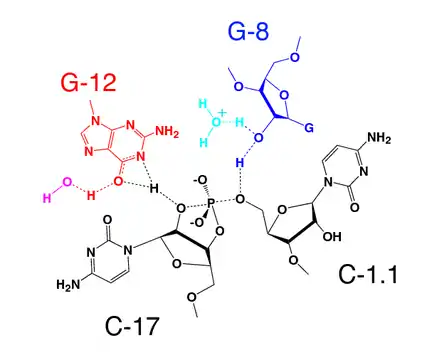 Interactions possible de l'état de transition telles qu'extrapolées à partir de la structure du ribozyme cristallisé.
Interactions possible de l'état de transition telles qu'extrapolées à partir de la structure du ribozyme cristallisé.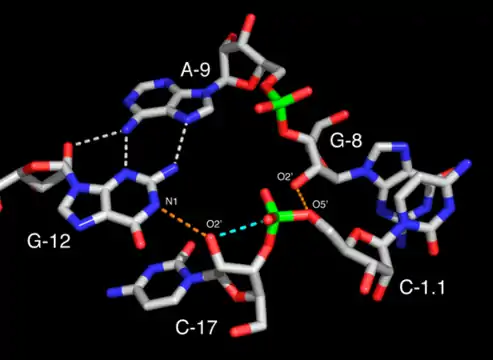 Site actif d'un ribozyme en tête de marteau complet. Le résidu G12 est positionné de façon cohérente avec son rôle de base dans la réaction de clivage tandis que l'hydroxyle 2' du résidu G8 est positionné conformément à son rôle d'acide pour une catalyse acide/basique. Les liaisons hydrogène potentiellement « actives » sont soulignées en pointillés orange. L'oxygène 2' du résidu C17 apparaît aligné pour une attaque nucléophile le long de la trajectoire matérialisée par une ligne pointillée bleue.
Site actif d'un ribozyme en tête de marteau complet. Le résidu G12 est positionné de façon cohérente avec son rôle de base dans la réaction de clivage tandis que l'hydroxyle 2' du résidu G8 est positionné conformément à son rôle d'acide pour une catalyse acide/basique. Les liaisons hydrogène potentiellement « actives » sont soulignées en pointillés orange. L'oxygène 2' du résidu C17 apparaît aligné pour une attaque nucléophile le long de la trajectoire matérialisée par une ligne pointillée bleue.
Notes et références
- (en) Monika Martick et William G. Scott, « Tertiary Contacts Distant from the Active Site Prime a Ribozyme for Catalysis », Cell, vol. 126, no 2, , p. 309-320 (PMID 16859740, DOI 10.1016/j.cell.2006.06.036, lire en ligne)
- (en) Anthony C. Forster et Robert H. Symons, « Self-cleavage of plus and minus RNAs of a virusoid and a structural model for the active sites », Cell, vol. 49, no 2, , p. 211-220 (PMID 2436805, DOI 10.1016/0092-8674(87)90562-9, lire en ligne)
- (en) Gerry A. Prody, John T. Bakos, Jamal M. Buzayan, Irving R. Schneider et George Bruening, « Autolytic Processing of Dimeric Plant Virus Satellite RNA », Science, vol. 231, no 4745, , p. 1577-1580 (PMID 17833317, DOI 10.1126/science.231.4745.1577, lire en ligne)
- (en) Cheryl J. Hutchins, Peter D. Rathjen, Anthony C. Forster et Robert H. Symons, « Self-cleavage of plus and minus RNA transcripts of avocado sunblotch viroid », Nucleid Acid Research, vol. 14, no 9, , p. 3627-3640 (PMID 3714492, DOI 10.1093/nar/14.9.3627, lire en ligne)
- (en) Anthony C. Forster et Robert H. Symons, « Self-cleavage of virusoid RNA is performed by the proposed 55-nucleotide active site », Cell, vol. 50, no 1, , p. 9-16 (PMID 3594567, DOI 10.1016/0092-8674(87)90657-X, lire en ligne)
- (en) Lloyd M. Epstein et Joseph G. Gall, « Self-cleaving transcripts of satellite DNA from the newt », Cell, vol. 48, no 3, , p. 535-543 (PMID 2433049, DOI 10.1016/0092-8674(87)90204-2, lire en ligne)
- (en) Gerardo Ferbeyre, James M. Smith et Robert Cedergren, « Schistosome Satellite DNA Encodes Active Hammerhead Ribozymes », Molecular and Cellular Biology, vol. 18, no 7, , p. 3880-3888 (PMID 9632772, PMCID 108972, lire en ligne)
- (en) Andres A. Rojas, Alejandro Vazquez-Tello, Gerardo Ferbeyre, Federica Venanzetti, Lutz Bachmann, Bruno Paquin, Valerio Sbordoni et Robert Cedergren, « Hammerhead-mediated processing of satellite pDo500 family transcripts from Dolichopoda cave crickets », Nucleid Acid Research, vol. 28, no 20, , p. 4037-4043 (PMID 11024185, PMCID 110794, DOI 10.1093/nar/28.20.4037, lire en ligne)
- (en) Rita Przybilski, Stefan Gräf, Aurélie Lescoute, Wolfgang Nellen, Eric Westhof, Gerhard Steger et Christian Hammann, « Functional Hammerhead Ribozymes Naturally Encoded in the Genome of Arabidopsis thaliana », The Plant Cell, vol. 17, no 7, , p. 1877-1885 (PMID 15937227, PMCID 1167538, DOI 10.1105/tpc.105.032730, lire en ligne)
- (en) Monika Martick, Lucas H. Horan, Harry F. Noller et William G. Scott, « A discontinuous hammerhead ribozyme embedded in a mammalian messenger RNA », Nature, vol. 454, no 7206, , p. 899-902 (PMID 18615019, PMCID 2612532, DOI 10.1038/nature07117, lire en ligne)
- (en) Marcos de la Peña et Inmaculada García-Robles, « Ubiquitous presence of the hammerhead ribozyme motif along the tree of life », RNA, vol. 16, no 10, , p. 1943-1950 (PMID 20705646, PMCID 2941103, DOI 10.1261/rna.2130310, lire en ligne)
- (en) Marcos de la Peña et Inmaculada García‐Robles, « Intronic hammerhead ribozymes are ultraconserved in the human genome », EMBO reports, vol. 11, no 9, , p. 711-716 (PMID 20651741, PMCID 2933863, DOI 10.1038/embor.2010.100, lire en ligne)
- (en) Carsten Seehafer, Anne Kalweit, Gerhard Steger, Stefan Gräf et Christian Hammann, « From alpaca to zebrafish: Hammerhead ribozymes wherever you look », RNA, vol. 17, no 1, , p. 21-26 (PMID 21081661, PMCID 3004062, DOI 10.1261/rna.2429911, lire en ligne)
- (en) Randi M. Jimenez, Eric Delwart et Andrej Lupták, « Structure-based Search Reveals Hammerhead Ribozymes in the Human Microbiome », Journal of Biological Chemistry, vol. 286, no 10, , p. 7737-7743 (PMID 21257745, PMCID 3048661, DOI 10.1074/jbc.C110.209288, lire en ligne)
- (en) Jonathan Perreault, Zasha Weinberg, Adam Roth, Olivia Popescu, Pascal Chartrand, Gerardo Ferbeyre et Ronald R. Breaker, « Identification of Hammerhead Ribozymes in All Domains of Life Reveals Novel Structural Variations », PLoS Computational Biology, vol. 7, no 5, , e1002031 (PMID 21573207, PMCID 3088659, DOI 10.1371/journal.pcbi.1002031, lire en ligne)
- (en) Christian Hammann, Andrej Luptak, Jonathan Perreault et Marcos de la Peña, « The ubiquitous hammerhead ribozyme », RNA, vol. 18, no 5, , p. 871-885 (PMID 22454536, PMCID 3334697, DOI 10.1261/rna.031401.111, lire en ligne)
- (en) Inmaculada García-Robles, Jesús Sánchez-Navarro et Marcos de la Peña, « Intronic hammerhead ribozymes in mRNA biogenesis », Biological Chemistry, vol. 393, no 11, , p. 1317-1326 (PMID 23109545, DOI 10.1515/hsz-2012-0223, lire en ligne)
-
(en) Anastasia Khvorova, Aurélie Lescoute, Eric Westhof et Sumedha D. Jayasena, « Sequence elements outside the hammerhead ribozyme catalytic core enable intracellular activity », Nature Structural & Molecular Biology, vol. 10, no 9, , p. 708-712 (PMID 12881719, DOI 10.1038/nsb959, lire en ligne)
(en) Anastasia Khvorova, Aurélie Lescoute, Eric Westhof et Sumedha D Jayasena, « Corrigendum: Sequence elements outside the hammerhead ribozyme catalytic core enable intracellular activity », Nature Structural Biology, vol. 10, no 10, (DOI 10.1038/nsb1003-872a, lire en ligne) - (en) Marcos De la Peña, Selma Gago et Ricardo Flores, « Peripheral regions of natural hammerhead ribozymes greatly increase their self‐cleavage activity », EMBO Journal, vol. 22, no 20, , p. 5561-5570 (PMID 14532128, PMCID 213784, DOI 10.1093/emboj/cdg530, lire en ligne)
- (en) Heinz W. Pley, Kevin M. Flaherty et David B. McKay, « Three-dimensional structure of a hammerhead ribozyme », Nature, vol. 372, no 6501, , p. 68-74 (PMID 7969422, DOI 10.1038/372068a0, lire en ligne)
- (en) William G. Scott, John T. Finch et Aaron Klug, « The crystal structure of an AII-RNAhammerhead ribozyme: A proposed mechanism for RNA catalytic cleavage », Cell, vol. 81, no 7, , p. 991-1002 (PMID 7541315, DOI 10.1016/S0092-8674(05)80004-2, lire en ligne)