Mononegavirales
Les Mononegavirales sont un ordre de virus à ARN monocaténaire de polarité négative (groupe V de la classification Baltimore) et à génome non segmenté. On y trouve notamment les familles suivantes :
- Bornaviridae (le virus de la maladie de Borna)
- Rhabdoviridae (par exemple le virus de la rage)
- Filoviridae (par exemple le virus Marburg, le virus Ebola)
- Paramyxoviridae (par exemple le virus de la maladie de Newcastle, virus de la rougeole, virus Nipah).
| Royaume | Riboviria |
|---|---|
| Règne | Orthornavirae |
| Embranchement | Negarnaviricota |
| Sous-embr. | Haploviricotina |
| Classe | Monjiviricetes |
Familles de rang inférieur
- Artoviridae
- Bornaviridae
- Filoviridae
- Lispiviridae
- Mymonaviridae
- Nyamiviridae
- Paramyxoviridae
- Pneumoviridae
- Rhabdoviridae
- Sunviridae
- Xinmoviridae
Un nombre important de maladies humaines, établies (oreillons, rougeole, rage) ou émergentes (maladie à virus Ebola, fièvre hémorragique, maladie de Borna, infection à virus Nipah ou à virus Hendra, sont causées par des virus de cette catégorie.
Structure
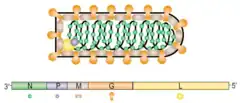
Les virions possèdent une enveloppe et une hélice de nucléocapsides contenant de l'ARN génomique et une ARN polymérase ARN-dépendante. Le génome contient de 6 à 10 gènes, qui peuvent coder davantage de protéines à travers un processus d'édition des ARN messagers.
Structure taxinomique
| Famille | Sous-famille | Genre | Espèce (* signifie « espèce-type ») |
| Bornaviridae | Virus de la maladie de Borna | Virus de la maladie de borna* | |
| Filoviridae | Marburgvirus | Marburgvirus du lac Victoria * | |
| Ebolavirus | ebolavirus Zaire * | ||
| ebolavirus Côte d'Ivoire | |||
| ebolavirus de Reston | |||
| ebolavirus Soudan | |||
| Paramyxoviridae | Paramyxovirinae | Respirovirus | Virus bovin parainfluenza 3 |
| Virus humain parainfluenza 3 | |||
| Virus humain parainfluenza 1 | |||
| Virus de Sendai* | |||
| Virus Siméen 10 | |||
| Morbillivirus | Virus canin de la maladie de Carré | ||
| Morbillivirus des cétacés | |||
| Virus des dauphins de la maladie de Carré | |||
| virus de la rougeole * | |||
| virus de la Peste-des-petits-ruminants | |||
| Phocine distemper virus | |||
| Virus de la peste bovine | |||
| Rubulavirus | Virus humain parainfluenza 2 | ||
| Virus humain parainfluenza 4 | |||
| Virus de l'homme parainfluenza 4a | |||
| Virus Mapuera | |||
| Virus ourlien ou virus des Oreillons* | |||
| rubulavirus Porcin | |||
| Virus siméen 5 | |||
| Virus siméen 41 | |||
| Henipavirus | Virus Hendra * | ||
| Virus Nipah | |||
| Avulavirus | paramyxovirus aviaire | ||
| Virus de la maladie de Newcastle | |||
| Pneumovirinae | Pneumovirus | Virus syncytial du système respiratoire humain* | |
| Virus syncytial du système respiratoire bovin | |||
| Virus de la pneumonie murine | |||
| Metapneumovirus | Metapneumovirus aviaire* | ||
| Metapneumovirus humain | |||
| Virus de la famille paramyxovirale qui ne sont pas assignés à un genre | paramyxovirus Tupaia | ||
| Virus Fer-de-Lance | |||
| Menangle virus | |||
| Nariva virus | |||
| Tioman virus | |||
| Rhabdoviridae | Vesiculovirus | Carajas virus | |
| Chandipura virus | |||
| Cocal virus | |||
| Isfahan virus | |||
| Maraba virus | |||
| Piry virus | |||
| Virus de la stomatite vésiculeuse Alagoas | |||
| Virus de la stomatite indienne* | |||
| Virus de la stomatite de New Jersey | |||
| Lyssavirus | Lyssavirus de la chauve souris australienne | ||
| Duvenhage virus | |||
| Lyssavirus de la chauve-souris européenne | |||
| Virus de la chauve-souris de Lagos | |||
| Mokola virus | |||
| Virus de la rage* | |||
| Ephemerovirus | Adelaide River virus | ||
| Berrimah virus | |||
| Virus de la fièvre éphémère bovine * | |||
| Novirhabdovirus | Hirame rhabdovirus | ||
| Virus de la nécrose hématopoïétique | |||
| Septicémie hémorragique virale | |||
| Virus de la tête de serpent | |||
| Cytorhabdovirus | Barley yellow striate mosaic virus | ||
| Broccoli necrotic yellows virus | |||
| Festuca leaf streak virus | |||
| Lettuce necrotic yellows virus * | |||
| Northern cereal mosaic virus | |||
| Sonchus virus | |||
| Strawberry crinkle virus | |||
| Wheat American striate mosaic virus | |||
| Nucleorhabdovirus | Datura yellow vein virus | ||
| Eggplant mottled dwarf virus | |||
| Maize mosaic virus | |||
| Potato yellow dwarf virus * | |||
| Rice yellow stunt virus | |||
| Sonchus yellow net virus | |||
| Sowthistle yellow vein virus | |||
| De nombreux virus dans la famille des rabdoviridae qui ne sont pas assignés à un genre | |||
Cycle de vie
Entrée
Les particules virales pénètrent une cellule en se liant à un récepteur membranaire (par exemple l'acide sialique) et induisent la fusion entre l'enveloppe virale et la membrane cellulaire. Les éléments qui composent l'intérieur du virus pénètrent alors le cytoplasme.
Synthèse d'ARNm
La séquence génomique ne code pas des protéines. Les séquences complémentaires doivent d'abord être transcrites par de l'ARN-polymérase. Cette incapacité du génome à la production de protéines est dû au transport, par le virion, du RDRP. Ceci contraste avec le volet positif des virus qui peuvent synthétiser le RDRP une fois à l'intérieur de la cellule.
Le RDRP libéré de la particule virale se lie à l'unique site promoteur à l'extrémité 3' du génome et commence la transcription. Il se crée une polarité de transcription, où les gènes près de l'extrémité 3' du génome sont transcrits en plus grande abondance, contrairement à celles vers l'extrémité 5' (les moins susceptibles d'être transcrites). le virus est en mesure d'utiliser cette polarité comme une forme de régulation transcriptionnelle. En conséquence, la plupart des génomes commencent avec le gène de la protéine nucléocapsides et finissent avec le gène de la RDRP.
La synthèse des protéines et du génome
Une fois que la production de l'ARNm a commencé, la protéine de traduction cellulaire est utilisée pour produire des protéines virales qui s'accumulent dans le cytoplasme.
Assemblage et sortie
Les nouvelles protéines virales et le génome s'auto-assemblent près de la membrane cellulaire. Les nouveaux virions utilisent la membrane à leur avantage, en prélevant une partie de celle-ci. La nouvelle particule virale infecte une autre cellule et le cycle se répète.
Édition de l'ARN
L'édition de l'ARN est un mécanisme utilisé par certains membres du genre mononegavirales pour produire plusieurs protéines à partir d'un seul gène. Cela se produit lorsque l'ARN polymérase-ARN dépendante insère des résidus supplémentaires dans l'ARNm. Cela crée un décalage du cadre de lecture ouvert et modifie donc la séquence d'acides aminés codés par l'ARNm.
Évolution
Comme avec d'autres rétrovirus, qui n'ont pas un intermédiaire ADN, les espèces de Mononegavirales sont capables d'évoluer à un rythme rapide. Un haut taux de mutation se produit dans la production de nouveaux génomes (jusqu'à 1 pour 1000 bases).
Notes et références
Référence biologique
- (en) Référence ICTV : Mononegavirales (consulté le )
Bibliographie
- C. Büchen-Osmond, 2003, « 01. Mononegavirales », in ICTVdB - The Universal Virus Database, version 3, sur www.ncbi.nlm.nih.gov (site accédé le ).
- S. Dewhurst, 2003, University of Rochester Medical Center Department of Microbiology and Immunology Selected Virology Lecture Notes, sur www.urmc.rochester.edu (document PDF accédé le )