Complexe pyruvate déshydrogénase
Le complexe pyruvate déshydrogénase (PDC) est l'association de trois enzymes intervenant séquentiellement pour catalyser la décarboxylation oxydative du pyruvate en acétyl-CoA. Le pyruvate est notamment issu de la glycolyse, tandis que l'acétyl-CoA est, par exemple, le point d'entrée du cycle de Krebs, qui fonctionne dans la foulée de la chaîne respiratoire pour parachever l'oxydation du groupement acétyle de l'acétyl-CoA.
| N° EC | EC |
|---|---|
| N° CAS | |
| Cofacteur(s) | TPP |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
S-acétyltransférase (E2)
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
| N° EC | EC |
|---|---|
| N° CAS | |
| Cofacteur(s) | FAD |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
Le PDC est structurellement apparenté au complexe α-cétoglutarate déshydrogénase et au complexe 3-méthyl-2-oxobutanoate déshydrogénase, constitués comme lui d'une décarboxylase, d'une acyltransférase et d'une oxydo-réductase. Chez les procaryotes, il est organisé selon une symétrie qui varie suivant les espèces, cubique chez les bactéries à Gram négatif, avec 24 sous-unités E2[1], et dodécaédrique chez les bactéries à Gram positif, avec 60 sous-unité E2[2]. Chez les eucaryotes, on le trouve dans la matrice mitochondriale, l'enzyme humaine étant constituée de 96 sous-unités organisées selon une symétrie dodécaédrique[3].
Enzyme Abrév. Cofacteurs Nombre de sous-unités Procaryotes Eucaryotes Pyruvate déshydrogénase
EC : décarboxylaseE1 Thiamine pyrophosphate
(TPP)24 30 Dihydrolipoamide S-acétyltransférase
EC : acyltransféraseE2 Lipoamide / dihydrolipoamide
Coenzyme A (CoA-SH)24-60 48-60[4] Dihydrolipoyl déshydrogénase
EC : oxydo-réductaseE3 Flavine adénine dinucléotide (FAD)
Nicotinamide adénine dinucléotide (NAD+)12 12
La réaction globale catalysée par le complexe pyruvate déshydrogénase est la suivante :
Le mécanisme de cette réaction, qui fait intervenir successivement les enzymes E1, E2 et E3, chacune avec ses cofacteurs, est assez complexe, et peut être résumé par le schéma simplifié ci-dessous :
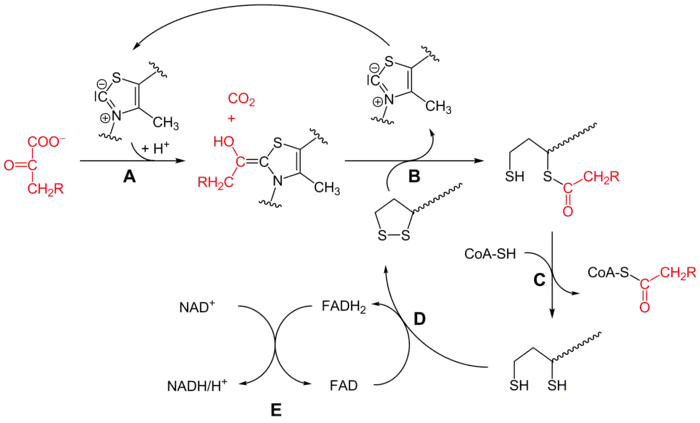
- la pyruvate déshydrogénase (E1) catalyse les étapes A et B avec la thiamine pyrophosphate (TPP),
- la dihydrolipoamide S-acétyltransférase (E2) catalyse l'étape C avec le lipoamide et la coenzyme A (CoA-SH),
- la dihydrolipoyl déshydrogénase (E3) catalyse les étapes D et E avec la FAD et la NAD+.
Notes et références
- A. Mattevi, G. Obmolova, E. Schulze, K. H. Kalk, A. H. Westphal, A. de Kok, W. G. Hol « Atomic structure of the cubic core of the pyruvate dehydrogenase multienzyme complex », dans Science, vol. 255, 1544-1550, 1992.
- (en) Ute Neveling, Stephanie Bringer-Meyer et Hermann Sahm, « Gene and subunit organization of bacterial pyruvate dehydrogenase complexes », Biochim. Biophys. Acta, vol. 1385, , p. 367-372 (PMID 9655937)
- M. Smolle, A. E. Prior, A. E. Brown, A. Cooper, O. Byron, J. G. Lindsay, « A new level of architectural complexity in the human pyruvate dehydrogenase complex », dans J. Biol. Chem. vol. 281, 19772-19780, 2006.
-
(en) S. Vijayakrishnan, S. M. Kelly, R. J. C. Gilbert, P. Callow, D. Bhella, T. Forsyth, J. G. Lindsay et O. Byron, « Solution Structure and Characterisation of the Human Pyruvate Dehydrogenase Complex Core Assembly », Journal of Molecular Biology, vol. 339, no 1, , p. 71-93 (ISSN 0022-2836, lire en ligne)
DOI 10.1016/j.jmb.2010.03.043