Sérotonine N-acétyltransférase
Aralkylamine N-acétyltransférase (AANAT) (EC ), également appelée arylalkylamine N-acétyltransférase ou sérotonine N-acétyltransférase (SNAT), est une enzyme impliquée dans la production rythmique jour/nuit de mélatonine, par modification de la sérotonine. C'est chez l'homme codé par le gène AANAT d'environ 2,5 kb [1] contenant quatre exons, situés sur le chromosome 17q 25[2]. Le gène est traduit en une grande enzyme de 23 kDa. Il est bien conservé au cours de l'évolution et la forme humaine de la protéine est identique à 80 % à l'AANAT de mouton et de rat. Il s'agit d'une enzyme dépendante de l'acétyl-CoA de la famille des N-acétyltransférases (GNAT) apparentée à GCN5. Il peut contribuer à des maladies génétiques multifactorielles telles que l'altération du comportement dans le cycle veille/sommeil [1] et des recherches sont en cours dans le but de développer des médicaments qui régulent la fonction AANAT.
Nomenclature
Le nom systématique de cette classe d'enzymes est l'acétyl-CoA:2-aryléthylamine N-acétyltransférase. Les autres noms d'usage courant incluent:
- AANAT
- Arylalkylamine N-acétyltransférase
- Enzyme rythmique de la mélatonine
- Sérotonine acétylase
- Sérotonine acétyltransférase
- Sérotonine N-acétyltransférase
Le nom officiellement accepté est l'aralkylamine N-acétyltransférase[3].
Fonction et mécanisme
Répartition tissulaire
Le transcrit d'ARNm de l'AANAT est principalement exprimé dans le système nerveux central (SNC). Il est détectable à de faibles niveaux dans plusieurs régions du cerveau, dont l'hypophyse, ainsi que dans la rétine. Il est le plus abondant dans la glande pinéale qui est le site de synthèse de la mélatonine. L'AANAT cérébrale et hypophysaire peut être impliquée dans la modulation des aspects dépendants de la sérotonine du comportement humain et de la fonction hypophysaire[2].
Fonction physiologique
Dans les cellules pinéalocytes de la glande pinéale, l'aralkylamine N-acétyltransférase est impliquée dans la conversion de la sérotonine en mélatonine. C'est l'avant-dernière enzyme de la synthèse de la mélatonine qui contrôle le rythme jour/nuit de la production de mélatonine dans la glande pinéale des vertébrés. La mélatonine est essentielle à la reproduction saisonnière, module la fonction de l'horloge circadienne dans le noyau suprachiasmatique et influence l'activité et le sommeil. En raison de son rôle important dans le rythme circadien, l'AANAT est soumis à une régulation importante qui réagit à l'exposition à la lumière (voir Régulation). Il peut contribuer à des maladies génétiques multifactorielles telles qu'une altération du comportement dans le cycle veille/sommeil et des troubles de l'humeur[1].
Réactions chimiques catalysées par AANAT
La réaction chimique primaire qui est catalysée par l'aralkylamine N-acétyltransférase utilise deux substrats, l'acétyl-CoA et la sérotonine. L'AANAT catalyse le transfert du groupe acétyle de l'acétyl-CoA vers l'amine primaire de la sérotonine, produisant ainsi du CoA et de la N-acétylsérotonine. Chez l'homme, d'autres substrats endogènes de l'enzyme comprennent des neuromodulateurs spécifiques d'amine trace, à savoir la phénéthylamine, la tyramine et la tryptamine, formant à leur tour la N-acétylphénéthylamine, la N-acétyltyramine et la N-acétyltryptamine[4].
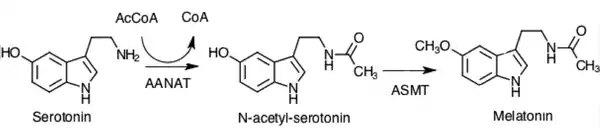
Dans la biosynthèse de la mélatonine, la N-acétylsérotonine est ensuite méthylée par une autre enzyme, la N-acétylsérotonine O-méthyltransférase (ASMT) pour générer de la mélatonine. La réaction de la N-acétyltransférase est suggérée comme étant l'étape déterminant la vitesse, et ainsi la sérotonine N-acétyltransférase est devenue une cible pour la conception d'inhibiteurs[5].
AANAT obéit à un mécanisme ternaire-complexe ordonné. Les substrats se lient séquentiellement (dans l'ordre) avec la liaison de l'acétyl-CoA à l'enzyme libre suivie de la liaison de la sérotonine pour former le complexe ternaire. Une fois le transfert du groupe acétyle effectué, les produits sont libérés de manière ordonnée avec la N-acétyl-sérotonine en premier et la CoA en dernier[6].
Structure
L'arylkylamine N-acétyltransférase est un polypeptide monomère d'une longueur de 207 résidus d'acides aminés et d'un poids moléculaire de 23 344 daltons. La structure secondaire est constituée d'hélices alpha et de feuillets bêta. Il est à 28 % hélicoïdal (10 hélices ; 60 résidus) et à 23 % en feuillet bêta (9 brins ; 48 résidus). Cette famille partage quatre motifs de séquence conservés désignés AD. Le motif B sert d'emplacement de la fente de liaison à la sérotonine. La structure est déterminée par diffraction des rayons X[7].
Plusieurs structures sont résolues pour cette classe d'enzymes, avec les codes d'accession PDB 1CJW[8] 1B6B[9] 1L0C[7] - [10] et 1KUV 1KUX 1KUY[7].
L'aralkylamine N-acétyltransférase est cristallisée en complexe avec 14-3-3ζ de la famille des protéines 14-3-3, avec le code d'accès PDB 1IB1[11].
La superfamille GNAT
L'aralkylamine N-acétyltransférase appartient à la superfamille des N-acétyltransférases liées à GCN5 (GNAT) qui comprend 10 000 acétyltransférases, nommées ainsi en raison de leur homologie de séquence avec une classe de facteurs de transcription eucaryotes, la levure GCN5. D'autres membres bien étudiés de la superfamille sont la glucosamine-6-phosphate N-acétyltransférase et les histones acétyltransférases.
Tous les membres de cette superfamille ont une structure conservée qui consiste en un brin N-terminal suivi de deux hélices, trois brins β antiparallèles, suivis d'une hélice centrale "signature", d'un cinquième brin β, d'une quatrième hélice α et d'un dernier brin β. Ces éléments sont presque universellement conservés en dépit d'une faible identité par paire dans les alignements de séquences[12].
Régulation
La régulation de l'AANAT varie selon les espèces. Chez certains, les niveaux d'AANAT oscillent considérablement entre les périodes claires et sombres, et contrôlent ainsi la synthèse de mélatonine. Dans d'autres, le rythme est régulé principalement au niveau des protéines[13]. Un exemple est chez les rongeurs, où les niveaux d'ARNm d'AANAT augmentent de plus de 100 fois pendant les périodes d'obscurité. Chez d'autres espèces, l'AMP cyclique joue un rôle important dans l'inhibition de la dégradation protéolytique de l'AANAT, élevant les niveaux de protéines la nuit. Des expériences utilisant l'AANAT humain exprimé dans une lignée cellulaire 1E7 montrent une augmentation d'environ 8 fois de l'activité enzymatique lors de l'exposition à la forskoline[14].
La dégradation dynamique de l'ARNm de l'AANAT s'est avérée essentielle à l'action circadienne de l'enzyme. Les séquences 3'UTR ont une importance en ce qui concerne la dégradation rythmique de l'ARNm de l'AANAT chez certaines espèces. Chez les rongeurs, divers hnRNP maintiennent la dégradation dynamique de l'ARNm de l'AANAT. Chez d'autres espèces, telles que les ongulés et les primates, les ARNm stables de l'AANAT avec un 3'UTR plus court sont soupçonnés de ne pas être sous le contrôle des hnRNP qui se lient et dirigent la dégradation de l'ARNm de l'AANAT chez les rongeurs[15].
L'exposition à la lumière induit des signaux à partir des cellules rétiniennes, entraînant finalement une baisse de la stimulation de la noradrénaline de la glande pinéale. Ceci, à son tour, conduit à une cascade de signalisation, entraînant la phosphorylation de la protéine kinase À de deux résidus clés Ser et Thr de la sérotonine N-acétyltransférase. La phosphorylation de ces résidus provoque des modifications de l'activité catalytique par recrutement et interaction avec les protéines 14-3-3, en particulier 14-3-3ζ[16].
Une autre protéine qui interagit et régule l'activité de l'AANAT est la protéine kinase C. La protéine kinase C agit, comme la protéine kinase A, sur les résidus thréonine et sérine, améliorant la stabilité et l'activité enzymatique de l'AANAT[17].
L'inhibition de la liaison de l'acétyl-CoA au site catalytique par la formation et le clivage de liaisons disulfure intramoléculaires est suggérée comme étant un mécanisme de régulation. La formation d'une liaison disulfure entre deux résidus de cystéine dans la protéine ferme l'entonnoir hydrophobe du site catalytique et agit ainsi comme un interrupteur marche/arrêt pour l'activité catalytique. Il n'est pas encore certain que ce mécanisme soit présent dans les cellules in vivo par la régulation des conditions redox intracellulaires, mais il est suggéré que le glutathion (GSH) pourrait être un régulateur in vivo de la formation et du clivage de ces liaisons disulfure[18].
Inhibiteurs de l'AANAT et pertinence clinique
Les inhibiteurs de l'AANAT pourraient éventuellement conduire au développement d'un médicament qui serait utile dans la recherche sur la biologie circadienne et dans le traitement des troubles du sommeil et de l'humeur. Des inhibiteurs synthétiques de l'enzyme sont découverts[19] - [20] - [21]. Cependant, aucun inhibiteur de l'AANAT avec une puissante activité in vivo n'est rapporté[22]. À ce jour, cinq classes d'inhibiteurs de l'AANAT sont décrites dans la littérature[5]. Ci-dessous les cinq classes :
Dérivés de la mélatonine
Puisqu'il est rapporté que la mélatonine est un inhibiteur compétitif de l'AANAT, ce neurotransmetteur semble exercer un contrôle autorégulateur sur sa propre biosynthèse. Ainsi, des analogues structurels lâches de l'hormone indolamine sont évalués sur AANAT et des inhibiteurs modérés sont découverts[23].
Inhibiteurs peptidiques
Des bibliothèques combinatoires de peptides de tri-, tétra- et pentapeptides avec diverses compositions d'acides aminés sont criblées en tant que sources potentielles d'inhibiteurs, pour voir si elles servent d'inhibiteur compétitif pur ou mixte pour l'enzyme hAANAT. La modélisation moléculaire et les études de relation structure-activité ont permis d'identifier le résidu d'acide aminé de l'inhibiteur pentapeptidique S 34461 qui interagit avec le site de liaison au cosubstrat[24].
Analogues bisubstrat
Il est suggéré que l'AANAT catalyse le transfert d'un groupe acétyle de l'acétyl-CoA à la sérotonine, avec l'implication d'un complexe ternaire intermédiaire, pour produire la N-acétylsérotonine. Sur la base de ce mécanisme, on pourrait s'attendre à ce qu'un inhibiteur analogue de bisubstrat, dérivé de la liaison des parties indole et CoASH, puisse potentiellement imiter le complexe ternaire et exercer une forte inhibition de l'AANAT[25]. Le premier analogue de bisubstrat (1), qui relie la tryptamine et le CoA via un pont acétyle, est synthétisé par Khalil et Cole et s'est avéré être un inhibiteur de l'AANAT très puissant et spécifique[26].
Dérivés N-haloacétylés
L'AANAT montre qu'elle possède également une activité alkyltransférase secondaire ainsi qu'une activité acétyltransférase[27]. Les N-haloacétyltryptamines sont développées et servent de substrats à l'AANAT alkyltransférase et sont également de puissants inhibiteurs in vitro (faible micromolaire) contre l'activité de l'AANAT acétyltransférase. L'AANAT catalyse la réaction entre la N-bromoacétyltryptamine (BAT) et la CoA réduite, ce qui donne un inhibiteur analogue de bisubstrat à liaison étroite[27] - [28]. Le premier inhibiteur perméable aux cellules synthétisé de l'AANAT N-bromoacétyltryptamine est étudié plus avant sur la sécrétion de mélatonine par les glandes pinéales de rat et de porc[29]. Nouveaux dérivés N-halogénoacétylés conduisant à une forte inhibition in situ de l'AANAT. Le concept derrière le mécanisme d'action de ces précurseurs est étudié en suivant la biosynthèse de l'inhibiteur à partir de tritiated-BAT dans une cellule vivante[20].
Composés à base de rhodanine
Les premiers inhibiteurs médicamenteux et sélectifs de l'AANAT sont identifiés. Lawrence M. Szewczuk et al. ont virtuellement criblé plus d'un million de composés par ancrage 3D à haut débit dans le site actif de la structure des rayons X pour l'AANAT, puis testé 241 composés en tant qu'inhibiteurs. Une classe de composés contenant un échafaudage de rhodanine montre une faible inhibition compétitive micromolaire contre l'acétyl-CoA et s'est avérée efficace pour bloquer la production de mélatonine dans les cellules pinéales[19].
L'étude récente sur l'inhibiteur de l'AANAT décrit la découverte d'une nouvelle classe d'inhibiteurs non peptidiques de l'AANAT basés sur un échafaudage 2,2′-bithiényle[22].
Articles connexes
Notes et références
- « Entrez Gene: arylalkylamine N-acetyltransferase »
- « The human serotonin N-acetyltransferase (EC 2.3.1.87) gene (AANAT): structure, chromosomal localization, and tissue expression », Genomics, vol. 34, no 1, , p. 76–84 (PMID 8661026, DOI 10.1006/geno.1996.0243)
- « IUBMB Enzyme Nomenclature EC 2.3.1.87 », Nomenclature Committee of the International Union of Biochemistry and Molecular Biology (NC-IUBMB) (consulté le )
- « EC 2.3.1.87 - aralkylamine N-acetyltransferase », BRENDA, Technische Universität Braunschweig, (consulté le )
- « Serotonin N-acetyltransferase: mechanism and inhibition », Curr. Med. Chem., vol. 9, no 12, , p. 1187–99 (PMID 12052171, DOI 10.2174/0929867023370013)
- J. De Angelis, J. Gastel, D. C. Klein et P. A. Cole, « Kinetic analysis of the catalytic mechanism of serotonin N-acetyltransferase (EC 2.3.1.87) », The Journal of Biological Chemistry, vol. 273, no 5, , p. 3045–3050 (PMID 9446620, DOI 10.1074/jbc.273.5.3045)
- PDB: 1KUX;
- « The structural basis of ordered substrate binding by serotonin N-acetyltransferase: enzyme complex at 1.8 A resolution with a bisubstrate analog », Cell, vol. 97, no 3, , p. 361–9 (PMID 10319816, DOI 10.1016/S0092-8674(00)80745-X, S2CID 18272015)
- « Melatonin biosynthesis: the structure of serotonin N-acetyltransferase at 2.5 A resolution suggests a catalytic mechanism », Mol. Cell, vol. 3, no 1, , p. 23–32 (PMID 10024876, DOI 10.1016/S1097-2765(00)80171-9)
- « Investigation of the roles of catalytic residues in serotonin N-acetyltransferase », J. Biol. Chem., vol. 277, no 20, , p. 18118–26 (PMID 11884405, DOI 10.1074/jbc.M200595200).
- « Crystal structure of the 14-3-3zeta:serotonin N-acetyltransferase complex. a role for scaffolding in enzyme regulation », Cell, vol. 105, no 2, , p. 257–67 (PMID 11336675, DOI 10.1016/S0092-8674(01)00316-6, S2CID 9564413)
- Matthew W. Vetting, Luiz Pedro S. de Carvalho, Michael Yu et Subray S. Hegde, « Structure and functions of the GNAT superfamily of acetyltransferases », Archives of Biochemistry and Biophysics, vol. 433, no 1, , p. 212–226 (PMID 15581578, DOI 10.1016/j.abb.2004.09.003)
- Klein, Coon, Roseboom et Weller, « The melatonin rhythm-generating enzyme: Molecular regulation of serotonin N-acetyltransferase in the pineal gland », Recent Progress in Hormone Research, vol. 52, , p. 307–357; discussion 357–8 (PMID 9238858)
- « cAmp regulation of arylalkylamine N-acetyltransferase (AANAT, EC 2.3.1.87): a new cell line (1E7) provides evidence of intracellular AANAT activation », J. Biol. Chem., vol. 276, no 26, , p. 24097–107 (PMID 11313340, DOI 10.1074/jbc.M011298200)
- « Rhythmic serotonin N-acetyltransferase mRNA degradation is essential for the maintenance of its circadian oscillation », Mol. Cell. Biol., vol. 25, no 8, , p. 3232–46 (PMID 15798208, PMCID 1069600, DOI 10.1128/MCB.25.8.3232-3246.2005)
- « Analysis of serotonin N-acetyltransferase regulation in vitro and in live cells using protein semisynthesis », Biochemistry, vol. 47, no 39, , p. 10407–19 (PMID 18771288, PMCID 2682328, DOI 10.1021/bi801189d)
- « Protein kinase C regulates the activity and stability of serotonin N-acetyltransferase », J. Neurochem., vol. 90, no 2, , p. 442–54 (PMID 15228600, DOI 10.1111/j.1471-4159.2004.02495.x, S2CID 42638894)
- « An intramolecular disulfide bridge as a catalytic switch for serotonin N-acetyltransferase », J. Biol. Chem., vol. 277, no 46, , p. 44229–35 (PMID 12215431, DOI 10.1074/jbc.M203305200)
- Lawrence M. Szewczuk, S. Adrian Saldanha, Surajit Ganguly et Erin M. Bowers, « De novo discovery of serotonin N-acetyltransferase inhibitors », Journal of Medicinal Chemistry, vol. 50, no 22, , p. 5330–5338 (PMID 17924613, PMCID 2531295, DOI 10.1021/jm0706463)
- « New substrate analogues of human serotonin N-acetyltransferase produce in situ specific and potent inhibitors », Eur. J. Biochem., vol. 271, no 2, , p. 418–28 (PMID 14717709, DOI 10.1046/j.1432-1033.2003.03942.x)
- « Novel bisubstrate analog inhibitors of serotonin N-acetyltransferase: the importance of being neutral », Bioorg. Chem., vol. 31, no 5, , p. 398–411 (PMID 12941292, DOI 10.1016/S0045-2068(03)00081-6)
- « Receptor- and ligand-based study on novel 2,2'-bithienyl derivatives as non-peptidic AANAT inhibitors », J Chem Inf Model, vol. 50, no 3, , p. 446–60 (PMID 20196559, DOI 10.1021/ci9004805)
- « Structure-activity relationships for substrates and inhibitors of pineal 5-hydroxytryptamine-N-acetyltransferase: preliminary studies », Eur. J. Pharmacol., vol. 307, no 2, , p. 133–40 (PMID 8832214, DOI 10.1016/0014-2999(96)00228-2)
- « Substrate specificity and inhibition studies of human serotonin N-acetyltransferase », J. Biol. Chem., vol. 275, no 12, , p. 8794–805 (PMID 10722724, DOI 10.1074/jbc.275.12.8794)
- Page, « Enzyme inhibition », Comprehensive Medicinal Chemistry, vol. 2, , p. 61–87
- Khalil et Philip A. Cole, « A Potent Inhibitor of the Melatonin Rhythm Enzyme », J. Am. Chem. Soc., vol. 120, no 24, , p. 6195–6196 (DOI 10.1021/ja981365a)
- « Mechanism-based inhibition of the melatonin rhythm enzyme: pharmacologic exploitation of active site functional plasticity », Proceedings of the National Academy of Sciences of the United States of America, vol. 96, no 22, , p. 12418–12423 (PMID 10535937, PMCID 22936, DOI 10.1073/pnas.96.22.12418, Bibcode 1999PNAS...9612418K)
- « Mechanistic studies on the alkyltransferase activity of serotonin N-acetyltransferase », Chemistry & Biology, vol. 8, no 4, , p. 379–389 (PMID 11325593, DOI 10.1016/s1074-5521(01)00020-5)
- « N-bromoacetyltryptamine strongly and reversibly inhibits in vitro melatonin secretion from mammalian pinealocytes », Neuroendocrinology Letters, vol. 26, no 5, , p. 581–592 (PMID 16264397)
Liens externes
- (en) MeSH Serotonin+N-Acetyltransferase
- AANAT human gene location in the UCSC Genome Browser.
- AANAT human gene details in the UCSC Genome Browser.