Ribozyme en épingle à cheveux
Le ribozyme en épingle à cheveux (hairpin ribozyme en anglais) est un court segment d'ARN doté d'activité catalytique présent dans l'ARN satellite de virus de plantes, à l'instar du ribozyme en tête de marteau. Il a tout d'abord été identifié dans l'ARN satellite du virus des taches en anneaux du tabac (TRSV) ou il catalyse l'auto-clivage et la jonction intervenant dans la réplication circulaire de cet ARN satellite. Il est semblable au ribozyme en tête de marteau par le fait qu'il ne requiert pas la présence de cations métalliques pour agir.
Occurrence naturelle et fonction biologique
Le ribozyme en épingle à cheveux n'a été identifié que dans trois séquences naturelles :
- l'ARN satellite du virus des taches en anneaux du tabac (sTRSV)[2] - [3] ;
- l'ARN satellite du virus de la mosaïque jaune de la chicorée (sCYMV)[4] ;
- l'ARN satellite du virus de la mosaïque de l'arabette (sARMV)[5].
Ce ribozyme se présente sous la forme d'une séquence d'ARN permettant la réplication des molécules d'ARN satellite dans lesquelles elle se trouve. Il s'agit d'une réaction autocatalytique. Le clivage et la jonction sont réalisés par le ribozyme, ce qui conduit à un mélange interconvertible de molécules d'ARN satellite linéaires et circulaires. Ces réactions sont importantes pour traiter les grandes molécules d'ARN produites par réplication circulaire. À la fin du cycle de réplication, ces grands intermédiaires de réplication de l'ARN satellite sont traitées pour produire des molécules circulaires ou linéaires de longueur unitaire avant d'être intégrées aux virus et transportées dans d'autres cellules pour de nouveaux cycles de réplication circulaire[6].
Des versions artificielles plus petites du ribozyme en épingle à cheveux ont été développées afin de permettre des analyses expérimentales plus poussées de la molécule[7]. Un domaine catalytique minimal de 50 nucléotides et un domaine substrat de 14 nucléotides a ainsi pu être identifié[3], permettant le développement d'un ribozyme catalysant le clivage de plusieurs substrats. Cette approche a été importante afin de pouvoir appliquer les méthodes de la biochimie pour l'analyse enzymatique de ce ribozyme, l'étude des éléments structuraux fondamentaux du complexe ribozyme-substrat et le développement par ingénierie moléculaire de ribozymes destinés à des applications biomédicales, notamment pour prévenir la réplication de virus pathogènes et pour étudier le rôle de certains gènes.
Mécanisme réactionnel et structure
Le clivage de brins d'ARN par ce ribozyme met en œuvre les mêmes réactions que plusieurs autres ribozymes et ribonucléases, produisant des fragments d'ARN terminés par un phosphate cyclique 2',3' et un groupe hydroxyle 5'. La réaction de jonction semble être simplement la réciproque de la réaction de clivage, c'est-à-dire la formation d'une liaison covalente entre une extrémité phosphate cyclique 2',3' et un hydroxyle 5' terminal pour produire une liaison phosphodiester 3'-5' standard. Ces réactions sont catalysées par le ribozyme lui-même sans devoir recourir à des cations métalliques, comme c'est également le cas pour d'autres enzymes et d'autres ribozymes[8].
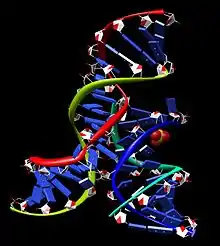
Le complexe formé par le ribozyme en épingle à cheveux minimal et le substrat possède une structure secondaire comprenant deux domaines, chacun constitué de deux courtes hélices appariées séparées par une boucle interne. Le domaine A (hélice 1, boucle A, hélice 2) contient le substrat et la première région de reconnaissance du substrat du ribozyme. Le domaine B (hélice 3, boucle B, hélice 4) est plus grand et contient les éléments déterminants du domaine catalytique du ribozyme. Ces deux domaines sont reliés par convalence via une liaison phosphodiester entre l'hélice 2 et l'hélice 3. Ces deux domaines doivent interagir l'un avec l'autre afin que la catalyse puisse avoir lieu[9].
À faible concentration ionique, le complexe formé par le substrat avec le ribozyme minimal se replie en formant une structure inactive ressemblant à une épingle à cheveux[10]. Pour que la catalyse puisse avoir lieu, les deux domaines doivent être parallèles l'un à l'autre dans une configuration qui ressemble à un trombone. Dans diverses publications, ce ribozyme a été appelé « en épingle à cheveux » (hairpin ribozyme en anglais) ou « en trombone » (paperclip ribozyme). Bien que ce dernier nom se soit révélé être le plus exact pour qualifier la structure fonctionnelle de ce ribozyme, c'est le premier terme qui s'est imposé dans la littérature. In vitro, l'interaction fonctionnelle entre les deux domaines est favorisée par l'addition de cations, dont la charge positive permet de compenser la répulsion électrostatique du squelette négativement chargé de l'ARN. In vivo, cette configuration fonctionnelle est assistée à la fois par des cations de magnésium Mg2+ et par la présence de deux domaines hélicoïdaux supplémentaires absents de la structure minimale mais servent à favoriser la configuration tridimensionnelle fonctionnelle. Ces deux domaines supplémentaires s'empilent sur les hélices 2 et 3 pour former ce qu'on appelle en anglais une four-way helical junction[11].
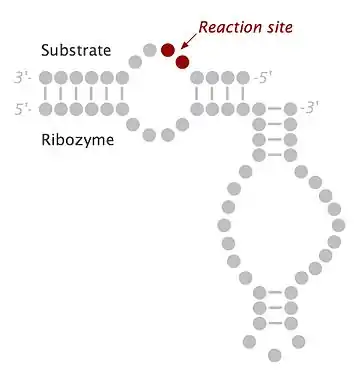 Structure secondaire d'un ribozyme en épingle à cheveux minimal (en bas) lié à son substrat (en haut). Les disques gris représentent les ribonucléotides et les tirets entre eux représentent les paires de bases canoniques (de type Watson-Crick).
Structure secondaire d'un ribozyme en épingle à cheveux minimal (en bas) lié à son substrat (en haut). Les disques gris représentent les ribonucléotides et les tirets entre eux représentent les paires de bases canoniques (de type Watson-Crick).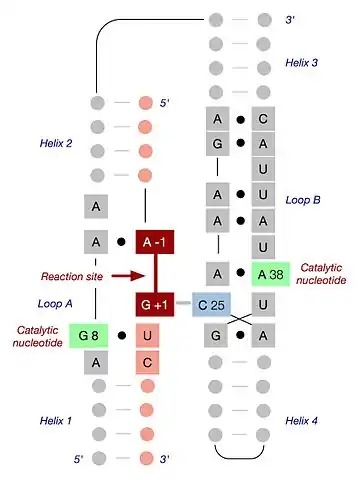 Structure tertiaire du ribozyme en épingle à cheveux montrant son repliement naturel. La séquence du ribozyme est représentée en gris et la séquence du substrat en rose. Le site de clivage et de jonction est représenté en bordeaux ; il est situé entre les nucléotides A-1 et G+1. Les séquences importantes des boucles A et B sont représentées, les points noirs indiquant les appariements non conventionnels (non Watson-Crick) entre nucléotides. Les deux nucléotides catalytiques sont représentés en vert, et le nucléotide critique C25, apparié au résidu G+1 au niveau du site actif, est représenté en bleu[12].
Structure tertiaire du ribozyme en épingle à cheveux montrant son repliement naturel. La séquence du ribozyme est représentée en gris et la séquence du substrat en rose. Le site de clivage et de jonction est représenté en bordeaux ; il est situé entre les nucléotides A-1 et G+1. Les séquences importantes des boucles A et B sont représentées, les points noirs indiquant les appariements non conventionnels (non Watson-Crick) entre nucléotides. Les deux nucléotides catalytiques sont représentés en vert, et le nucléotide critique C25, apparié au résidu G+1 au niveau du site actif, est représenté en bleu[12].
Notes et références
- (en) Jason Salter, Jolanta Krucinska, Shabnam Alam, Valerie Grum-Tokars et Joseph E. Wedekind, « Water in the Active Site of an All-RNA Hairpin Ribozyme and Effects of Gua8 Base Variants on the Geometry of Phosphoryl Transfer », Biochemistry, vol. 45, no 3, , p. 686-700 (PMID 16411744, PMCID 2546605, DOI 10.1021/bi051887k, lire en ligne)
- (en) Paul A. Feldstein, Jamal M. Buzayan et George Bruening, « Two sequences participating in the autolytic processing of satellite tobacco ringspot virus complementary RNA », Gene, vol. 82, no 1, , p. 53-61 (PMID 2583519, DOI 10.1016/0378-1119(89)90029-2, lire en ligne)
- (en) Arnold Hampel et Richard Tritz, « RNA catalytic properties of the minimum (-)sTRSV sequence », Biochemistry, vol. 28, no 12, , p. 4929-4933 (PMID 2765519, DOI 10.1021/bi00438a002, lire en ligne)
- (en) L. Rubino, M. E. Tousignant, G. Steger et J. M. Kaper, « Nucleotide sequence and structural analysis of two satellite RNAs associated with chicory yellow mottle virus », Journal of General Virology, vol. 71, no 9, , p. 1897-1903 (PMID 1698918, DOI 10.1099/0022-1317-71-9-1897, lire en ligne)
- (en) J. M. Kaper, M. E. Tousignant et G. Steger, « Nucleotide sequence predicts circularity and self-cleavage of 300-ribonucleotide satellite of arabis mosaic virus », Biochemical and Biophysical Research Communications, vol. 154, no 1, , p. 318-325 (PMID 3395334, DOI 10.1016/0006-291X(88)90687-0, lire en ligne)
- (en) Robert H. Symons, « Plant pathogenic RNAs and RNA catalysis », Nucleic Acid Research, vol. 25, no 14, , p. 2683-2689 (PMID 9207012, PMCID 146833, DOI 10.1093/nar/25.14.2683, lire en ligne)
- (en) Paul A. Feldstein et George Bruening, « Catalytically active geometry in the reversible circularization of ‘mini-monomer’ RNAs derived from the complementary strand of tobacco ringspot virus satellite RNA », Nucleic Acid Research, vol. 21, no 8, , p. 1991-1998 (PMID 7684131, PMCID 309442, DOI 10.1093/nar/21.8.1991, lire en ligne)
- (en) James B. Murray, Attila A. Seyhan, Nils G. Walter, John M. Burke et William G. Scott, « The hammerhead, hairpin and VS ribozymes are catalytically proficient in monovalent cations alone », Cell, vol. 5, no 10, , p. 587-595 (PMID 9818150, DOI 10.1016/S1074-5521(98)90116-8, lire en ligne)
- (en) Samuel E. Butcher, Joyce E. Heckman et John M. Burke, « Reconstitution of Hairpin Ribozyme Activity following Separation of Functional Domains », Journal of Biological Chemistry, vol. 270, no 50, , p. 29648-29651 (PMID 8530348, DOI 10.1074/jbc.270.50.29648, lire en ligne)
- (en) José A. Esteban, Nils G. Walter, Gerd Kotzorek et Joyce E. Heckman et John M. Burke, « Structural basis for heterogeneous kinetics: Reengineering the hairpin ribozyme », Proceedings of the National Academy of Sciences of the United States of America, vol. 95, no 11, , p. 6091-6096 (PMID 9600922, PMCID 27590, DOI 10.1073/pnas.95.11.6091, Bibcode 1998PNAS...95.6091E, lire en ligne)
- (en) T. J. Wilson, M. Nahas, T. Ha et D. M. J. Lilley, « Folding and catalysis of the hairpin ribozyme », Biochemical Society transactions, vol. 33, no part 3, , p. 461-465 (PMID 15916541, DOI 10.1042/BST0330461, lire en ligne)
- (en) Shabnam Alam, Valerie Grum-Tokars, Jolanta Krucinska, Melisa L. Kundracik et Joseph E. Wedekind, « Conformational Heterogeneity at Position U37 of an All-RNA Hairpin Ribozyme with Implications for Metal Binding and the Catalytic Structure of the S-Turn », Biochemistry, vol. 44, no 44, , p. 14396-14408 (PMID 16262240, DOI 10.1021/bi051550i, lire en ligne)