NADH déshydrogénase
La NADH déshydrogénase, ou complexe I de la chaîne respiratoire, est une oxydoréductase membranaire qui catalyse la réaction :
- NADH + ubiquinone + 5 H+matriciel NAD+ + ubiquinol + 4 H+intermembranaire.
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
Cette enzyme est présente chez un très grand nombre d'êtres vivants, allant des procaryotes jusqu'aux humains. Elle constitue le premier point d'entrée des électrons à haut potentiel de transfert dans la respiration cellulaire et la phosphorylation oxydative dans les mitochondries[2] et assure le transfert des électrons du NADH vers la coenzyme Q10 dissoute dans la bicouche lipidique. Chez les eucaryotes, elle est située dans la membrane mitochondriale interne. Elle jouerait également un rôle dans le déclenchement de l'apoptose[3] ; une corrélation a pu être établie entre l'activité des mitochondries et l'apoptose lors du développement somatique de l'embryon[4].
Structure et fonctionnement
Le complexe I est l'enzyme la plus grande et la plus compliquée de la chaîne respiratoire[5]. Chez les mammifères, elle est constituée de 44 chaînes polypeptidiques, dont sept sont encodées par le génome mitochondrial[6] - [7]. Elle contient notamment un groupe prosthétique FMN et huit clusters fer-soufre dont sept sont alignés pour permettre la circulation des électrons depuis le NADH vers la coenzyme Q10. Le transfert de ces électrons d'un couple rédox dont le potentiel standard est −0,32 V vers un couple rédox dont le potentiel standard est +0,06 V libère suffisamment d'énergie pour actionner une pompe à protons expulsant quatre protons H+ de la matrice mitochondriale vers l'espace intermembranaire à travers la membrane interne de la mitochondrie.
La structure générale adopte une forme en L avec un long domaine membranaire constitué d'une soixantaine d'hélices et un domaine hydrophile périphérique où se trouvent tous les centres rédox et le site de liaison au NADH. La structure du complexe I des eucaryotes n'est pas encore bien caractérisée dans son ensemble mais celle du domaine hydrophile de la bactérie Thermus thermophilus (en) a pu être établie (PDB 2FUG[1]) ainsi que celle des domaines membranaires d'E. coli (PDB 3RKO) et de T. thermophilus (PDB 4HE8). La structure complète d'une NADH déshydrogénase de T. thermophilus a été publiée pour la première fois en (PDB 4HEA[8]).
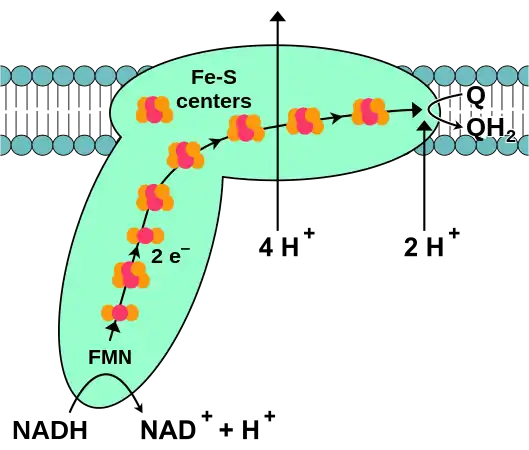 (en) Schéma de fonctionnement de la NADH déshydrogénase montrant sa forme générale constituée d'un domaine hydrophile (en bas) baignant dans la matrice mitochondriale et jalonné par sept clusters fer-soufre alignés conduisant les électrons issus du NADH matriciel via un groupe prosthétique FMN jusqu'au domaine hydrophobe (en haut) inclus dans la membrane mitochondriale interne et dans laquelle la coenzyme Q10 est réduite en ubiquinol Q10H2. Quatre protons H+ sont expulsés vers l'espace intermembranaire mitochondrial au cours de cette réaction, ce qui contribue à générer un gradient de concentration de protons à travers la membrane mitochondriale interne.
(en) Schéma de fonctionnement de la NADH déshydrogénase montrant sa forme générale constituée d'un domaine hydrophile (en bas) baignant dans la matrice mitochondriale et jalonné par sept clusters fer-soufre alignés conduisant les électrons issus du NADH matriciel via un groupe prosthétique FMN jusqu'au domaine hydrophobe (en haut) inclus dans la membrane mitochondriale interne et dans laquelle la coenzyme Q10 est réduite en ubiquinol Q10H2. Quatre protons H+ sont expulsés vers l'espace intermembranaire mitochondrial au cours de cette réaction, ce qui contribue à générer un gradient de concentration de protons à travers la membrane mitochondriale interne.
Inhibiteurs
La roténone est l'inhibiteur le plus connu du complexe I. Elle est couramment utilisé comme pesticide et comme insecticide. Avec les isoflavonoïdes (en), les roténoïdes sont présents chez plusieurs genres de plantes tropicales telles que Antonia (Loganiaceae), Derris et Lonchocarpus (Faboideae, Fabaceae). Tout comme la piéricidine A (en), la roténone se lie au site de liaison à l'ubiquinone du complexe I.
L'adénosine diphosphate ribose (ADPR) est également un inhibiteur de la NADH déshydrogénase en se liant au site de liaison nucléotidique[9], ce qui bloque la liaison du NADH.
La famille des acétogénines forme les inhibiteurs les plus puissants du complexe I. Ces molécules se lient à la sous-unité ND2, ce qui tend à montrer que celle-ci serait essentielle à la liaison de l'ubiquinone[2]. L'une de ces molécules, la rolliniastatine-2, est le premier inhibiteur du complexe I qui ne se lie pas au même site que la roténone[10].
La metformine, un antidiabétique, est également un inhibiteur partiel du complexe I, ce qui semble jouer un rôle essentiel dans son mode d'action[11].
Notes et références
- (en) Leonid A. Sazanov et Philip Hinchliffe, « Structure of the Hydrophilic Domain of Respiratory Complex I from Thermus thermophilus », Science, vol. 311, no 5766, , p. 1430-1436 (PMID 16469879, DOI 10.1126/science.1123809, Bibcode 2006Sci...311.1430S, lire en ligne)
- (en) Eiko Nakamaru-Ogiso, Hongna Han, Akemi Matsuno-Yagi, Ehud Keinan, Subhash C. Sinha, Takao Yagi et Tomoko Ohnishi, « Structure of the Hydrophilic Domain of Respiratory Complex I from Thermus thermophilus », FEBS Letters, vol. 584, no 5, , p. 883-888 (PMID 20074573, PMCID 2836797, DOI 10.1016/j.febslet.2010.01.004, lire en ligne)
- (en) Maria Chomova et Peter Racay, « Mitochondrial complex I in the network of known and unknown facts », General Physiology and Biophysics, vol. 29, no 1, , p. 3-11 (PMID 20371875, DOI 10.4149/gpb_2010_01_3, lire en ligne)
- (en) Elisa Petrussa, Alberto Bertolini, Valentino Casolo, Jana Krajňáková, Francesco Macrì et Angelo Vianello, « Mitochondrial bioenergetics linked to the manifestation of programmed cell death during somatic embryogenesis of Abies alba », Planta, vol. 231, no 1, , p. 93-107 (PMID 19834734, DOI 10.1007/s00425-009-1028-x, lire en ligne)
- (en) Ulrich Brandt, « Energy Converting NADH: Quinone Oxidoreductase (Complex I) », Annual Review of Biochemistry, vol. 75, , p. 69-92 (PMID 16756485, DOI 10.1146/annurev.biochem.75.103004.142539, lire en ligne)
- (en) Joe Carroll, Ian M. Fearnley, J. Mark Skehel1, Richard J. Shannon, Judy Hirst et John E. Walker2, « Bovine Complex I Is a Complex of 45 Different Subunits », Journal of Biological Chemistry, vol. 281, no 43, , p. 32724-32727 (PMID 16950771, DOI 10.1074/jbc.M607135200, lire en ligne)
- (en) Eduardo Balsa, Ricardo Marco, Ester Perales-Clemente, Radek Szklarczyk, Enrique Calvo, Manuel O. Landázuri et José Antonio Enríquez, « NDUFA4 Is a Subunit of Complex IV of the Mammalian Electron Transport Chain », Cell Metabolism, vol. 16, no 3, , p. 378-386 (PMID 22902835, DOI 10.1016/j.cmet.2012.07.015, lire en ligne)
- (en) Rozbeh Baradaran, John M. Berrisford, Gurdeep S. Minhas et Leonid A. Sazanov, « Crystal structure of the entire respiratory complex I », Nature, vol. 494, no 7438, , p. 443-448 (PMID 23417064, PMCID 3672946, DOI 10.1038/nature11871, Bibcode 2013Natur.494..443B, lire en ligne)
- (en) Tatyana V. Zharova et Andrei D. Vinogradov, « A competitive inhibition of the mitochondrial NADH-ubiquinone oxidoreductase (Complex I) by ADP-ribose », Biochimica et Biophysica Acta (BBA) - Bioenergetics, vol. 1320, no 3, , p. 256-264 (PMID 9230920, DOI 10.1016/S0005-2728(97)00029-7, lire en ligne)
- (en) M. Degli Esposti, A. Ghelli, M. Ratta, D. Cortes et E. Estornell, « Natural substances (acetogenins) from the family Annonaceae are powerful inhibitors of mitochondrial NADH dehydrogenase (Complex I) », Biochemical Journal, vol. 301, no 1, , p. 161-167 (PMID 8037664, PMCID 1137156, DOI 10.1042/bj3010161, lire en ligne)
- (en) Benoit Viollet, Bruno Guigas, Nieves Sanz Garcia, Jocelyne Leclerc, Marc Foretz et Fabrizio Andreelli, « Cellular and molecular mechanisms of metformin: an overview », Clinical Science, vol. 122, no 6, , p. 253-270 (PMID 22117616, PMCID 3398862, DOI 10.1042/CS20110386, lire en ligne)
