Multiomique
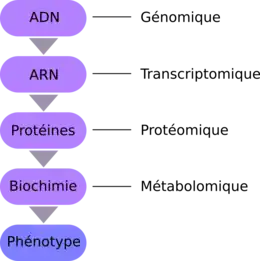
La multiomique est une discipline de la biotechnologie alliant les dernières avancées et analyses des champs de recherche de la génomique, de la métabolomique, de la transcriptomique et de la protéomique. Elle est parfois aussi appelée: omique intégrative, panomique ou pan-omique[1].
| Partie de |
|---|
Le terme peut être décrit comme une approche d'analyse biologique dans laquelle les ensembles de données sont des « omes » multiples, tels que le génome, le protéome, le transcriptome, l'épigénome, le métabolome et le microbiome (c'est-à-dire un métagénome et/ou un métatranscriptome, selon la manière dont il est séquencé) en d'autres termes, l'utilisation de multiples technologies omiques pour étudier la vie de manière croisée.
En combinant ces omiques, on peut analyser des mégadonnées biologiques pour trouver de nouvelles associations entre des entités biologiques, ou encore repérer des biomarqueurs pertinents et construire des marqueurs physiologiques. Ce faisant, la multiomique intègre diverses données omiques pour trouver une relation ou une association « géno-phéno-envirotype » cohérente.
Historique
La publication de la structure de l'ADN par Francis Crick et James Watson le 25 avril 1953 marqua un tournant dans l'étude de la génomique[2]. Elle peut être vue comme le point de départ historique du développement d'études transversales et croisées dans le futur domaine de la biotechnologie. Ces avancées amèneront la création de la multiomique comme discipline, à la fin du XXe siècle.
La lecture et l'étude du transcriptome, à partir des années 1980, permet progressivement d’identifier et de quantifier les produits de l’expression des gènes d’une cellule ou d’un tissu dans un environnement donné[2] ; c'est le début d'une vision « méta » des effets de l'expression génétique dans l'organisme humain (ex: la phénotypologie).
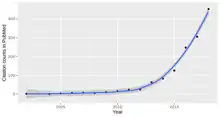
Une évolution nette du nombre de publications incluant la multiomique — dans leur méthodologie ou leur sujet — apparaît à la fin des années 2000 (cf. graphique Pubmed ci-contre) ; ce nombre passant de zéro en 2000 à plus de 400 par an en 2018 ; ce chiffre croissait alors de manière exponentielle.
Multiomique unicellulaire
Une branche de la multiomique s'intéresse à l'analyse des données « multiniveau » d'une cellule unique, appelée « multiomique unicellulaire[3] ». Cette approche nous donne une résolution sans précédent dans l'examen des transitions au niveau unicellulaire, applicable notamment (mais pas exclusivement) dans tous les domaines médicaux[4]. Un avantage par rapport à l'analyse en masse est d'atténuer les facteurs de confusion, provenant de la variation d'une cellule à l'autre, ce qui permet de découvrir des architectures tissulaires hétérogènes.
Ces méthodes parallèles d'analyse génomique et transcriptomique à l'échelle unicellulaire peuvent être basées sur « l'amplification simultanée »[5] ou la séparation physique de l'ARN et de l'ADN génomique. Elles permettent d'obtenir des informations qui ne peuvent être obtenues uniquement à partir de l'analyse transcriptomique, car les données ARN ne contiennent pas de régions génomiques non codantes ni d'informations concernant la variation du nombre de copies, par exemple. Une extension de cette méthodologie est l'intégration des transcriptomes unicellulaires aux méthylomes unicellulaires, en combinant le séquençage au bisulfite à l'ARN-Seq unicellulaire[6]. Il existe également d'autres techniques pour interroger l'épigénome, comme l'« ATAC-Seq unicellulaire »[7] et le « Hi-C unicellulaire ».
Un défi différent, mais connexe, est l'interaction des données protéomiques et transcriptomiques[8]. Cette approche consiste à séparer physiquement les lysats de cellules uniques en deux, en traitant la moitié pour l'ARN et l'autre moitié pour les protéines. La teneur en protéines des lysats peut être mesurée par des « essais d'extension de proximité » (en anglais: « PEA »)[9], par exemple, qui utilisent des anticorps codés par l'ADN. Une autre approche utilise une combinaison de sondes ARN à métaux lourds et d'anticorps protéiques pour adapter la cytométrie de masse à une analyse multiomique.
Multiomique et apprentissage automatique
Parallèlement aux progrès de la « biologie à haut débit » – utilisant notamment les mégadonnées, les applications de l'apprentissage automatique aux besoins de l'analyse de données biomédicales sont toujours en plein essor. L'intégration de l'analyse de données multiomiques et de l'apprentissage automatique conduisit à la découverte de nouveaux biomarqueurs. Par exemple, l'une des méthodes du projet mixOmics met en œuvre un procédé basé sur la régression des moindres carrés partiels pour la sélection de caractéristiques (on appelle alors celles-ci des « biomarqueurs putatifs »). Un cadre statistique unifié pour l'intégration de données hétérogènes intitulé "Regularized Generalized Canonical Correlation Analysis" (RGCCA[10] - [11] - [12] - [13]) permet de réaliser ces analyses intégratives pour l'identification de biomarqueurs putatifs. RGCCA est disponible au travers du package R RGCCA.
Dans la Santé
La multiomique promet actuellement de combler des lacunes dans la compréhension de la santé et des maladies humaines. De nombreux chercheurs travaillent sur des moyens de générer et d'analyser des données multiomiques liées aux maladies[14].
Les applications vont de la compréhension des interactions hôte-pathogène et des maladies infectieuses[15] à une meilleure compréhension des maladies chroniques et complexes non transmissibles et à l'amélioration de la médecine personnalisée[16].
Projet microbiote humain
Le Projet microbiote humain utilise largement les techniques de la multiomique décrits ci-dessus. La deuxième phase du projet (170 000 000$) est axée sur l'intégration des données des patients à différents ensembles de données omiques, en tenant compte de la génétique de l'hôte, des informations cliniques et de la composition du microbiome[17].
La phase 1 était axée sur la caractérisation des communautés cellulaires dans différents sites corporels. La phase 2 se concentre sur l'intégration des données multiomiques de l'hôte et du microbiome aux maladies humaines. Plus précisément, le projet utilise la multiomique pour améliorer la compréhension de l'interaction entre les microbiomes intestinaux et nasaux et le diabète de type 2[18]; les microbiomes intestinaux et les maladies inflammatoires de l'intestin; et les microbiomes vaginaux et la naissance prématurée[19].
Immunologie
La complexité des interactions dans le système immunitaire humain entraine la production d'une multitude de données omiques multi-échelles liées à l'immunologie[20]. L'analyse de données multi-omiques est déjà utilisée pour recueillir de nouvelles informations sur une réponse immunitaire aux maladies infectieuses (comme le chikungunya pédiatrique) ainsi qu'à des maladies auto-immunes non transmissibles. Par exemple, la multiomique fut centrale dans la découverte de l'association entre les changements des métabolites du plasma et le transcriptome du système immunitaire et la réponse à la vaccination contre le zona.
Liste de logiciels pour l'analyse multi-omique

Le projet Bioconductor regroupe une variété de paquets R (un ensemble exprimable de lignes de code) visant à intégrer des données omiques :
- (en) omicade4, pour l'analyse de co-inertie multiple d'ensembles de données multi omiques[21]
- (en) MultiAssayExperiment, qui offre une interface Bioconductor pour les échantillons superposés[22].
- (en) IMAS, un paquetage axé sur l'utilisation de données multi omiques pour l'évaluation de l'épissage alternatif[23]
- (en) bioCancer, un paquetage pour la visualisation de données multiomiques sur le cancer[24]
- (en) mixOmics, une suite de méthodes multivariées pour l'intégration de données[25].
- (en) MultiDataSet, un paquetage pour l'encapsulation de plusieurs ensembles de données[26].
Le package RGCCA implémente un cadre statistique flexible pour l'intégration de données hétérogènes. Le package RGCCA est disponible sur le CRAN.
D'autres outils existent:
- (en) PaintOmics, une ressource Web pour la visualisation d'ensembles de données multi-omiques[27].
- (en) SIGMA, un programme Java axé sur l'analyse intégrée des ensembles de données sur le cancer[28].
- (en) iOmicsPASS, un outil en C++ pour la prédiction de phénotypes basée sur les données multiomiques[29].
- (en) Grimon, une interface graphique « R » pour la visualisation de données multiomiques[30].
- (en) Omics Pipe, un cadre en Python pour l'automatisation reproductible de l'analyse des données multiomiques[31].
Bases de données multiomiques
L'une des principales limitations des études omiques classiques est l'analyse d'un seul niveau de complexité biologique. Par exemple, les études transcriptomiques peuvent fournir des informations au niveau du transcrit, mais de nombreuses entités différentes contribuent à l'état biologique de l'échantillon (variantes génomiques, modifications post-traductionnelles, produits métaboliques, organismes en interaction, etc.). Avec l'avènement de la « biologie à haut débit », il devient de plus en plus abordable d'effectuer des mesures multiples, ce qui permet d'établir des corrélations et des déductions interdisciplinaires ou « transdomaines » (par exemple, les niveaux d'ARN et de protéines). Ces corrélations aident à la construction de réseaux biologiques plus complets, comblant ainsi les lacunes de nos connaissances.
L'intégration des données n'est cependant pas une tâche facile. Pour faciliter le processus, des groupes ont créé des bases de données pour explorer systématiquement les données multiomiques :
- (en) The Pancreatic Expression Database, intégrant des données relatives au tissu pancréatique[32];
- (en) LinkedOmics reliant les données des ensembles de données sur le cancer (notamment issues du TCGA)[33];
- (en) OASIS, une ressource en ligne pour les études générales sur le cancer[34];
- (en) BCIP, une plateforme pour les études sur le cancer du sein;
- (en) C/VDdb, reliant les données de plusieurs études sur les maladies cardiovasculaires[35];
- (en) ZikaVR, une ressource multiomique pour les données sur le virus Zika[36];
- (en) Ecomics, une base de données multi-omique normalisée pour les données sur Escherichia coli[37];
- (en) GourdBase, intégrant les données d'études sur la courge[38];
- (en) MODEM, une base de données pour les données multi-niveaux sur le maïs[39];
- (en) SoyKB, une base de données pour des données multiniveaux sur le soja[40];
- (en) ProteomicsDB, une ressource multi-omique (et multi-organismes) pour la recherche en sciences de la vie. Le portail contient aussi une boîte à outils d'analyse de schémas génétiques de co-expressions.
Recherche
De nombreux organes de recherche (publiques et privés) à travers le monde utilisent la multiomique de façon croissante depuis les années 1990:
L'Institut Pasteur (France) mène des recherches multiomiques depuis les années 2000; En 2018, il inaugure deux bâtiments consacrés intégralement à la recherche des omiques (« omics »), spécialement en biomique et bioinformatique[41], encourageant ainsi au niveau mondial la pratique de l'analyse croisée de mégadonnées telle que pratiquée en multiomique.
Des centres et instituts américains, tels que l'université de Californie[42], l'Institut Bonanza Creek pour la recherche en biologie arctique[43] (Université de l'Alaska), ou encore le département des sciences informatiques l'université Stony Brook[44] de New York.
L'Institut Francis Crick (Royaume-Uni) mène depuis les années 2010 des études multiomiques[45].
Depuis les années 2010 également, la Chine a lancé un plan gouvernemental pour l'utilisation de la multiomique dans certains laboratoires de recherche[46], notamment à l'Académie chinoise des sciences agricoles[47].
Jusqu'en 2018, le site Omicx (basé à Rouen, France) répertoriait une centaine de logiciels liés à l'analyse de données multiomiques, ainsi que la plupart des bases de données sur le sujet[48]. Le site est à présent à l'arrêt.
En 2021, l'Institut Max Planck pour la génétique moléculaire-MPIMG (Allemagne) crée un algorithme capable d'identifier des expressions génétiques cancéreuses, même en l'absence de modification de l'ADN en question[49]. Cette avancée permet d'identifier 165 gènes cancéreux supplémentaires.
Exemples d'application
Grâce à la protéomique chez des bactéries, la multiomique peut par exemple faire avancer drastiquement la recherche de nouveaux antibiotiques. En 2020, une équipe de l'université de la Ruhr (Allemagne) a profilé le protéome de Bacillus subtilis et établi une bibliothèque des modifications de ses 486 protéines face à 91 médicaments antibiotiques sur le marché. Leur objectif était de définir le mode d'action d'une molécule candidate au titre d'antibiotique, en le comparant à ceux de médicaments connus. L'expérience a validé deux médicaments candidats prometteurs, appelés oxadiazioles, au mode d'action complètement novateur[50]. Cette méthode peut être étendue à d'autres molécules, chez d'autres espèces bactériennes, afin de faire émerger de nouveaux traitements en médecine conventionnelle.
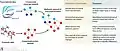 « Réseaux de coexpression » à partir de plusieurs types de données omiques, servant à mieux appréhender les manières dont ces différents constituants génétiques interagissent.
« Réseaux de coexpression » à partir de plusieurs types de données omiques, servant à mieux appréhender les manières dont ces différents constituants génétiques interagissent.
Sources
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Multiomics » (voir la liste des auteurs).
Notes et références
- (en) Bersanelli, Matteo; Mosca, Ettore; Remondini, Daniel; Giampieri, Enrico; Sala, Claudia; Castellani, Gastone; Milanesi, Luciano, « Methods for the integration of multi-omics data: mathematical aspects », BMC Bioinformatics, vol. 17, no Suppl 2, , S15. (PMID 26821531, PMCID PMC4959355, DOI 10.1186/s12859-015-0857-9)
- (fr) https://www.jeanmann.com/a3999541.html
- (en) Han, Jing-Dong Jackie (2018). Faculty of 1000 evaluation for Single-Cell Multiomics: Multiple Measurements from Single Cells. doi:10.3410/f.727213649.793550351
- (en) Youjin Hu, Qin An, Katherine Sheu, Brandon Trejo, Shuxin Fan, Ying Guo, « Single Cell Multi-Omics Technology: Methodology and Application », Front Cell Dev Biol, no 6, , p. 28. (PMID 29732369, PMCID PMC5919954, DOI 10.3389/fcell.2018.00028)
- (en) Kester, Lennart Spanjaard, Bastiaan Bienko, Magda van Oudenaarden, Alexander Dey, Siddharth S (2015). Integrated genome and transcriptome sequencing of the same cell. Nature Biotechnology. 33 (3): 285–289. doi:10.1038/nbt.3129. OCLC 931063996. PMC 4374170. .
- (en) Angermueller, Christof; Clark, Stephen J; Lee, Heather J; Macaulay, Iain C; Teng, Mabel J; Hu, Tim Xiaoming; Krueger, Felix; Smallwood, Sébastien A; Ponting, Chris P (2016-01-11).Parallel single-cell sequencing links transcriptional and epigenetic heterogeneity. Nature Methods. 13 (3): 229–232. doi:10.1038/nmeth.3728. ISSN 1548-7091. PMC 4770512.
- (en) Greenleaf, William J.; Chang, Howard Y.; Snyder, Michael P.; Michael L. Gonzales; Ruff, Dave; Litzenburger, Ulrike M.; Wu, Beijing; Buenrostro, Jason D. (2015). Single-cell chromatin accessibility reveals principles of regulatory variation. Nature. 523 (7561): 486–490.
- (en) https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4713867/
- (en) Assarsson, Erika; Lundberg, Martin; Holmquist, Göran; Björkesten, Johan; Bucht Thorsen, Stine; Ekman, Daniel; Eriksson, Anna; Rennel Dickens, Emma; Ohlsson, Sandra (2014). Homogenous 96-Plex PEA Immunoassay Exhibiting High Sensitivity, Specificity, and Excellent Scalability. PLOS ONE.
- Arthur Tenenhaus et Michel Tenenhaus, « Regularized Generalized Canonical Correlation Analysis », Psychometrika, vol. 76, no 2, , p. 257–284 (ISSN 0033-3123 et 1860-0980, DOI 10.1007/s11336-011-9206-8, lire en ligne, consulté le )
- A. Tenenhaus, C. Philippe, V. Guillemot et K.-A. Le Cao, « Variable selection for generalized canonical correlation analysis », Biostatistics, vol. 15, no 3, , p. 569–583 (ISSN 1465-4644 et 1468-4357, DOI 10.1093/biostatistics/kxu001, lire en ligne, consulté le )
- Arthur Tenenhaus, Cathy Philippe et Vincent Frouin, « Kernel Generalized Canonical Correlation Analysis », Computational Statistics & Data Analysis, vol. 90, , p. 114–131 (ISSN 0167-9473, DOI 10.1016/j.csda.2015.04.004, lire en ligne, consulté le )
- Michel Tenenhaus, Arthur Tenenhaus et Patrick J. F. Groenen, « Regularized Generalized Canonical Correlation Analysis: A Framework for Sequential Multiblock Component Methods », Psychometrika, vol. 82, no 3, , p. 737–777 (ISSN 0033-3123 et 1860-0980, DOI 10.1007/s11336-017-9573-x, lire en ligne, consulté le )
- (en) Hasin, Yehudit; Seldin, Marcus; Lusis, Aldons (2017). Multi-omics approaches to disease. Genome Biology.
- (en) Aderem, Alan; Adkins, Joshua N.; Ansong, Charles; Galagan, James; Kaiser, Shari; Korth, Marcus J.; Law, G. Lynn; McDermott, Jason G.; Proll, Sean C. (2011). A Systems Biology Approach to Infectious Disease Research: Innovating the Pathogen-Host Research Paradigm. mBio.
- (en) He, Feng Q.; Ollert, Markus; Balling, Rudi; Bode, Sebastian F. N.; Delhalle, Sylvie (2018). A roadmap towards personalized immunology. NPJ Systems Biology and Applications.
- (en) Proctor, Lita M.; Creasy, Heather H.; Fettweis, Jennifer M.; Lloyd-Price, Jason; Mahurkar, Anup; Zhou, Wenyu; Buck, Gregory A.; Snyder, Michael P.; Strauss, Jerome F. (2019). The Integrative Human Microbiome Project. Nature.
- (en)Snyder, Michael; Weinstock, George M.; Sodergren, Erica; McLaughlin, Tracey; Tse, David; Rost, Hannes; Piening, Brian; Kukurba, Kim; Rose, Sophia Miryam Schüssler-Fiorenza (May 2019). Longitudinal multi-omics of host–microbe dynamics in prediabetes. Nature.
- (en)Buck, Gregory A.; Strauss, Jerome F.; Jefferson, Kimberly K.; Hendricks-Muñoz, Karen D.; Wijesooriya, N. Romesh; Rubens, Craig E.; Gravett, Michael G.; Sexton, Amber L.; Chaffin, Donald O. (June 2019). The vaginal microbiome and preterm birth. Nature Medicine.
- (en)Kidd, Brian A; Peters, Lauren A; Schadt, Eric E; Dudley, Joel T (2014). Unifying immunology with informatics and multiscale biology. Nature Immunology.
- (en) https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4053266/
- (en) Ramos, Marcel; Schiffer, Lucas; Re, Angela; Azhar, Rimsha; Basunia, Azfar; Rodriguez Cabrera, Carmen; Chan, Tiffany; Chapman, Philip; Davis, Sean (2017). Software For The Integration Of Multi-Omics Experiments In Bioconductor. Sur biorxiv.org.
- (en) https://bioconductor.org/packages/release/bioc/html/IMAS.html
- (en) Karim Mezhoud (2017), bioCancer, Bioconductor
- (en) Rohart, Florian; Gautier, Benoît; Singh, Amrit; Lê Cao, Kim-Anh (2017). mixOmics: an R package for 'omics feature selection and multiple data integration. PLOS Computational Biology
- (en) Hernandez-Ferrer, Carles; Ruiz-Arenas, Carlos; Beltran-Gomila, Alba; González, Juan R. (2017). MultiDataSet: an R package for encapsulating multiple data sets with application to omic data integration. BMC Bioinformatics.
- (en) http://www.paintomics.org/
- (en) Chari, Raj; Coe, Bradley P.; Wedseltoft, Craig; Benetti, Marie; Wilson, Ian M.; Vucic, Emily A.; MacAulay, Calum; Ng, Raymond T.; Lam, Wan L. (2008). SIGMA2: A system for the integrative genomic multi-dimensional analysis of cancer genomes, epigenomes, and transcriptomes. BMC Bioinformatics.
- (en) Choi, Hyungwon; Ewing, Rob; Choi, Kwok Pui; Fermin, Damian; Koh, Hiromi W. L. (2018). iOmicsPASS: a novel method for integration of multi-omics data over biological networks and discovery of predictive subnetworks. bioRxiv: 374520.
- (en) Kanai, Masahiro; Maeda, Yuichi; Okada, Yukinori (2018). Grimon: graphical interface to visualize multi-omics networks. Bioinformatics.
- (en) Su, Andrew I.; Loguercio, Salvatore; Carland, Tristan M.; Ducom, Jean-Christophe; Gioia, Louis; Meißner, Tobias; Fisch, Kathleen M. (2015). Omics Pipe: a community-based framework for reproducible multi-omics data analysis. Bioinformatics.
- (en) http://pancreasexpression.org:9000/
- (en) http://linkedomics.org/login.php
- (en) https://www.nature.com/articles/nmeth.3692
- (en) Husi, Holger; Patel, Alisha; Fernandes, Marco (2018). C/VDdb: A multi-omics expression profiling database for a knowledge-driven approach in cardiovascular disease (CVD). PLOS ONE.
- (en) Gupta, Amit Kumar; Kaur, Karambir; Rajput, Akanksha; Dhanda, Sandeep Kumar; Sehgal, Manika; Khan, Md. Shoaib; Monga, Isha; Dar, Showkat Ahmad; Singh, Sandeep (2016). ZikaVR: An Integrated Zika Virus Resource for Genomics, Proteomics, Phylogenetic and Therapeutic Analysis. Scientific Reports.
- (en) Tagkopoulos, Ilias; Violeta Zorraquino; Rai, Navneet; Kim, Minseung (2016). Multi-omics integration accurately predicts cellular state in unexplored conditions for Escherichia coli. Nature Communications.
- (en) Li, Guojing; Lu, Zhongfu; Lin, Jiandong; Hu, Yaowen; Yunping Huang; Wang, Baogen; Wu, Xinyi; Wu, Xiaohua; Xu, Pei (2018). GourdBase: a genome-centered multi-omics database for the bottle gourd ( Lagenaria siceraria ), an economically important cucurbit crop. Scientific Reports.
- (en) https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4976297/
- (en) https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3965117/
- (fr) https://www.pasteur.fr/fr/journal-recherche/actualites/omics-biologie-heure-du-numerique-institut-pasteur
- (en) http://bisep.karnataka.gov.in/images/pdf/news/13_05.05.2017.pdf
- (en) http://www.lter.uaf.edu/publications/publications
- (en) https://pdfs.semanticscholar.org/46ce/5fb1e83568ee2ccbec22c62fd4d2e989b7be.pdf
- (en) https://www.crick.ac.uk/research/publications/biodip-a-multiomics-study-to-identify-biomarkers-of-disease-and-to-understand-further-the-pathogenesis-of-psoriasis-and-atopic-dermatitis
- (en) https://liebertpub.com/doi/10.1089/omi.2019.0199
- (en) https://gut.bmj.com/content/gutjnl/early/2021/02/14/gutjnl-2020-320951.full.pdf?with-ds=yes
- (fr) https://web.archive.org/web/20180821082925/https://omicx.com/
- (en) https://www.sciencedaily.com/releases/2021/04/210412142730.htm
- (fr) https://www.science-et-vie.com/corps-et-sante/une-formidable-acceleration-de-la-biomedecine-61630
Voir aussi
Bibliographie
- Pierre Douzou, Les Biotechnologies, PUF, 1984.
- A. Henco International Biotechnology Economics and Policy: Science, Business Planning and Entrepreneurship; Impact on Agricultural Markets and Industry; Opportunities in the Healthcare Sector. 2007. (ISBN 978-0-7552-0293-5).
- Axel Kahn et Dominique Lecourt, Bioéthique et liberté, PUF/Quadrige essai, Paris, 2004).
- Dominique Lecourt (dir.), Dictionnaire d’histoire et philosophie des sciences (1999), 4e rééd. «Quadrige»/PUF, 2006.
- Dominique Lecourt (dir.), Dictionnaire de la pensée médicale (2004), rééd. PUF/Quadrige, Paris, 2004.
Articles connexes
- Association française des biotechnologies végétales
- Bioéthique
- Biomimétisme
- Bioremédiation
- Centre de ressources biologiques
- Directive sur la brevetabilité des inventions biotechnologiques
- DisGeNET
- Génie génétique
- Nouvelles techniques de sélection des plantes
- Organisme génétiquement modifié
- Phytoremédiation
- Protéomique
- Transhumanisme
Liens externes
- Recherche sur le méta-moteur académique: scinapse.io.