Haplogroupe M (ADNmt)
L’haplogroupe M de l'ADN mitochondrial est un macro-haplogroupe mitochondrial humain (ADNmt). Représenté aujourd'hui sur tous les continents, il est, comme le macro-haplogroupe frère N, issu de l’haplogroupe L3.
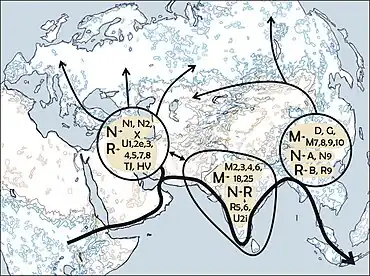
Tous les haplogroupes d'ADNmt apparus hors d'Afrique descendent, soit de l'haplogroupe M, soit de l'haplogroupe N[11]. L'haplogroupe M est relativement récent, puisque son dernier ancêtre commun est postérieur à certains sous-clades de l'haplogroupe N, comme l'haplogroupe R[12].
Origines
Les origines géographiques de l'haplogroupe M et de l'haplogroupe-frère N sont très controversées. On pense que ces deux lignées sont les deux principales branches survivantes de la dernière sortie d'Afrique, parce que toutes les lignées indigènes recensées hors d'Afrique se rattachent soit à l'haplogroupe M, soit à l'haplogroupe N. Mais pour pouvoir conclure définitivement en ce sens, il reste encore à prouver que les mutations qui définissent ces deux haplogroupes sont survenues en Afrique avant la migration de leurs porteurs hors d'Afrique. Cette question des origines de l'haplogroupe M est d'autant plus délicate qu'on en a aussi trouvé en Afrique[4].
Il est toutefois généralement admis que l'haplogroupe M est apparu peu après l'haplogroupe L3 et qu'il est son descendant. Hormis les haplogroupes M et N, les nombreux sous-clades de L3 sont très majoritairement confinés à l'Afrique, ce qui suggère que L3 est apparu en Afrique.
La question de l'haplogroupe M1
L'essentiel des questions relatives à l'origine de l'haplogroupe M tourne autour de l'importance qu'il convient d'apporter au sous-clade M1, la seule variante du macro-haplogroupe M connue en Afrique[11]. Sa présence en Afrique peut s'expliquer de deux façons :
Un élément nouveau : l'haplogroupe M23
En 2009, deux publications indépendantes ont signalé l'existence d'un sous-clade rare et ancien de l'haplogroupe M, dénommé M23, à Madagascar[14] - [15].
L'île de Madagascar a été colonisée au cours des 2000 dernières années par un métissage de colons bantous et indonésiens (austronésiens). L'haplogroupe M23 semble confiné à Madagascar (on ne l'a pas pour l'instant recensé ailleurs) : il pourrait avoir été importé à Madagascar depuis l'Asie, d'où sont originaires les plus anciens sous-clades de l'haplogroupe M.
Hypothèse d'une origine asiatique
Selon cette théorie, des hommes modernes porteurs de l'haplogroupe L3 ont quitté l'Afrique de l'Est vers l'Asie, où des mutations ont donné naissance aux haplogroupes M et N. L'haplogroupe L3 a dû s'effacer par dérive génétique puisqu'il est rare en dehors de l'Afrique. L’hypothèse selon laquelle le macro-haplogroupe M est apparu en Asie est confortée par les considérations suivantes :
- La plus forte densité au monde de porteurs du macrohaplogroupe M est observée en Asie, et notamment au Bangladesh, en Chine, en Inde, au Japon, en Corée, au Népal et au Tibet, avec une proportion variable entre 60% et 80%. La densité totale en sous-clades de M est encore plus élevée chez certaines populations de Sibérie ou des Amériques, mais ce sont des îlots de populations, qui présentent de forts effets de dérive génétique[1] - [2] - [16].
- L'ancienneté des haplogroupes « indiens » M2, M38, M54, M58, M33, M6, M61, M62 (supérieure à 50 000 ans) ainsi que la répartition du macrohaplogroupe M, n’exclut pas l'éventualité que le macrohaplogroupe M soit apparu dans la population indienne[17].
- À l'exception de l'ADN M1 spécifique à l'Afrique, l'Inde compte plusieurs lignées M directement issues de l'haplogroupe M[1] - [16].
- On ne retrouve en Afrique que deux sous-clades de l'haplogroupe M, à savoir M1 et M23, alors qu'il y en a plusieurs autres hors d'Afrique[1] - [4] (seul le cas du sous-clade M1 est controversé, cf. ci-dessous).
- En ce qui concerne plus particulièrement M1 :
- L'haplogroupe M1 est concentré dans certaines parties de l'Afrique : il n'est présent, et encore en faibles proportions, que chez les Nord-Africains et les Éthiopiens. Si M était originaire d'Afrique avant la migration de l'Homme hors d'Afrique, il serait représenté sur une aire beaucoup plus étendue[16].
- Selon Gonzalez et al. 2007, M1 se serait diffusé à une date relativement récente, car les mutations propres à M1 sont plus récentes que les lignées M exclusivement asiatiques[4].
- La répartition géographique de M1 en Afrique se concentre très majoritairement en Afrique du Nord[4] et elle est confinée aux populations de locuteurs afro-asiatiques[18], alors que les sous-clades sub-sahariens des haplogroupes L3 et L2 ont une ancienneté comparable[11].
- L'une des lignées basales de M1 a été identifiée au Maghreb et au Proche-Orient, mais elle est absente d'Afrique de l'Est[4].
- M1 n'est pas confiné à l'Afrique : il est assez répandu sur le pourtour méditerranéen, avec un pic de concentration dans la péninsule Ibérique. M1 est également largement attesté dans les populations du Moyen-Orient, entre le Sud de la Péninsule arabique et l’Anatolie, et du Levant à l'Iran. En outre, les haplotypes M1 ont été recensés ponctuellement au Caucase et en Transcaucasie, sans voisinage avec des porteurs de lignées L[4] - [11]. M1 a encore été recensé en Asie centrale, et semble s'être diffusé jusqu'au Tibet[4].
- Le fait que les mutations du sous-clade M1 ont commencé avant celles de l'haplogroupe U6 (propre à des Eurasiens dont la présence en Afrique s'explique par une re-migration depuis le Proche-Orient) et se sont poursuivies au-delà ; et le fait que l'aire de répartition de U6 en Afrique soit elle aussi confinée à l'Afrique du Nord et à la Corne de l'Afrique, renforce le scenario selon lequel M1 et U6 sont les indices d'un même mouvement de population d'Asie vers l'Afrique[18].
- La datation de la re-migration supposée des porteurs de M1 et de U6 du Proche-Orient vers l'Afrique (entre 45 000 et 40 000 ans AP) est confortée par sa simultanéité avec le changement climatique qui a repoussé les zones désertiques d'Afrique du Nord, rendant l'Afrique plus hospitalière à des populations venues d'Arabie. Ce changement climatique est d'ailleurs en partie contemporain de l'arrivée en Europe de populations porteuses de l'haplogroupe U5, clade européen parent de l'haplogroupe U6[18].
Hypothèse d'une origine africaine
Selon cette théorie, les haplogroupes M et N seraient des mutations de L3 survenues parmi une population d'Afrique de l'est qui se serait isolée des autres populations de ce continent. Les membres de cette population auraient migré hors d'Afrique et n'auraient été porteurs que des lignées M et N. À l'exception éventuelle de l'haplogroupe M1, tous les autres clades M et N d'Afrique se seraient éteints par dérive génétique[7] - [13].
L'origine africaine de l'haplogroupe M est confortée par les arguments et preuves suivants :
- L3, le clade parent de l'haplogroupe M, se retrouve dans toute l'Afrique, et fort peu hors d'Afrique[13]. Selon Toomas Kivisild (2003), « l'absence de lignées L3 autres que M et N en Inde et dans les mitochondries non-africaines en général, suggère que les premières migrations de l'homme moderne emportaient déjà ces deux ancêtres ADNmt, via route différente empruntant la corne de l'Afrique[7]. »
- Cette même étude fournit des indices montrant que M1, ou son ancêtre, avait une origine asiatique. La plus ancienne voie de diffusion de M1 hors d'Afrique a emprunté la voie nord-ouest, peuplant la Péninsule ibérique et même affectant les Basques. La majorité des lignées M1a venues, elles, d'Afrique orientale, ont une origine plus récente. Les deux lignées M1 (ouest et est) ont participé à la colonisation du Sahara au Néolithique. Le parallélisme frappant entre l'âge des sous-clade et la répartition géographique de M1 et de son homologue Nord-africain U6 confirme nettement ce scénario. Enfin, une fraction considérable des lignées M1a présentes actuellement sur le continent européen et les îles périphériques ont probablement une ascendance maternelle juive plutôt qu'arabo-berbère[4].
Diffusion
Plusieurs études suggèrent que les ancêtres de l'haplogroupe M moderne se sont diffusés de l'Afrique à la Nouvelle-Guinée et l'Australie via une route méridionale, longeant la Corne de l'Afrique et les côtes de l'Asie. Ces études avancent que les migrations des haplogroupes M et N ont suivi des routes très différentes : les porteurs de l'haplogroupe N ont migré depuis l'Afrique de l'Est vers le nord et le Levant. Toutefois, les résultats de plusieurs études récentes indiquent que la migration hors d'Afrique s'est faite en une fois et que les groupes porteurs des haplogroupes M et N ont participé à cette même migration. Cela repose sur l'analyse de populations génétiquement isolées vivant encore le long de la route de migration présumée d'Afrique en Australie, où l'on retrouve à la fois les haplogroupes N et M[3] - [19].
L'haplogroupe M35a a été retrouvé chez un individu au Sri Lanka daté entre 5 455 et 5 375 ans. L'âge de l'haplogroupe est estimé entre 16 000 et 10 000 ans. L'haplogroupe M18a a été retrouvé chez un individu daté entre 9 695 et 9 545 ans et situé géographiquement à quelques kilomètres du premier. L'âge de l'haplogroupe est estimé à 9 200 ans. Ces deux haplogroupes existent aujourd'hui au Sri Lanka: M18a parmi les Cingalais, et les Tamouls. M35a est plus fréquent chez les Cingalais et moins fréquent chez d'autres populations du Sri Lanka comme les Adivasis[20].
Répartition
L'haplogroupe M de l'ADNmt est le plus répandu en Asie[21], avec des pics de fréquence au Japon et au Tibet, où il représente plus de 70% des lignées maternelles (160/216 = 74% des Tibétains[22], 205/282 = 73% des habitants du Tōkai[23], 231/326 = 71% des habitants de l'île d'Okinawa[23], 148/211 = 70% des Japonais[2], 150/217 = 69% des habitants de l'île d'Hokkaidō[24], 24/35 = 69% des Tibétains de Shangri-La, 175/256 = 68% des habitants du nord de l'île de Kyūshū[23], 38/56 = 68% des Tibétains du Qinghai, 16/24 = 67% des Tibétains du Diqing, 66/100 = 66% des habitants de Miyazaki, 33/51 = 65% des Aïnous, 214/336 = 64% des habitants de Tōhoku[23], 75/118 = 64% des habitants de l'aire métropolitaine de Tokyo (JPT)[25]) et est partout présent en Inde[1] - [11] - [26] et en Corée du Sud[23] - [27] - [28] - [29] - [30], où il est porté par environ 60% de la population. Chez les Chinois, résidents ou non en Chine, l'haplogroupe M représente approximativement 50% de l'ADNmt, mais sa frequence varie entre 40% parmi les Han du Hunan et du Fujian en Chine méridionale, à 60% au Shenyang, dans le Liaoning en Chine du nord-est[22] - [23] - [25] - [28].
L'haplogroupe M33c est partagée entre populations en Chine (les Han, les Zhuang, les Yao, les Miao et les Tibétains)[31] et en Asie du Sud-Est (les Viêt du Viêt Nam et les Thaï de la Thaïlande)[31] et les Juifs ashkénazes (de Biélorussie, Lituanie, Lettonie, Ukraine, Pologne, Hongrie et Roumanie)[32].
L'haplogroupe M9 comprend deux macro-branches (M9a'b et E) dans les populations humaines actuelles. La macro-branche M9a'b est actuellement distribuée en Asie de l'Est continentale avec une origine méridionale et une expansion vers le nord entre environ 28 000 à 18 000 ans. En revanche, la macro-branche E est principalement distribuée dans l'île d'Asie du Sud-Est (ISEA) et les îles Salomon de Mélanésie, reflétant l'expansion néolithique des locuteurs austronésiens[33].
La découverte d'une lignée basale M9 éteinte dans le Yunnan suggère une riche diversité matrilinéaire des populations humaines dans le sud de l'Asie de l'Est à la fin du Pléistocène[33].
Références
- Cf. Rajkumar et al., « Phylogeny and antiquity of M macrohaplogroup inferred from complete mt DNA sequence of Indian specific lineages », BMC Evolutionary Biology, vol. 5, , p. 26 (PMID 15804362, PMCID 1079809, DOI 10.1186/1471-2148-5-26)
- Cf. Sayaka Maruyama, Kiyoshi Minaguchi et Naruya Saitou, « Sequence polymorphisms of the mitochondrial DNA control region and phylogenetic analysis of mtDNA lineages in the Japanese population », Int J Legal Med, vol. 117, no 4, , p. 218–225 (PMID 12845447, DOI 10.1007/s00414-003-0379-2)
- Cf. V Macaulay, C Hill, A. Achilli et al., « Single, Rapid Coastal Settlement of Asia Revealed by Analysis of Complete Mitochondrial Genomes », Science, vol. 308, no 5724, , p. 1034–6 (PMID 15890885, DOI 10.1126/science.1109792): « Haplogroup L3 (the African clade that gave rise to the two basal non-African clades, haplogroups M and N) is 84 000 ans old, and haplogroups M and N themselves are almost identical in age at 63,000 years old, with haplogroup R diverging rapidly within haplogroup N 60,000 years ago. »
- Cf. Gonzalez et al., « Mitochondrial lineage M1 traces an early human backflow to Africa », BMC Genomics, vol. 8, , p. 223 (PMID 17620140, PMCID 1945034, DOI 10.1186/1471-2164-8-223)
- Cf. KK Abu-Amero, JM Larruga, VM Cabrera, AM González et al., « Mitochondrial DNA structure in the Arabian Peninsula », BMC Evolutionary Biology, vol. 8, , p. 45 (PMID 18269758, PMCID 2268671, DOI 10.1186/1471-2148-8-45)
- Cf. M Kivisild, T Kivisild et E. Metspalu, « Most of the extant mtDNA boundaries in South and Southwest Asia were likely shaped during the initial settlement of Eurasia by anatomically modern humans », BMC Genetics, vol. 5, , p. 26 (PMID 15339343, PMCID 516768, DOI 10.1186/1471-2156-5-26)
- Cf. T. Kivisild, S. Rootsi, M. Metspalu, S. Mastana et al., « The Genetic Heritage of the Earliest Settlers Persists Both in Indian Tribal and Caste populations », American Journal of Human Genetics, vol. 72, no 2, , p. 313–32 (PMID 12536373, PMCID 379225, DOI 10.1086/346068)
- Patricia Marrero, Khaled K. Abu-Amero, Jose M. Larruga et Vicente M. Cabrera, « Carriers of human mitochondrial DNA macrohaplogroup M colonized India from southeastern Asia », BMC Evolutionary Biology, vol. 16, no 1, , p. 246 (PMID 27832758, PMCID 5105315, DOI 10.1186/s12862-016-0816-8)
- Cf. Lluís Quintana-Murci et al., « Where West Meets East: The Complex mtDNA Landscape of the Southwest and Central Asian Corridor », Am. J. Hum. Genet., vol. 74, no 5, , p. 827–45 (PMID 15077202, PMCID 1181978, DOI 10.1086/383236)
- Cf. M. van Oven, M. Kayser et al., « Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation », Human Mutation, vol. 30, no 2, , E386–E394 (PMID 18853457, DOI 10.1002/humu.20921)
- Cf. M Metspalu, T Kivisild, E Metspalu, J Parik et al., « Most of the extant mtDNA boundaries in South and Southwest Asia were likely shaped during the initial settlement of Eurasia by anatomically modern humans », BMC Genetics, vol. 5, , p. 26 (PMID 15339343, PMCID 516768, DOI 10.1186/1471-2156-5-26)
- (en) R. Fregel, V. Cabrera, J.M. Larruga, K.K. Abu-Amero et A.M. González, « Carriers of Mitochondrial DNA Macrohaplogroup N Lineages Reached Australia around 50,000 Years Ago following a Northern Asian Route », PLOS ONE, vol. 10, no 6, , e0129839 (PMID 26053380, PMCID 4460043, DOI 10.1371/journal.pone.0129839)
- Quintana et al., « Genetic evidence of an early exit of Homo sapiens sapiens from Africa through eastern Africa », nature genetics, vol. 23, (lire en ligne)
- V Dubut, F Cartault, C Payet, MD Thionville, P Murail et al., « Complete mitochondrial sequences for haplogroups M23 and M46: insights into the Asian ancestry of the Malagasy population », Human Biology, vol. 81, no 4, , p. 495–500 (PMID 20067372, DOI 10.3378/027.081.0407)
- Cf. FX Ricaut, H Razafindrazaka, M.P. Cox et al., « A new deep branch of eurasian mtDNA macrohaplogroup M reveals additional complexity regarding the settlement of Madagascar », BMC Genomics, vol. 10, , p. 605 (PMID 20003445, PMCID 2808327, DOI 10.1186/1471-2164-10-605)
- Cf. Thangaraj et al., « In situ origin of deep rooting lineages of mitochondrial Macrohaplogroup 'M' in India] », BMC Genomics, vol. 7, , p. 151 (lire en ligne).
- Cf. Kumar S Chandrasekar Adimoolam, J Sreenath, BN Sarkar, BP Urade et al., « Updating Phylogeny of Mitochondrial DNA Macrohaplogroup M in India: Dispersal of Modern Human in South Asian Corridor », PLoS ONE, vol. 4, no 10, , e7447 (PMID 19823670, PMCID 2757894, DOI 10.1371/journal.pone.0007447)
- Olivieri et al. (2006), The mtDNA legacy of the Levantine early Upper Palaeolithic in Africa, Science. 2006 Dec 15;314(5806):1767-70
- G Hudjashov, T Kivisild, PA Underhill et al., « Revealing the prehistoric settlement of Australia by Y chromosome and mtDNA analysis », Proceedings of the National Academy of Sciences of the United States of America, vol. 104, no 21, , p. 8726–30 (PMID 17496137, PMCID 1885570, DOI 10.1073/pnas.0702928104)
- (en) A. S. Fernando, A. Wanninayaka, D. Dewage et al., The mitochondrial genomes of two Pre-historic Hunter Gatherers in Sri Lanka, Journal of Human Genetics, 30 novembre 2022, doi.org/10.1038/s10038-022-01099-w
- Cf. Ghezzi et al., « Mitochondrial DNA haplogroup K is associated with a lower risk of Parkinson's disease in Italians », European Journal of Human Genetics, vol. 13, no 6, , p. 748–752 (PMID 15827561, DOI 10.1038/sj.ejhg.5201425)
- Cf. Fuyun Ji, Mark S. Sharpley, Olga Derbeneva et al., « Mitochondrial DNA variant associated with Leber hereditary optic neuropathy and high-altitude Tibetans », PNAS, vol. 109, no 19, , p. 7391–7396 (PMID 22517755, PMCID 3358837, DOI 10.1073/pnas.1202484109)
- Cf. Kazuo Umetsu, Masashi Tanaka, Isao Yuasa et al., « Multiplex amplified product-length polymorphism analysis of 36 mitochondrial single-nucleotide polymorphisms for haplogrouping of East Asian populations », Electrophoresis, vol. 26, no 1, , p. 91–98 (PMID 15624129, DOI 10.1002/elps.200406129)
- Cf. Masaru Asari, Kazuo Umetsu, Noboru Adachi et al., « Utility of haplogroup determination for forensic mtDNA analysis in the Japanese population », Legal Medicine, vol. 9, no 5, , p. 237–240 (PMID 17467322, DOI 10.1016/j.legalmed.2007.01.007)
- Cf. H-X Zheng, S Yan, Z-D Qin, Y Wang, J-Z Tan et al., « Major Population Expansion of East Asians Began before Neolithic Time: Evidence of mtDNA Genomes », PLoS ONE, vol. 6, no 10, , e25835 (PMID 21998705, PMCID 3188578, DOI 10.1371/journal.pone.0025835)
- Cf. Edwin et al., « Mitochondrial DNA diversity among five tribal populations of southern India », Current Science, vol. 83, (lire en ligne)
- Cf. W Kim, T-K Yoo, D-J Shin, H-W Rho, H-J Jin et al., « Mitochondrial DNA Haplogroup Analysis Reveals no Association between the Common Genetic Lineages and Prostate Cancer in the Korean Population », PLoS ONE, vol. 3, no 5, , e2211 (PMID 18493608, PMCID 2376063, DOI 10.1371/journal.pone.0002211)
- Cf. H-J Jin, C Tyler-Smith et W Kim, « The Peopling of Korea Revealed by Analyses of Mitochondrial DNA and Y-Chromosomal Markers », PLoS ONE, vol. 4, no 1, , e4210 (PMID 19148289, PMCID 2615218, DOI 10.1371/journal.pone.0004210)
- Cf. Miroslava Derenko, Boris Malyarchuk, Tomasz Grzybowski et al., « Phylogeographic Analysis of Mitochondrial DNA in Northern Asian Populations », Am. J. Hum. Genet., vol. 81, no 5, , p. 1025–1041 (PMID 17924343, PMCID 2265662, DOI 10.1086/522933)
- Cf. Seung Beom Hong, Ki Cheol Kim et Wook Kim, « Mitochondrial DNA haplogroups and homogeneity in the Korean population », Genes & Genomics, vol. 36, no 5, , p. 583–590 (DOI 10.1007/s13258-014-0194-9)
- (en) Jiao-Yang Tian, Hua-Wei Wang, Yu-Chun Li, Wen Zhang, Yong-Gang Yao et al., « A Genetic Contribution from the Far East into Ashkenazi Jews via the Ancient Silk Road », Scientific Reports, vol. 5, , numéro de l'article 8377 (PMID 25669617, DOI 10.1038/srep08377)
- (en) Kevin A. Brook, The Maternal Genetic Lineages of Ashkenazic Jews, Academic Studies Press, (ISBN 978-1644699843, DOI 10.2307/j.ctv33mgbcn), p. 80
- (en) Xiaoming Zhang, Xueping Ji, Chunmei Li et al., A Late Pleistocene human genome from Southwest China, cell.com, 14 juillet 2022, doi.org/10.1016/j.cub.2022.06.016