Haplogroupe N (ADNmt)
L'haplogroupe N est un haplogroupe d'ADN mitochondrial humain (ADNmt). N est un macrohaplogroupe dont les lignées descendantes sont présentes sur de nombreux continents. Comme son groupe frère M, le macrohaplogroupe N est un descendant de l'haplogroupe L3.
Eurasie
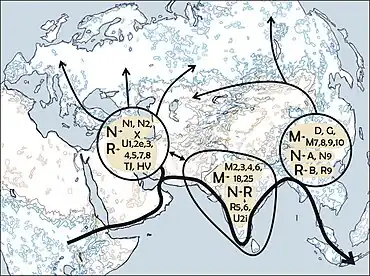
Tous les haplogroupes d’ADNmt trouvés en dehors de l'Afrique sont les descendants de l’haplogroupe N ou de son groupe frère M. M et N illustrent la théorie de l'origine africaine de l'Homme moderne et de sa sortie d'Afrique à une date récente vers l'Eurasie. La répartition mondiale des haplogroupes N et M semble indiquer que N et M ont évolué en dehors du continent africain[2]. Une division majeure de population près du golfe Persique expliquerait l'omniprésence de l'haplogroupe N et l'absence de l'haplogroupe M en Eurasie occidentale. La question de savoir si les mutations qui définissent l'haplogroupe N lui-même se sont produites pour la première fois en Asie ou en Afrique reste toutefois débattue.
Buran-Kaya (Crimée)
L'analyse du génome d'un individu fossile daté de 36 000 ans environ, originaire de Buran-Kaya III en Crimée (Buran-Kaya3A), confirme que les groupes du Gravettien seraient issus d'une branche orientale de la population fondatrice de chasseurs-cueilleurs européens qui se serait étendue depuis la Russie actuelle vers l'ouest[3] - [4]. Il a été déterminé que l'haplogroupe mitochondrial de Buran-Kaya3A appartenait à l'une des premières branches de la lignée N, à savoir N1. De manière surprenante, cette attribution se situe en dehors des lignées précédemment rapportées pour l'Europe du Paléolithique supérieur, qui proviennent presque toutes de branches N plus récentes (haplogroupes U et R). Le N1 de Buran-Kaya3A se distingue notablement de l'haplogroupe mitochondrial N identifié à partir de la mandibule âgée d'environ 40 000 ans de Peștera cu Oase, en Roumanie, qui appartient à une branche plus basale et qui n'a pas de descendants modernes. En outre, le N1 de Buran-Kaya3A est porteur de trois des huit mutations survenues avant N1b, un rare haplogroupe, fréquent au Proche-Orient et diffus de l'Eurasie occidentale à l'Afrique[4]. Les descendants du nœud N1b incluent N1b2, avec les mutations A1719G, C5507A, A12026G, T15784C et A16258C, présent actuellement en Somalie[5] et Yémen[6], et N1b1b1, avec la mutation T14581C, présent dans près de 6 % des haplogroupes Juifs ashkénazes et ça existe aussi chez les Juifs séfarades[7].
Ainsi, les séquences mitochondriales ramifiées à la fois en amont et en aval de la séquence Buran-Kaya3A peuvent être retracées jusqu'au Proche-Orient, et la présence moderne en Europe d'haplogroupes descendants de la branche N1 (N1b1b et N1a1a) à laquelle appartient Buran-Kaya3A semble être due à des migrations ultérieures depuis le Proche-Orient[4]. L'analyse a déterminé que Buran-Kaya3A possédait 3,4 % d'ascendance néandertalienne, un niveau typique des Eurasiens de l'époque, mais légèrement supérieur aux Eurasiens actuels (environ 2 %)[4].
Néolithique
L'haplogroupe N1a1a est l'une des deux lignées d'ADNmt - l'autre étant K1a - observées chez les agriculteurs du Néolithique ancien d'Anatolie centrale. En Grèce, il n'est enregistré qu'au Néolithique moyen et tardif[8].
N1a est l’haplogroupe le plus caractéristique des premiers agriculteurs de la plaine de Pannonie et d'Europe centrale. La présence de N1a chez les premiers agriculteurs de Pannonie (6,82 à 10,26 %) et en Europe centrale renforce encore son rôle de marqueur de la route danubienne de l'expansion néolithique. A contrario, les haplogroupes N1a de l'ADNmt et NRY G2a sont rares parmi les populations européennes d'aujourd'hui, ce qui reflète la différence entre les populations des cultures du VIe millénaire av. J.-C. et les populations actuelles vivant en Europe[9].
Références
- Metspalu et al., 2004
- (en) A. Olivieri, A. Achilli, M. Pala, V. Battaglia, S. Fornarino, N. Al-Zahery, R. Scozzari, F. Cruciani, D. M. Behar, J.-M. Dugoujon, C. Coudray, A. S. Santachiara-Benerecetti, O. Semino, H.-J. Bandelt et A. Torroni, « The mtDNA Legacy of the Levantine Early Upper Palaeolithic in Africa », (PMID 17170302, DOI 10.1126/science.1135566, Bibcode 2006Sci...314.1767O) : « The scenario of a back-migration into Africa is supported by another feature of the mtDNA phylogeny. Haplogroup M's Eurasian sister clade, haplogroup N, which has a very similar age to M and no indication of an African origin », p. 1767–70
- (en) David Reich, Who We Are and How We Got Here, Oxford University Press, 2018
- (en) E. Andrew Bennett et al, The origin of the Gravettians: genomic evidence from a 36,000-year-old Eastern European, biorxiv.org, juillet 2019
- YFull MTree: N1b2
- « Homo sapiens isolate Y333 haplogroup N1b2 mitochondrion, complete genome » dans la base GenBank
- (en) Kevin A. Brook, The Maternal Genetic Lineages of Ashkenazic Jews, Academic Studies Press, (ISBN 978-1644699843, DOI 10.2307/j.ctv33mgbcn), p. 15, 82-83
- (en) Nuno M. Silva, Susanne Kreutzer et al., Ancient mitochondrial diversity reveals population homogeneity in Neolithic Greece and identifies population dynamics along the Danubian expansion axis, nature.com, Scientific Reports, volume 12, n° 13474, 2022, doi.org/10.1038/s41598-022-16745-8
- (en) Anna Szécsényi-Nagy et al., « Tracing the genetic origin of Europe's first farmers reveals insights into their social organization », 2015 apr 22