Barnase
La barnase est une ribonucléase bactérienne de 110 résidus d'acides aminés ; le nom « barnase » est un mot-valise pour l'anglais bacterial RNase. Cette enzyme catalyse le clivage de l'ARN à la suite d'un résidu de guanosine, c'est-à-dire sur son côté 3’. La catalyse emploie un mécanisme acide-base faisant intervenir les résidus de glutamate Glu73 et d'histidine His102, le premier comme base et le second comme acide. Le résidu de lysine Lys27 est également indispensable à l'activité enzymatique en se liant à l'état de transition du substrat, et ce bien qu'il n'intervienne pas directement dans la catalyse[2].
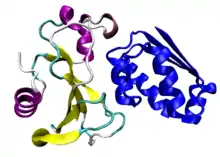
L'inhibiteur barstar est ici représenté en bleu.
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
La barnase est synthétisée et sécrétée par la Bacillus amyloliquefaciens, une bactérie. Elle est létale pour la cellule lorsqu'elle n'est pas exprimée avec son inhibiteur, la barstar, une protéine qui forme avec cette enzyme un complexe à l'interaction protéine-protéine particulièrement forte tant que l'ensemble demeure dans le cytoplasme[3] - [4]. Cet inhibiteur couvre le site actif de l'enzyme, ce qui empêche cette dernière de dégrader l'ARN à l'intérieur de la cellule.
La barnase n'a pas de pont disulfure et n'a besoin d'aucun cation métallique divalent ni de groupe prosthétique pour adopter la conformation de son état natif. Elle a par conséquent fait l'objet de nombreuses recherches pour comprendre les mécanismes du repliement des protéines[5] - [6] - [7].
Notes et références
- (en) Ashley M. Buckle, Gideon Schreiber et Alan R. Fersht, « Protein-protein recognition: Crystal structural analysis of a barnase-barstar complex at 2.0-Å resolution », Biochemistry, vol. 33, no 30, , p. 8878-8889 (PMID 8043575, DOI 10.1021/bi00196a004, lire en ligne)
- (en) Danuta E. Mossakowska, Kerstin Nyberg et Alan R. Fersht, « Kinetic characterization of the recombinant ribonuclease from Bacillus amyloliquefaciens (barnase) and investigation of key residues in catalysis by site-directed mutagenesis », Biochemistry, vol. 28, no 9, , p. 3843-3850 (PMID 2665810, DOI 10.1021/bi00435a033, lire en ligne)
- (en) Robert W. Hartley, « Barnase and barstar: two small proteins to fold and fit together », Trends in Biochemical Science, vol. 14, no 11, , p. 450-454 (PMID 2696173, DOI 10.1016/0968-0004(89)90104-7, lire en ligne)
- (en) Robert W. Hartley, « Barnase and barstar: Expression of its cloned inhibitor permits expression of a cloned ribonuclease », Journal of Molecular Biology, vol. 202, no 4, , p. 913-915 (PMID 3050134, DOI 10.1016/0022-2836(88)90568-2, lire en ligne)
- (en) Luis Serrano, James T. Kellis Jr, Pauline Cann, Andreas Matouschek et Alan R. Fersht, « The folding of an enzyme: II. Substructure of barnase and the contribution of different interactions to protein stability », Journal of Molecular Biology, vol. 224, no 3, , p. 783-804 (PMID 1569557, DOI 10.1016/0022-2836(92)90562-X, lire en ligne)
- (en) Luis Serrano, Andreas Matouschek et Alan R. Fersht, « The folding of an enzyme: III. Structure of the transition state for unfolding of barnase analysed by a protein engineering procedure », Journal of Molecular Biology, vol. 224, no 3, , p. 805-818 (PMID 1569558, DOI 10.1016/0022-2836(92)90563-Y, lire en ligne)
- (en) Andreas Matouschek, Luis Serrano et Alan R. Fersht, « The folding of an enzyme: IV. Structure of an intermediate in the refolding of barnase analysed by a protein engineering procedure », Journal of Molecular Biology, vol. 224, no 3, , p. 819-835 (PMID 1569559, DOI 10.1016/0022-2836(92)90564-Z, lire en ligne)