Nucléase micrococcale
La Nucléase micrococcale (Nomenclature EC 3.1.31.1) est une endo-exonucléase qui agit de préférence sur acides nucléiques simple brin en produisant des mononucléotides et oligonucléotides 3' phosphate. Le taux d'excision de cette enzyme est 30 fois plus fort lorsqu'elle agit sur l'extrémité 5' d'une adénine ou thymine par rapport à la guanine ou la cytosine. Cette enzyme possède aussi une activité contre l'ARN et l'ADN double brin, résultant finalement en un clivage de toutes les séquences.
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
Synonymes
Les autres noms de cette enzyme sont : nucléase de micrococque, nucléase S7, MNase, endonucléase de la rate, thermonucléase, nucléase T, endonucléase micrococcale, nucléase T', nucléase staphylococcale, phosphodiestérase de la rate, nucléase Staphylococcus aureus, nucléase B Staphylococcus aureus, 3'-nucléotidohydrolase ribonucléate (desoxynucléate)
Caractéristiques
Cette enzyme possède un poids moléculaire de 16,9 KDa, et est strictement Ca2+-dépendant. Son pH optimal varie selon la concentration de Ca2+, et elle est de ce fait facilement inactivée par l'agent complexant EGTA[1].
Source
Cette enzyme est la nucléase extracellulaire de Staphylococcus aureus. Deux souches, V8 et Foggi produisent des formes pratiquement identiques de cette enzyme[2]. Une source courante de production est une souche recombinante de Escherichia coli qui possède le gène de la nucléase de staphylocoque.
Structure
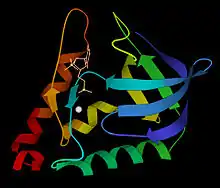
La structure tridimensionnelle de la nucléase micrococcale (qui par alors était connue sous le nom de nucleasa staphyilococcale) a été résolue en 1969, un des premiers accomplissements dans l'histoire de la cristallographie par rayons X de protéines, en étant déposée sur la Protein Data Bank avec le code d'archive 1SNS (maintenant obsolète)[3]. Des structures plus récentes et de grande résolution sont disponibles pour la forme apolipoprotéine (code PDB 1SNO), et pour la forme inhibée liée au diphosphate de thymidine (codes PDB 3H6M 3H6M ou PDB 1SNO 1SNC)
Comme on peut remarquer sur le diagramme en ruban ci-dessus, la molécule de cette nucléase possède 3 hélices alpha et un feuillet beta en forme de tonneau formée par cinq chaînes, un arrangement appelé repliement OB (par oligonucleótide-binding, c'est-à-dire fixateur d'oligonucléotides en anglais), selon la classification de la base de données Structural Classification of Proteins (SCOP).
Applications
- Hydrolyse d'acides nucléiques en extraits cellulaires crus.
- Séquençage de l'ARN (du fait de la dégradation de l'ADN)
- Préparation de lysats de réticulocytes de lapin.
- Études de la structure de la chromatine.
- Ablation d'acides nucléiques de préparations de protéines en laboratoire, pour permettre des études de conformations tridimensionnelles protéiques et relations structure-fonction.
- Recherche sur les mécanismes de repliement protéique.
Notes et références
- Heins JN, Suriano JR, Taniuchi H, Anfinsen CB (1967).
- Cusumano CL, Taniuchi H, Anfinsen CB (1968).
- Arnone A, Bier J, et al. (1971).
- http://www.thermoscientificbio.com/uploadedfiles/resources/en0181-usa-msds.pdf - Fiche normes de sécurité du produit (anglais)
- http://www.thermoscientificbio.com/uploadedfiles/resources/en018-product-information.pdf - Fiche d'information produit (anglais)
Liens externes
- (en) MeSH Micrococcal+Nuclease: Micrococcal+Nuclease (En anglais)
- Nomenclature EC 3.1.31.1