Clustered Regularly Interspaced Short Palindromic Repeats
En génétique, les Clustered Regularly Interspaced Short Palindromic Repeats (« Courtes répétitions palindromiques groupées et régulièrement espacées »), plus fréquemment désignées sous le nom de CRISPR (acronyme prononcé /ˈkrɪspəʳ/), sont des familles de séquences répétées dans l'ADN. De telles familles se caractérisent par des séries de répétitions directes courtes (de 21 à 37 paires de bases) et régulièrement espacées par des séquences appelées « spacer », généralement uniques, de 20 à 40 paires de bases.
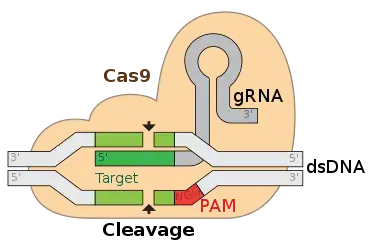
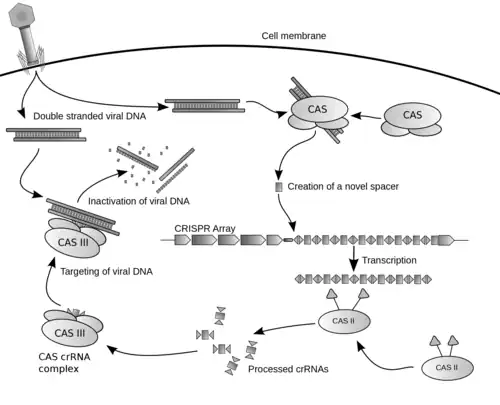
En génie génétique, le système CRISPR-Cas9, d'abord utilisé pour typer des souches de bactéries est récemment devenu un outil de manipulation génétique à fort potentiel[1]. CRISPR-Cas9 est notamment utilisé comme ciseau moléculaire afin d'introduire des modifications locales du génome (manipulations souvent qualifiées d'édition génomique) de nombreux organismes modèles.
Première observation et redécouvertes successives
Cette structure répétée a été observée pour la première fois par Yoshizumi Ishino[2] en 1987 chez Escherichia coli. Elle a ensuite été décrite plusieurs fois sous différents noms :
- LCTR pour Long Clusters of Tandem Repeats. Deux de ces régions sont associées à des LCTR (typique des Archæa) ce qui supporte l’idée que ces LCTR soient impliqués dans le transfert de gènes[3] ;
- SPIDR pour Spacer Interspaced Direct Repeats[4] ;
- TREP pour Tandem REPeats[5] ;
- DVR pour Direct Variable Repeats[6] ;
- SRSR pour Short Regularly Spaced Repeats[7].
Un article de Juan Francisco Martínez Mojica de 2000[7] démontre que toutes les descriptions précédentes n'étaient que des facettes d'une seule et même entité. En 2002, Jansen décide, avec l'accord du groupe de Mojica, de clarifier la nomenclature en créant l'acronyme CRISPR[8].
Si la séquence répétée est bien conservée au sein d'un organisme, le nombre d'unités au sein d'un train, le nombre de trains et même la présence de trains sont des quantités hautement variables d'une lignée à une autre[9]. De fait, les CRISPR ont été utilisés pour typer des souches bactériennes, une technique appelée spoligotypage[10] - [11].
Répartition dans le monde vivant
Ces répétitions se rencontrent dans les lignées d'archées et de bactéries, mais n'ont pas encore été observées chez les eucaryotes. Chez les virus, il a été montré que des bactériophages codent le système CRISPR-Cas[12]. Les CRISPR pourraient être la famille de répétition la plus largement répandue dans le monde vivant[7], avec un peu moins de la moitié des organismes séquencés porteurs de ce type de répétitions (sur plus de 200 génomes testés à la fin de 2005[13]). Les régions CRISPR sont présents chez la plupart des archées thermophiles et la moitié des espèces bactériennes (jusqu'à 10 % du génome chez certaines archées analysées) [14], concernant aussi bien les bactéries à Gram+ que les bactéries à Gram-[15]. Certains plasmides d'archées sont porteurs de CRISPR homologues de ceux portés par l'organisme hôte. C'est le cas par exemple de Sulfolobus et du plasmide pNOB8[16] - [17]. Certains auteurs ont signalé la présence de CRISPR dans l'ADN mitochondrial (Flamand, 1992), mais ces résultats n'ont pas pu être reproduits.
Structure
Les locus CRISPR sont caractérisés par une alternance de répétitions directes ou « direct repeats » (c'est-à-dire toutes orientées dans le même sens de lecture), courtes (de 21 à 37 paires de bases) et régulièrement espacées par des séquences appelées « spacers », généralement uniques, de 20 à 40 paires de bases. Les séquences nucléotidiques et la longueur des locus CRISPR sont conservées pour une même espèce, mais varient d'une espèce à l'autre[4] - [18]. Les locus CRISPR sont généralement adjacents aux gènes cas (pour « CRISPR associated »)[18], dont ils sont séparés par une séquence de 300 à 500 paires de bases, appelée séquence « leader »[8].
Les premiers éléments sur la façon dont la structure CRISPR elle-même (c'est-à-dire la portion constituée de la succession de direct repeats et de spacers) évolue ont été fournis par le travail de Pourcel et al.[19]. Ce travail, portant sur les locus CRISPR de Yersinia pestis, a montré que l'acquisition de nouveaux spacers était polarisée, alors que la perte d'un ou plusieurs « spacers » pouvait survenir tout au long du locus CRISPR. L'acquisition survient de façon adjacente au leader. Le leader est conservé dans la lignée mais pas entre les lignées[4].
Gènes cas
Rencontrés uniquement dans les génomes porteurs de CRISPR, les gènes cas sont généralement situés à proximité des locus CRISPR. Dès 2005, au moins 45 familles de gènes de ce type ont été décrites. Les 4 premières étant strictement associées[13]. Le plus important de ces gènes est cas1, présent dans presque tous les complexes CRISPR-Cas (parfois notés CRISPR/Cas). La distribution sporadique des sous-types CRISPR-Cas suggère plusieurs évènements de transferts horizontaux au cours de l'évolution microbienne. Les systèmes CRISPR-Cas peuvent être très étendus (jusqu'à 20 gènes différents) et semblent présenter des schémas différents d'une lignée à l'autre, qui ne se retrouvent que dans un nombre très limité d'espèces. Les gènes cas repérés chez des organismes hyperthermophiles ont d'abord été vus comme jouant un rôle de réparation de l'ADN[17].
Rôles
Si les fonctions des CRISPR n'ont pas encore été clairement identifiées, un certain nombre d'hypothèses ont été avancées :
- les CRISPR sont impliqués dans la répartition des copies de génomes au cours de la division (expériences menées sur Haloferax mediterranei[5]). Des similitudes avec certains mécanismes de partition de plasmides suggèrent que les CRISPR sont des analogues des séquences de partitionnement bactériennes. Sans être vital pour la cellule puisque certaines lignées en sont dépourvues[7] ;
- les CRISPR ont un rôle de perturbateur chromosomique en permettant des recombinaisons et sont impliqués dans le système de restauration chromosomique à la suite d'un réarrangement[7] : il a été montré qu'il y a une relation forte entre les CRISPR et les réarrangements[20]. Le motif répété faciliterait les recombinaisons ;
- deux de ces régions sont associées à des LCTR (typiques des Archæa) ce qui supporte l’idée que ces LCTR sont impliqués dans le transfert de gènes[3]. Nelson a proposé que les CRISPR sont associés à l'origine de réplication et jouent un rôle dans le processus de réplication du chromosome (il a placé le nucléotide 1 comme étant le premier d'un CRISPR) ;
- chez les archées, site de fixation pour la formation de nucléosomes (enroulement de l’ADN autour de protéines similaires à des histones) permettant de protéger l’ADN ou de réguler l’expression génique[21] ;
- ces séquences forment des structures secondaires (épingles, Z-DNA et H-DNA) ayant des fonctions de régulation ou de protection[21].
Rôle « immunitaire » du système CRISPR-Cas
En 2005, on observe que les séquences de certains spacers sont identiques à celles de certains éléments génétiques mobiles, notamment de virus et de plasmides[22] - [19] - [23].
En mars 2007, l'équipe de Philippe Horvath a montré que l'exposition de cellules porteuses de CRISPR à des phages entraîne l'apparition de nouveaux intervalles, que ces intervalles dérivent du matériel génomique des phages et que le retrait ou l'ajout de ces intervalles modifie la résistance des bactéries face aux phages[24].
Le système CRISPR-Cas (CASS) est un mécanisme de défense contre les phages et plasmides invasifs, fonctionnant d'une manière analogue à celle du système ARNi des eucaryotes. En intégrant des fragments de gènes étrangers dans des parties non codantes de leur chromosomes, archées ou bactéries acquièrent une résistance aux phages et plasmides. Il s'agit donc d'une forme de système immunitaire héritable par transmission aux cellules filles, permettant aux archées et aux bactéries de s'adapter rapidement à l'évolution des phages et des plasmides[8] - [22] - [19] - [25]. En 2020, une étude explicite comment des marqueurs dérivés des génomes des différents virus rencontrés s'accumulent d'une manière chronologiquement ordonnée (à la manière d'un « enregistreur ADN ») dans la région CRISPR de leur génome[26]. C'est la phase d’immunisation. S'ensuit une immunité : les protéines Cas utilisent ces informations pour reconnaitre et désactiver tout virus de signature déjà connue[27].
Il a été montré que les phages pouvaient contourner le système CRISPR-Cas via des gènes spécifiques[28].
Les systèmes CRISPR-Cas observés chez les bactéries et les archées d'une part et le système ARNi observé chez les eucaryotes d'autre part ne semblent pas dériver d'un ancêtre commun. Ils ne sont donc pas homologues.
Mécanismes moléculaires
Les systèmes CRISPR utilisent les gènes cas1 et cas2 qui sont impliqués dans l'intégration, en tant que spacer de fragments de gènes étrangers dans le CRISPR.
Trois types de systèmes CRISPR-Cas sont connus[29] :
- les systèmes de types I, utilisent un complexe Cascade pour cliver les transcrits de CRISPR au niveau des épingles. Lorsqu'un complexe Cascade/spacer s'associe à un ADN cible (reconnaissance par hybridations) il recrute la protéine Cas3 qui clive un brin de l'ADN cible ;
- les systèmes de types II, utilisent la RNAse III pour séparer les répétitions des transcrits. La protéine Cas9 s'associe avec un fragment de transcrit et, lors de la reconnaissance d'un ADN cible, Cas9 clive les 2 brins de cet ADN ;
- les systèmes de type III, utilisent la protéine Cas6 pour cliver les transcrits de CRISPR au niveau des épingles, les segments de transcrits obtenus s'associent avec un complexe Cas10. Ce système requiert qu'il y ait transcription de l'ADN cible, le complexe Cas10/spacer clive alors un brin de l'ADN cible (brin non transcrit), ainsi que l'ARN en cours de transcription.
Utilisation en biologie moléculaire
Le système CRISPR-Cas9, notamment tel que développé par la chercheuse française Emmanuelle Charpentier sur la base d'une idée qu'elle a eue à Vienne au début des années 2000 (pour lequel elle obtient le prix Nobel) est depuis les années 2010 devenu un outil de génie génétique révolutionnaire permettant de modifier plus facilement et plus précisément les séquences d’ADN, et utilisé pour des thérapies géniques associé à un vecteur souvent viral[30].
Il est aussi utilisé pour exprimer des gènes grâce à l'activation CRISPR, entre autres pour reprogrammer des cellules.
Découverte
La technique d'édition de génome CRISPR-Cas9 a été découverte par l'équipe de la chercheuse française Emmanuelle Charpentier aidée par la professeure américaine Jennifer Doudna. Elle a été par la suite développée à partir de 2012 par plusieurs chercheurs, dont notamment le biologiste moléculaire Feng Zhang, du Broad Institute (associé à Harvard et au MIT).
En avril 2016 Charpentier a présenté dans le journal Nature une nouvelle version plus performante d'utilisation du CRISPR[1].
Brevets et entreprise
CRISPR Therapeutics, jeune entreprise cofondée par Emmanuelle Charpentier pour breveter et développer cet outil est devenue l'une des sociétés de biotechnologies précliniques les plus richement financées dans le monde, mais est en conflit concernant le brevetage de cette technologie[31] - [1].
En 2021, Wageningen University & Research a annoncé avoir décidé d'ouvrir ses brevets d'édition de gènes CRISPR-Cas à des ONG à but non lucratif (sous forme de licences gratuites) pour des applications non commerciales (par exemple pour améliorer les végétaux alimentaire cultivés dans les pays pauvres, plus rapidement que via la sélection végétale conventionnelle)[32].
Berkeley conteste devant une commission d'appel du United States Patent and Trademark Office le brevet accordé au Broad Institute pour cette découverte [33]. Le 15 février 2017, l'United States Patent and Trademark Office a considéré que les brevets déposés par le Broad Institute sur l'usage de CRISPR/Cas9 dans le cas de cellules eucaryotes étaient valides. Pour autant, les revendications de l'Université de Berkeley (à l'origine des dépôts de brevets de Jennifer Doudna et d'Emmanuelle Charpentier) quant à l'emploi de CRISPR/Cas9 sur tous types de matériel génétique (y compris les cellules eucaryotes) n'ont pas été rejetées[34] - [35]. En janvier 2018, l'Office européen des brevets a révoqué un des principaux brevets concernant CRISPR-Cas9 déposé (et accepté dans un premier temps) par le Broad Institute. Ce dernier a fait appel de la décision[36].
Éthique
Cette technique suscite des questions d'éthique médicale et environnementale quant à l'eugénisme et aux conséquences environnementales de la manipulation du génome, quand cet outil est appliqué à des cellules héréditaires[37] - [38]. Un certain nombre de scientifiques et d'autres personnalités ont lancé un appel pour une éthique de la conservation sans le pilotage des gènes, considérant qu'elle détient le potentiel de transformer absolument le monde de la nature et les rapports des humains avec celui-ci. Ce qui revient à proposer délibérément l’extinction comme outil[39].
Le congrès mondial de la nature de septembre 2016 a voté une motion demandant à la directrice générale et aux commissions de l'UICN d'évaluer « de toute urgence les incidences des techniques de forçage génétique et d'autres techniques apparentées et leurs effets possibles sur la conservation et l'utilisation durable de la diversité biologique, ainsi que le partage équitable des avantages découlant des ressources génétiques, afin que l'UICN élabore des orientations sur ce thème, tout en s'abstenant de soutenir ou d'approuver des activités de recherche, y compris des essais sur le terrain, portant sur l'utilisation de techniques de forçage génétique à des fins de conservation ou autres tant que cette évaluation n'aura pas été réalisée »[40]
Culture populaire
- Le système CRISPR-cas9 est à la base de l'expérience ratée du film Rampage : Hors de contrôle.
- Le système CRISPR-cas9 intervient dans l'intrigue du roman de Robin Cook : Pandémie.
Annexes
Bibliographie
- (en) Joy Y. Wang et Jennifer A. Doudna, « CRISPR technology: A decade of genome editing is only the beginning », Science, vol. 379, no 6629, (DOI 10.1126/science.add8643)
Articles connexes
Liens externes
- (en) « CRISPRfinder program online », sur Université Paris Sud, (consulté le ).
- Des "ciseaux" pour découper l’ADN, une révolution génétique qui n’est pas sans risque", Secrets d’info, 10 avril 2021, Jacques Monin , Sylvain Tronchet , Cellule investigation de Radio France.
- Bases de données interactives listant les CRISPR découverts à ce jour :
- (en) « CRISPRdb », (consulté le )[41].
- (en) « CRISPI: a CRISPR Interactive database », (consulté le )[42].
- Ressource relative à la santé :
Notes et références
- A. Abbott, « The quiet revolutionary: How the co-discovery of CRISPR explosively changed Emmanuelle Charpentier’s life The microbiologist spent years moving labs and relishing solitude. Then her work on gene-editing thrust her into the scientific spotlight », Nature, no 532, 28 avril 2016, pp. 432-434, doi:10.1038/532432a.
- (en) Yoshizumi Ishino, Hideo Shinagawa, Kozo Makino, Mitsuko Amemura et Atsuo Nakata, « Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product », Journal of Bacteriology, vol. 169, no 12, , p. 5429-5433 (PMID 3316184).
- (en) K. E. Nelson et al., « Evidence for lateral gene transfer between Archæa and bacteria from genome sequence of Thermotoga maritima », Nature, vol. 399, no 6734, , p. 323-9 (PMID 10360571).
- (en) Ruud Jansen, Jan D. A. van Embden, Wim Gaastra et Leo M. Schouls, « Identification of a novel family of sequence repeats among prokaryotes », OMICS, vol. 6, no 1, , p. 23-33 (PMID 11883425).
- (en) Mojica Francisco et al., « Long stretches of short tandem repeats are present in the largest replicons of the Archæa Haloferax mediterranei and Haloferax volcanii and could be involved in replicon partitioning », Molecular Microbiology, vol. 17, no 1, , p. 85-93 (PMID 7476211).
- (en) A. Aranaz, « Spoligotyping profile change caused by deletion of a direct variable repeat in a Mycobacterium tuberculosis isogenic laboratory strain », Journal of Clinical Mirobiology, vol. 42, no 11, , p. 5388-91 (PMID 15528751).
- (en) Francisco Mojica, Cesar Diez-Villaseñor, Elena Soria et Guadalupe Juez, « Biological significance of a family of regularly spaced repeats in the genomes of Archæa, Bacteria and mitochondria », Molecular Microbiology, vol. 36, no 1, , p. 244-246 (PMID 10760181).
- (en) Ruud Jansen, Jan D. A. van Embden, Wim Gaastra et Leo M. Schouls, « Identification of genes that are associated with DNA repeats in prokaryotes », Molecular Microbiology, vol. 43, no 6, , p. 1565-1575 (PMID 11952905, lire en ligne, consulté le ).
- (en) J. D. van Embden et al., « Genetic variation and evolutionary origin of the direct repeat locus of Mycobacterium tuberculosis complex bacteria », Journal of Bacteriology, vol. 182, no 9, , p. 2393-401 (PMID 10762237).
- (en) J. Kamerbeek et al., « Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology », Journal of Clinical Microbiology, vol. 35, no 4, , p. 907-14 (PMID 9157152).
- (en) Hoe et al., « Rapid molecular genetic subtyping of serotype M1 group A Streptococcus strains », Emerging Infectious Diseases, vol. 5, no 2, , p. 254-63 (PMID 10221878).
- (en) Kimberley D. Seed, David W. Lazinski, Stephen B. Calderwood et Andrew Camilli, « A bacteriophage encodes its own CRISPR/Cas adaptive response to evade host innate immunity », Nature, vol. 494, no 7438, , p. 489-91 (PMID 23446421, PMCID 3587790, DOI 10.1038/nature11927, lire en ligne, consulté le ).
- (en) Daniel H. Haft, Jeremy Selengut, Emmanuel F. Mongodin et Karen E. Nelson, « A Guild of 45 CRISPR-Associated (Cas) Protein Families and Multiple CRISPR/Cas Subtypes Exist in Prokaryotic Genomes », PLOS Computational Biology, vol. 1, no 6, , p. 1-10 (PMID 16292354, PMCID PMC1282333, lire en ligne, consulté le ).
- (en) K. S. Makarova, Y. I. Wolf et E. V. Koonin, « Comparative genomics of defense systems in archæa and bacteria », Nucleic Acids Research, vol. 41, no 8, , p. 4360-4377 (PMID 23470997, PMCID PMC3632139, DOI 10.1093/nar/gkt157, lire en ligne, consulté le ).
- (en) Reidun K. Lillestøl, Peter Redder, Roger A. Garrett et Kim Brüger, « A putative viral defense mechanism in archæal cells », Archæa, vol. 2, no 1, , p. 59-72 (PMID 16877322, PMCID PMC2685585, lire en ligne, consulté le ).
- (en) Xu Peng, Kim Brügger, Biao Shen, Lanming Chen, Qunxin She et Roger A. Garrett, « Genus-Specific Protein Binding to the Large Clusters of DNA Repeats (Short regularly Spaced Repeats) Present in Sulfolobus Genomes », Journal of Bacteriology, vol. 185, no 8, , p. 2410-7 (PMID 12670964, PMCID PMC152625, lire en ligne, consulté le ).
- (en) Makarova KS, L. Aravind, N. V. Grishin, I. B. Rogozin et E. V. Koonin, « A DNA repair system specific for thermophilic archæa and bacteria predicted by genomic context analysis », Nucleic Acid Research, vol. 30, no 2, , p. 482-96 (PMID 11788711, PMCID PMC99818, lire en ligne, consulté le ).
- (en) John van der Oost, Edze R. Westra, Ryan N. Jackson et Blake Wiedenheft, « Unravelling the structural and mechanistic basis of CRISPR-Cas systems », Nature Reviews Microbiology, vol. 12, no 7, , p. 479-492 (PMID 24909109, PMCID PMC4225775, DOI 10.1038/nrmicro3279, lire en ligne, consulté le ).
- (en) C. Pourcel, G. Salvignol et G. Vergnaud, « CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies », Microbiology, vol. 151, no 3, , p. 653-63 (PMID 15758212).
- (en) R. T. DeBoy, E. F. Mongodin, J. B. Emerson et K. E. Nelson, « Chromosome evolution in the Thermotogales: large-scale inversions and strain diversification of CRISPR sequences », Journal of Bacteriology, vol. 188, no 7, , p. 2364-74 (PMID 16547022, PMCID PMC1428405, lire en ligne, consulté le ).
- (en) Ahmed Fadiel, Stuart Lithwick, Gopi Ganji et Stephen W. Scherer, « Remarkable sequence signatures in archæal genomes », Archæa, vol. 1, no 3, , p. 185-90 (PMCID PMC2685567, lire en ligne, consulté le ).
- (en) F. J. Mojica, C. Díez-Villaseñor, J. García-Martínez et E. Soria, « Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements », Journal of molecular evolution, vol. 60, no 2, , p. 174-82 (PMID 15791728, lire en ligne, consulté le ).
- (en) A. Bolotin, B. Quinquis, A. Sorokin et S. D. Ehrlich, « Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin », Microbiology, vol. 151, no 8, , p. 2551-61 (PMID 16079334, DOI 10.1099/mic.0.28048-0, lire en ligne, consulté le ).
- (en) Rodolphe Barrangou, Christophe Fremaux, Hélène Deveau, Melissa Richards, Patrick Boyaval, Sylvain Moineau, Dennis A. Romero et Philippe Horvath, « CRISPR provides acquired resistance against viruses in prokaryotes », Science, vol. 315, no 5819, , p. 1709-1712 (PMID 17379808, lire en ligne, consulté le ).
- (en) Julie E. Samson, Alfonso H. Magadán, Mourad Sabri et Sylvain Moineau, « Revenge of the phages: defeating bacterial defences », Nature Reviews Microbiology, vol. 11, no 10, , p. 675-87 (PMID 23979432, DOI 10.1038/nrmicro3096, lire en ligne, consulté le ).
- Sungchul Kim et al., Selective loading and processing of prespacers for precise CRISPR adaptation, 2020. DOI 10.1038/s41586-020-2018-1
- La Recherche, p. 22
- (en) Joe Bondy-Denomy, April Pawluk, Karen L. Maxwell et Alan R. Davidson, « Bacteriophage genes that inactivate the CRISPR/Cas bacterial immune system », Nature (revue), vol. 493, no 7432, , p. 429-32 (PMID 23242138, DOI 10.1038/nature11723, lire en ligne, consulté le ).
- Luciano A. Marraffini, « CRISPR-Cas immunity in prokaryotes », Nature, vol. 526, , p. 55-61 (DOI 10.1038/nature15386, lire en ligne).
- (en) « Oncology : Studying cancer genomes gene by gene could improve treatment », The Economist, (lire en ligne, consulté le ).
- Heidi Ledford, How the US CRISPR patent probe will play out ; Decision could determine who profits from the gene-editing technique in future, 7 mars 2016.
- (en) John van der Oost et Louise O. Fresco, « Waive CRISPR patents to meet food needs in low-income countries », Nature, vol. 597, no 7875, , p. 178–178 (DOI 10.1038/d41586-021-02397-7, lire en ligne, consulté le )
- Kelly Servick, « Accusations of errors and deception fly in CRISPR patent fight », Science, 8 mars 2016.
- Sharon Begley, « Broad Institute prevails in heated dispute over CRISPR patents », Stat, le 15 février 2017, consulté le 17 février 2017.
- Sharon Begley, « 6 Takeaways from the CRISPR Patent Decision », Scientific American, le 16 février 2017, consulté le 17 février 2017.
- S. Willquist, « CRISPR patent revoked at the EPO », Lexology, 29 janvier 2018.
- Jean-Philippe Braly, « CRISPR-Cas9 : le couteau suisse qui révolutionne la génétique », sur Sciences actualités, (consulté le ).
- Morgane Heuclin-Reffait, « Avec «Crispr/Cas9», modifier un ADN devient presque aussi simple qu'un copier-coller », sur Libération Sciences, (consulté le )
- A Call for Conservation with a Conscience: No Place for Gene Drives in Conservation ; Vandana Shiva et d’autres écologistes lancent l’alerte sur le « pilotage des gènes ».
- Élaboration d’une politique de l’UICN sur la conservation de la biodiversité et la biologie de synthèse.
- (en) Ibtissem Grissa, Gilles Vergnaud et Christine Pourcel, « The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats », BMC Bioinformatics, vol. 8, (PMID 17521438, lire en ligne, consulté le ).
- (en) Christine Rousseau, Mathieu Gonnet, Marc Le Romancer et Jacques Nicolas, « CRISPI: a CRISPR interactive database », Bioinformatics, vol. 25, no 24, , p. 3317-3318 (PMID 19846435, PMCID PMC2788928, DOI 10.1093/bioinformatics/btp586, lire en ligne, consulté le ).