5-Énolpyruvylshikimate-3-phosphate synthase
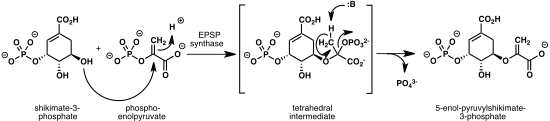
L'EPSP synthase (EPSPS), également appelée 5-énolpyruvylshikimate-3-phosphate synthase, est une transférase qui catalyse la réaction :
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
Cette enzyme est présente chez les bactéries, les mycètes, les protistes et les plantes, où elle intervient à la sixième étape de la voie du shikimate réalisant la biosynthèse de trois acides aminés aromatiques : la phénylalanine, la tyrosine et le tryptophane. Dans la mesure où cette voie métabolique est absente chez les animaux, les enzymes qui la constituent sont des cibles privilégiées pour le développement d'antibiotiques, de parasiticides et d'herbicides non toxiques pour l'homme ; l'EPSP synthase est ainsi la cible du glyphosate, principe actif du Roundup, herbicide distribué par la société Monsanto.
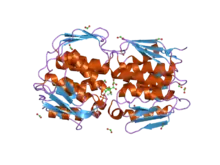
Structure et occurrence naturelle
L'EPSP synthase est un monomère dont la masse moléculaire est d'environ 46 kDa[2] - [3] - [4]. Elle est composée de deux domaines reliés par des brins peptidiques. Ces brins agissent comme une charnière, permettant le rapprochement des deux domaines. Quand un substrat se lie à l'enzyme, la liaison au ligand provoque le serrage des deux parties de l'enzyme autour du substrat dans le site actif.
Les EPSP synthases ont été classées en deux groupes en fonction de leur sensibilité au glyphosate. Les enzymes de classe I, présentes chez les plantes et chez certaines bactéries, sont inhibées à de faibles concentrations micromolaires de glyphosate, tandis que les enzymes de classe II, présentes chez d'autres bactéries, sont résistantes à l'inhibition par le glyphosate[5].
L'EPSPS intervient à la sixième étape de la voie du shikimate. Elle est absente du génome des mammifères[6] - [7], mais elle peut être présente dans le microbiote intestinal de certains animaux[8].
Inhibition par le glyphosate
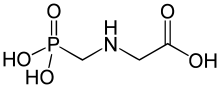
L'EPSP synthase est la cible biologique du glyphosate, un désherbant total développé par la société Monsanto et distribué sous le nom commercial de Roundup. Le glyphosate agit comme inhibiteur compétitif de l'EPSPS en se liant étroitement au complexe EPSP–3-phosphoshikimate, ce qui empêche le phosphoénolpyruvate de se lier à son tour au site actif, d'où blocage de l'enzyme, et de toute la voie du shikimate : privées d'acides aminés aromatiques, les plantes exposées au glyphosate ne peuvent plus synthétiser leurs protéines, et finissent par mourir[5] - [9].
Les équipes de Monsanto ont identifié une souche de bactéries du genre Agrobacterium, appelée CP4, qui demeure viable en présence de glyphosate, et ont pu développer des plantes génétiquement modifiées pourvues du gène CP4 EPSPS, c'est-à-dire exprimant la forme d'EPSP synthase résistante au glyphosate[5] - [10].
Notes et références
- (en) Ernest G. Jaworski, « Mode of action of N-phosphonomethylglycine. Inhibition of aromatic amino acid biosynthsis », Journal of Agricultural and Food Chemistry, vol. 20, no 6, , p. 1195-1198 (DOI 10.1021/jf60184a057, lire en ligne)
- (en) Peter Goldsbrough et al., « Gene amplification in glyphosate tolerant tobacco cells. », Plant science, vol. 72, no 1, , p. 53-62 (DOI 10.1016/0168-9452(90)90186-r, lire en ligne).
- (en) Abdel-Meguid SS, Smith WW, Bild GS, « Crystallization of 5-enolpyruvylshikimate 3-phosphate synthase from Escherichia coli », Journal of Molecular Biology, vol. 186, no 3, , p. 673 (PMID 3912512, DOI 10.1016/0022-2836(85)90140-8).
- (en) Ream JE, Steinrücken HC, Porter CA, Sikorski JA, « Purification and Properties of 5-Enolpyruvylshikimate-3-Phosphate Synthase from Dark-Grown Seedlings of Sorghum bicolor », Plant Physiology, vol. 87, no 1, , p. 232–8 (PMID 16666109, PMCID 1054731, DOI 10.1104/pp.87.1.232).
- (en) Loredano Pollegioni, Ernst Schonbrunn et Daniel Siehl, « Molecular basis of glyphosate resistance: Different approaches through protein engineering », The FEBS Journal, vol. 278, no 16, , p. 2753-2766 (PMID 21668647, PMCID 3145815, DOI 10.1111/j.1742-4658.2011.08214.x, lire en ligne)
- (en) Todd Funke, Huijong Han, Martha L. Healy-Fried, Markus Fischer et Ernst Schönbrunn, « Molecular basis for the herbicide resistance of Roundup Ready crops », Proceedings of the National Academy of Sciences of the United States of America, vol. 103, no 35, , p. 13010-13015 (PMID 16916934, PMCID 1559744, DOI 10.1073/pnas.0603638103, JSTOR 30050705, lire en ligne)
- (en) Hiroshi Maeda et Natalia Dudareva, « The Shikimate Pathway and Aromatic Amino Acid Biosynthesis in Plants », Annual Review of Plant Biology, vol. 63, , p. 73-105 (PMID 22554242, DOI 10.1146/annurev-arplant-042811-105439, lire en ligne)
- (en) Antonio L. Cerdeira et Stephen O. Duke, « The current status and environmental impacts of glyphosate-resistant crops: a review », Journal of Environmental Quality, vol. 35, no 5, , p. 1633-1658 (PMID 16899736, DOI 10.2134/jeq2005.0378, lire en ligne)
- (en) Ernst Schönbrunn, Susanne Eschenburg, Wendy A. Shuttleworth, John V. Schloss, Nikolaus Amrhein, Jeremy N. S. Evans et Wolfgang Kabsch, « Interaction of the herbicide glyphosate with its target enzyme 5-enolpyruvylshikimate 3-phosphate synthase in atomic detail », Proceedings of the National Academy of Sciences of the United States of America, vol. 98, no 4, , p. 1376-1380 (PMID 11171958, PMCID 29264, DOI 10.1073/pnas.98.4.1376, Bibcode 2001PNAS...98.1376S, lire en ligne)
- (en) Jerry M. Green et Micheal D. K. Owen, « Herbicide-resistant crops: utilities and limitations for herbicide-resistant weed management », Journal of Agricultural and Food Chemistry, vol. 59, no 11, , p. 5819-5829 (PMID 20586458, PMCID 3105486, DOI 10.1021/jf101286h, lire en ligne)
