Nucléomorphe
Un nucléomorphe est un petit noyau vestigial présent entre les paires de membranes interne et externe de certains plastes chez des eucaryotes unicellulaires de l'embranchement (phylum) des cryptophytes[1] et du sous-embranchement des chlorarachniophytes[2]. L'analyse de l'organisation génomique et de la phylogénie moléculaire de ces nucléomorphes tend à montrer qu'il s'agirait des vestiges de noyaux d'algues rouges pour les premiers et d'algues vertes pour les seconds qui auraient été englobées dans ces microorganismes par endocytose : le fait qu'ils se situent entre deux paires de membranes appuie la théorie endosymbiotique de formation des plastes — et notamment des chloroplastes — et des mitochondries par incorporation de bactéries libres dans des cellules plus grosses au sein desquelles elles seraient devenues des endosymbiotes ; il s'agirait plus précisément ici d'une endosymbiose secondaire résultant de l'incorporation d'une algue photoautotrophe par un procaryote formant le plaste dont le nucléomorphe serait le vestige du noyau, plaste lui-même incorporé par la suite dans un eucaryote hôte pour former le microorganisme actuel[3] - [4].
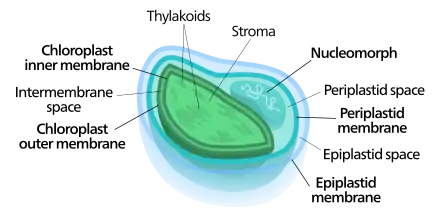
Les génomes de nucléomorphes comptent parmi les plus petits jamais séquencés. Après l'endocytose des algues rouges et vertes respectivement dans leur hôte cryptophyte et chlorarachniophyte, leur génome s'est simplifié pour atteindre une taille comparable à partir de génomes plus grands, ne conservant que trois chromosomes tandis que de nombreux gènes étaient ou bien transférés dans le génome de l'hôte, ou bien entièrement perdu[3].
Les endosymbioses successives à l'origine de ces microorganismes confèrent à ces derniers une organisation génomique particulièrement complexe, avec quatre génomes différents :
- deux génomes procaryotiques, dans les mitochondries et les plastes des algues rouges et vertes ;
- deux génomes eucaryotiques, dans le noyau de la cellule hôte et dans les nucléomorphes.
Voir aussi
Notes et références
- (en) Christopher E. Lane, Krystal van den Heuvel, Catherine Kozera, Bruce A. Curtis, Byron J. Parsons, Sharen Bowman et John M. Archibald, « Nucleomorph genome of Hemiselmis andersenii reveals complete intron loss and compaction as a driver of protein structure and function », Proceedings of the National Academy of Sciences of the United States of America, vol. 104, no 50, , p. 19908-19913 (lire en ligne) DOI 10.1073/pnas.0707419104
- (en) Paul R. Gilson, Vanessa Su, Claudio H. Slamovits, Michael E. Reith, Patrick J. Keeling et Geoffrey I. McFadden, « Complete nucleotide sequence of the chlorarachniophyte nucleomorph: Nature’s smallest nucleus », Proceedings of the National Academy of Sciences of the United States of America, vol. 103, no 25, , p. 9566-9571 (lire en ligne) DOI 10.1073/pnas.0600707103
- (en) John M. Archibald et Christopher E. Lane, « Going, Going, Not Quite Gone: Nucleomorphs as a Case Study in Nuclear Genome Reduction », Journal of Heredity, vol. 100, no 5, , p. 582-590 (lire en ligne) DOI 10.1093/jhered/esp055
- (en) Adrian Reyes-Prieto, Andreas P.M. Weber et Debashish Bhattacharya, « The Origin and Establishment of the Plastid in Algae and Plants », Annual Review of Genetics, vol. 41, , p. 147-168 (lire en ligne) DOI 10.1146/annurev.genet.41.110306.130134