Protéines alternatives
Les protéines alternatives sont des protéines traduites à partir de séquences codantes alternatives localisées dans les ARNm matures ou les ARN annotés comme non codants.
Rappels sur la biosynthèse des protéines
Les protéines sont des molécules essentielles à la vie ; elles sont synthétisées à partir de gènes codant des protéines, en deux étapes : la transcription et la traduction.
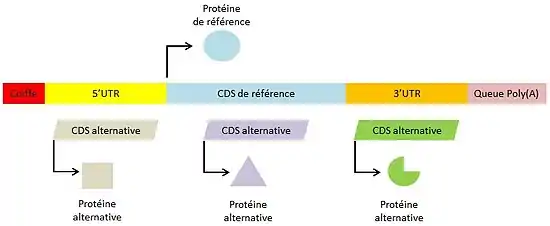
La transcription permet la synthèse d'ARN pré-messagers qui subissent un certain nombre de modifications pour donner des ARNm matures. Dans les ARNm, la région codante ou séquence codante (aussi appelée CDS pour Coding DNA Sequence), code une protéine spécifique.
Nouveauté et enjeux scientifiques et médicaux
Concernant les eucaryotes, le dogme actuel de la biologie moléculaire associe une seule séquence codante à chaque ARNm mature, et donc une seule protéine. Cette séquence codante est appelée « séquence codante de référence » et la protéine correspondante est appelée « protéine de référence ». Mieux comprendre les principes régissant la synthèse de ces protéines, les maladies qui pourraient leur être liées et leur rôle dans la biodiversité et l'évolution sont des enjeux nouveaux pour la médecine et la biologie.
Les « protéines de référence » sont répertoriées dans des bases de données telle que Uniprot.
Il existe des séquences codantes alternatives différentes des séquences codantes de référence[1]. Ces séquences codantes alternatives codent des « protéines alternatives ». Ainsi, un seul ARNm mature pourrait coder plusieurs protéines, et non pas une seule.
Le protéome, c'est-à-dire l'ensemble des protéines exprimées dans une cellule, serait donc beaucoup plus complexe que le nombre d'ARNm matures.
Localisation des « séquences codantes alternatives »
Ces séquences peuvent être localisées dans différentes régions de l'ARNm. Elles peuvent être présentes dans les trois cadres de lecture[2] des régions non traduites ou 5'-UTR (de l'anglais 5' UnTranslated Region) et 3'-UTR (de l'anglais 3' UnTranslated Region), ou bien chevaucher la séquence codante de référence dans les cadres de lecture +2 et +3. En effet, par définition, la séquence codante de référence est située sur le cadre de lecture +1.
Les séquences codantes alternatives peuvent aussi être présentes dans les ARN annotés comme "non codants".
Protéines alternatives et diversité protéique
Chez les eucaryotes, l'épissage alternatif permet la production de plusieurs ARNm matures à partir d'un seul ARN pré-messager, et les séquences codantes peuvent varier entre les différents ARNm matures. Un gène peut ainsi coder plusieurs protéines. Si les protéines produites à partir d'un gène sont différentes mais qu'elles conservent entre elles des grandes régions identiques, on les qualifie d'isoformes.
Les protéines alternatives sont entièrement différentes des protéines de référence ; elles ne sont donc pas des isoformes des protéines de référence[3] - [4].
Le nombre de séquences codantes alternatives est de 83886 chez l'espèce humaine, 82305 chez la souris, et 5019 chez la levure. La contribution des protéines alternatives à la diversité protéique pourrait donc être très importante[3].
Détection des protéines alternatives
Avant le développement et l'utilisation de la protéomique, seul un petit nombre de protéines alternatives humaines ont pu être détectées[5].
Certaines sont associées à des gènes impliqués dans des maladies neurodégénératives[3] - [4]. La protéomique permet désormais de détecter l'expression des protéines alternatives à grande échelle[1] - [6] - [7].
Médiatisation
La découverte et la détection des protéines alternatives ont d'abord fait l'objet d'articles scientifiques puis ont été mentionnées dans des articles de vulgarisation[8] ainsi que dans la presse (ex : La Tribune[9] - [10], Sherbrooke Innopole[11] - [12]). Le protéome alternatif s'est aussi mérité le titre d'une des 10 découvertes de l'année 2013 par le magazine Québec Science[13].
Notes et références
- Benoît Vanderperre, Jean-François Lucier, Cyntia Bissonnette, Julie Motard, Guillaume Tremblay, Solène Vanderperre, Maxence Wisztorski, Michel Salzet, François-Michel Boisvert et Xavier Roucou, « Direct detection of alternative open reading frames translation products in human significantly expands the proteome », PLoS One, vol. 8, no 8, (PMID 23950983, DOI 10.1371/journal.pone.0070698, lire en ligne)
- Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts et Peter Walter, « From RNA to protein », Molecular Biology of the Cell, New York, Garland Science, (lire en ligne)
- Benoît Vanderperre, Antanas B Staskevicius, Guillaume Tremblay, Marie McCoy, Megan A O'Neill, Neil R Cashman et Xavier Roucou, « An overlapping reading frame in the PRNP gene encodes a novel polypeptide distinct from the prion protein », FASEB Journal, vol. 25, no 7, , p. 2373-2386 (PMID 21478263, DOI 10.1096/fj.10-173815, lire en ligne)
- Danny Bergeron, Catherine Lapointe, Cyntia Bissonnette, Guillaume Tremblay, Julie Motard et Xavier Roucou, « An out-of-frame overlapping reading frame in the ataxin-1 coding sequence encodes a novel ataxin-1 interacting protein », Journal of Biological Chemistry, vol. 288, no 30, , p. 21824-21835 (PMID 23760502, DOI 10.1074/jbc.M113.472654, lire en ligne).
- Joel Abramowitz, Dagoberto Grenet, Mariel Birnbaumer, Hector N. Torres et Lutz Birnbaumer, « XLalphas, the extra-long form of the alpha-subunit of the Gs G protein, is significantly longer than suspected, and so is its companion Alex », Proc Natl Acad Sci U S A, vol. 101, no 22, , p. 8366-8371 (PMID 15148396, DOI 10.1073/pnas.0308758101, lire en ligne).
- Masaaki Oyama, Hiroko Kozuka-Hata, Yutaka Suzuki, Kentaro Semba, Tadashi Yamamoto et Sumio Sugano, « Diversity of translation start sites may define increased complexity of the human short ORFeome », Molecular and Cellular Proteomics, vol. 6, no 6, , p. 1000-1006 (PMID 17317662, DOI 10.1074/mcp.M600297-MCP200, lire en ligne).
- Sarah A Slavoff, Andrew J Mitchell, Adam G Schwaid, Moran N Cabili, Jiao Ma, Joshua Z Levin, Amir D Karger, Bogdan A Budnik, John L Rinn et Alan Saghatelian, « Peptidomic discovery of short open reading frame-encoded peptides in human cells », Nature Chemical Biology, vol. 9, no 1, , p. 59-64 (PMID 23160002, DOI 10.1038/nchembio.1120, lire en ligne).
- Université de Sherbrooke.
- La tribune 26 septembre 2013
- La tribune 7 janvier 2014
- Sherbrooke Innopole 10 septembre 2013
- Sherbrooke Innopole 7 janvier 2014
- Québec Science
Voir aussi
Bibliographie
- Larsen, C. J. (1998). La protéine alternative p19 ARF: un gène suppresseur de tumeur à part entière. Bulletin du cancer, 85(4), 304-0. (résumé)
- Larsen, C. J. (2001). L'imbroglio p16INK4a-p14/p19ARF: un dénouement en vue?. Bulletin du cancer, 88(11), 1055-8. (résumé)
- Pagliuzza, Amélie (2013). Étude de l’implication de la protéine F du virus de l’hépatite C dans le développement de pathologie hépatique chez deux lignées de poissons zébrés transgéniques ; Université de Montréal ; Mémoire présenté à la Faculté des études supérieures en vue de l’obtention du grade de Maitrise (M. Sc.) en microbiologie et immunologie ; PDF, 94 pages ; voir notamment p 23.