G-quadruplex
Un G-quadruplex (G4) est une structure secondaire à quatre brins que peuvent adopter les acides nucléiques (ADN ou ARN) riches en résidus de guanine. Cette structure repose sur des appariements de bases de type Hoogsteen formant un plateau de quatre résidus de guanine (G), également appelé « quartet ». L’empilement parallèle et ininterrompu d’au moins deux quartets, intercalés par un cation monovalent (sodium ou potassium) stabilisant la structure, constitue le G4. Ceci implique une structure primaire d’ADN contenant quatre paires de G pouvant se situer sur la même molécule d’ADN (G4 intramoléculaire) ou sur des molécules d’ADN différentes (G4 intermoléculaire).
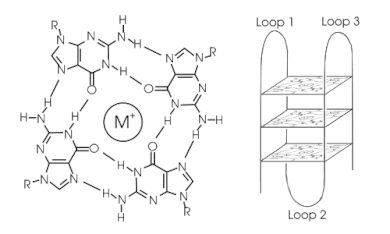
Le placement et les liaisons pour former des G-quadruplex n'ont rien d'aléatoire et ont des fonctions peu communes. Leur structure est d'autant plus stabilisée par la présence d'un cation, le plus souvent le potassium, qui se trouve dans un canal central entre chaque paire de tétrades[1]. Ils peuvent être formés d'ADN, d'ARN, de LNA et de PNA, et peuvent être intramoléculaires, bimoléculaires ou tétramoléculaires[2]. Selon la direction des brins ou des parties d'un brin qui forment les tétrades, les structures peuvent être décrites comme parallèles, antiparallèles, ou mixtes. Les structures des G-quadruplex peuvent être prédites par ordinateur à partir de motifs de séquences d'ADN ou d'ARN[3] - [4], mais leurs structures réelles peuvent être très variées au sein et entre les motifs, jusqu'à plus de 100 000 par génome. Leurs activités dans les processus génétiques de base sont un domaine de recherche actif dans les domaines des télomères, de l'épigénétique, de la génomique fonctionnelle, et de la recherche contre le cancer[5] - [6].
Découverte et caractérisation
Les G-quartets et les G-quadruplex ont été caractérisés pour la première fois en 1962 par diffraction de rayon X[7]. Elles ont été étudiées de manière détaillée in vitro, et il a été montré qu’elles se formaient dans des conditions de salinité et de pH physiologiques[8], et que de nombreuses protéines étaient capables de fixer, stabiliser, ou au contraire dérouler ces structures in vitro (pour revue[9]).
Les G-quadruplex existent dans le vivant
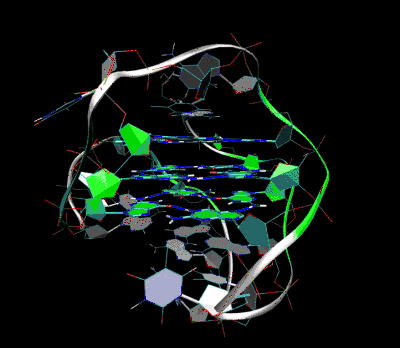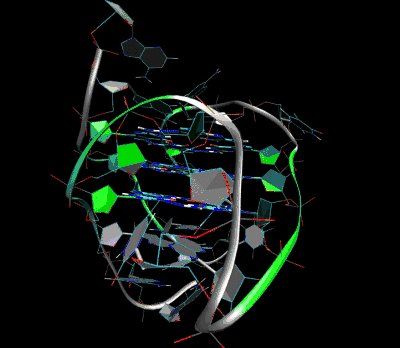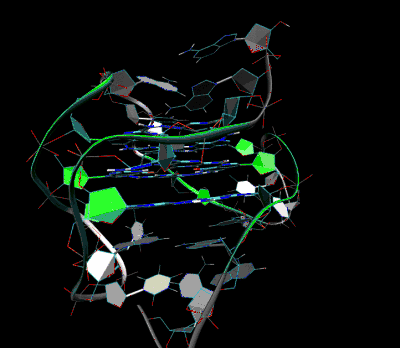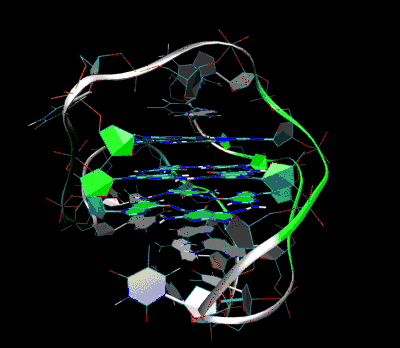
Outre les télomères, des études bioinformatiques ont révélé une forte présence de séquences potentiellement capables de former des G-quadruplex dans tous les génomes analysés jusqu’alors (plus de 270 000 chez l'homme). Ces séquences sont fortement enrichies à certains loci notamment au niveau des promoteurs, et de l'extrémité 5'-UTR des ARN messagers et cela concerne plus particulièrement certaines classes de gènes, alors que d’autres en sont dépourvus. Ceci suggère un rôle conservé de contrôle transcriptionnel, d’épissage ou de traduction pour ces structures secondaires. Outre ce rôle putatif dans le contrôle de l’expression des gènes, les G4 semblent intervenir dans de nombreux autres processus cellulaires, comme la biogenèse des ribosomes et la maturation des ARN ribosomiques, la recombinaison homologue, la régulation de la structure des télomères, et l’inhibition ou l'initialisation[10] de la réplication des ADN ribosomiques et des télomères. L’implication des G4 dans ces mécanismes où l’ADN est activement ouvert est cohérente avec l’idée que le G4 ne peut se former que lorsque l’ADN (ou l’ARN) se trouve sous forme monocaténaire.
Plusieurs études ont récemment mis en évidence, directement ou indirectement, l'existence des G-quadruplex dans les organismes vivant. Chez les ciliés par visualisation directe à l’aide d’anticorps extrêmement affins des G-quadruplex formés par les télomères de Stylonychia lemnae[11] ainsi que le rôle de deux protéines télomériques (TEBPα et ß) dans la formation de cette structure démontrant un rôle important des G-quadruplex dans le métabolisme des télomères[12]. Chez l'homme, l'existence des G-quadruplex a été démontrée à l'aide de ligands liant spécifiquement les G-quadruplex télomériques[13] - [14]. Chez la bactérie, la formation de G-quadruplex au cours de la transcription a été mis en évidence par microscopie électronique chez E. coli[15]. Un champ de l'étude des G-quadruplex réside également dans leur visualisation en direct dans une cellule vivante, notamment via des méthodes de spectroscopie[16].
Notes et références
- Eric Largy, Jean-Louis Mergny et Valérie Gabelica, « The Alkali Metal Ions: Their Role in Life », Metal Ions in Life Sciences, Springer, vol. 16, , p. 203–258 (PMID 26860303, DOI 10.1007/978-3-319-21756-7_7, lire en ligne)
- Bochman ML, Paeschke K, Zakian VA, « DNA secondary structures: stability and function of G-quadruplex structures », Nature Reviews. Genetics, vol. 13, no 11, , p. 770–80 (PMID 23032257, PMCID 3725559, DOI 10.1038/nrg3296)
- Yadav VK, Abraham JK, Mani P, Kulshrestha R, Chowdhury S, « QuadBase: Genome-Wide Database of G4 DNA--occurrence and Conservation in Human, Chimpanzee, Mouse and Rat Promoters and 146 Microbes », Nucleic Acids Research, vol. 36, no Database, , D381‐D385 (PMID 17962308, PMCID 2238983, DOI 10.1093/nar/gkm781)
- Dhapola P, Chowdhury S, « QuadBase2: Web Server for Multiplexed Guanine Quadruplex Mining and Visualization », Nucleic Acids Research, vol. 44, no W1, , W277‐W283 (PMID 27185890, PMCID 4987949, DOI 10.1093/nar/gkw425)
- Modèle:CArticle
- Borman S, « Promoter quadruplexes folded DNA structures in gene-activation sites may be useful cancer drug targets », Chemical and Engineering News, vol. 87, no 44, , p. 28–30 (DOI 10.1021/cen-v087n044.p028)
- GELLERT M, LIPSETT MN, DAVIES DR. Helix formation by guanylic acid.Proc Natl Acad Sci U S A. 1962 Dec 15;48:2013-8
- Parkinson GN, Lee MP, Neidle S. Crystal structure of parallel quadruplexes from human telomeric DNA. Nature. 2002 Jun 20;417(6891):876-80. Epub 2002 May 26.
- In vivo veritas: Using yeast to probe the biological functions of G-quadruplexes. Johnson JE, Smith JS, Kozak ML, Johnson FB. Biochimie. 2008 Feb 21
- (en) Anne-Laure Valton et al., « G4 motifs affect origin positioning and efficiency in two vertebrate replicators », EMBO J., vol. 33(7), , p. 732-746
- Schaffitzel C, Berger I, Postberg J, Hanes J, Lipps HJ, Plückthun A. In vitro generated antibodies specific for telomeric guanine-quadruplex DNA react with Stylonychia lemnae macronuclei. Proc Natl Acad Sci U S A. 2001 Jul 17;98(15):8572-7
- Paeschke K, Simonsson T, Postberg J, Rhodes D, Lipps HJ. Telomere end-binding proteins control the formation of G-quadruplex DNA structures in vivo. Nat Struct Mol Biol. 2005 Oct;12(10):847-54
- Gomez D, Paterski R, Lemarteleur T, Shin-ya K, Mergny J.L. and Riou J.F. Interaction of telomestatin with the telomeric single-strand overhang.J Biol Chem. 2004 Oct 1;279(40):41487-94.
- (en) Giulia Biffi et al., « Quantitative visualization of DNA G-quadruplex structures in human cells », Nature Chemistry, vol. 5, , p. 182-186
- Duquette ML, Handa P, Vincent JA, Taylor AF, Maizels N.Intracellular transcription of G-rich DNAs induces formation of G-loops, novel structures containing G4 DNA.Genes Dev. 2004 Jul 1;18(13):1618-29.
- (en) Aurélien Laguerre et al., « Direct visualization of both DNA and RNA quadruplexes in human cells via an uncommon spectroscopic method », Scientific Reports, vol. 6 : 32141,
Bibliographie
- Quadruplex Nucleic Acids (ISBN 0-85404-374-8) Neidle & Balasubramanian (Eds.) 2006
Communiqués de presse académiques
Sites internet dédiés aux G-quadruplex
- Quadruplex.org - a website to serve the quadruplex community
- Quadbase - downloadable data on predicted G-quadruplexes
- Greglist - a database listing potential G-quadruplex regulated genes
- Database on Quadruplex information: QuadBase from IGIB
- GRSDB- a database of G-quadruplexes near RNA processing sites.
- GRS_UTRdb- a database of G-quadruplexes in the UTRs.
- G-quadruplex Resource Site