ADN triplex
L'ADN triplex, ou ADN triple brin, correspond à l'ADN H, structure dans laquelle trois brins d'ADN s'enroulent l'un autour de l'autre pour former une triple hélice. Plus précisément, le troisième brin d'ADN s'insère dans le grand sillon de l'ADN B, bicaténaire et uni par liaisons hydrogène de type Watson–Crick, à travers un appariement Hoogsteen direct ou inverse.
.png.webp)
Structures et typologies
Une paire de bases Watson–Crick telle que thymine–adénine, notée T-A, peut former un appariement Hoogsteen avec une autre thymine, donnant un triplet de bases noté T-A*T[2]. En conditions acides, une cytosine protonée, notée C+, peut se lier à une paire cytosine–guanine, notée C-G, l'ensemble donnant un triplet C-G*C+. Les triplets de bases T-A*T et C-G*C+ sont les plus stables, tandis que les triplets de bases T-A*G et C-G*G sont les moins stables[3].
On connaît deux types d'ADN triplex : les formations intermoléculaires et les formations intramoléculaires. Les premières font intervenir un ADN bicaténaire et un troisième brin d'une autre molécule d'ADN, ce troisième brin pouvant provenir d'un chromosome voisin ou d'un fragment de TFO, ou Triplex-Forming Oligonucleotides en anglais. Les ADN triplex intramoléculaires, quant à eux, se forment à partir d'un ADN bicaténaire présentant des brins d'homopurine et d'homopyrimidine ayant une symétrie de séquence en miroir[4]. Le degré de surenroulement de l'ADN influence sa capacité à former des triplex[5].
Il existe deux types de triplex intramoléculaires l'ADN H et l'ADN H*. L'ADN H est stabilisé par des conditions acides et la présence de cations divalents tels que ceux de magnésium Mg2+. Dans cette configuration, le brin d'homopyrimidine de l'ADN bicaténaire se replie pour se lier au brin de purine de manière parallèle. Les triplets de bases qui stabilisent cette configuration sont T-A*T et C-G*C+. La cytosine de ce triplet doit être protonée afin de pouvoir établir la liaison hydrogène stabilisant cette configuration par appariement Hoogsteen, raison pour laquelle cela nécessite des conditions acides[6]. L'ADN H* se forme à pH neutre et en présence de cations divalents[5]. Il s'agit d'un repliement intramoléculaire permettant la liaison antiparallèle de brins homopurine et purine. Cette configuration est stabilisée par les triplets T-A*A et C-G*G[4] - [6].
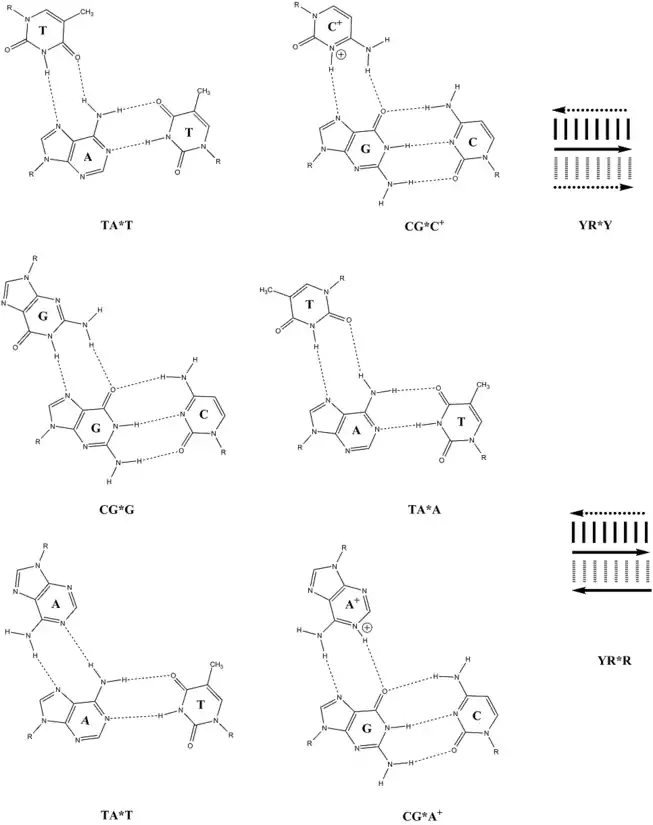 Appariements Hoogsteen d'ADN triplex. Sont représentés de gauche à droite et de haut en bas les triplets TA*T, CG*C+, CG*G, TA*T, TA*A et CG*A+.
Appariements Hoogsteen d'ADN triplex. Sont représentés de gauche à droite et de haut en bas les triplets TA*T, CG*C+, CG*G, TA*T, TA*A et CG*A+.
Fonction
La formation d'ADN triplex a été reliée à la régulation de plusieurs gènes. Par exemple, le gène régulateur (en) c-myc (MYC (en)) a été intensivement étudié en y introduisant des mutations pour y observer l'effet de l'ADN triplex par rapport à celui de l'ADN bicaténaire dans la régulation. Un élément promoteur, dit NSE pour Nuclease-Sensitive Element, peut former des triplexes intramoléculaires en tandem de type ADN H et présente des séquences (ACCCTCCCC)4 répétées en tandem.
Un NSE muté a été étudié par rapport à l'activité de transcription et à sa capacité à former des triplex intramoléculaires et intermoléculaires. L'activité de transcription de ces NSE mutants peut être prédite par la capacité de l'élément à former de l'ADN H et non en fonction du nombre ou de la position des paires de bases altérées par mutation. L'ADN lui-même intervient donc peut-être de façon active dans la transcription du gène c-myc[7].
Notes et références
- (en) Juan Luis Asensio, Tom Brown et Andrew N. Lane, « Solution conformation of a parallel DNA triple helix with 5′ and 3′ triplex–duplex junctions », Structure, vol. 7, no 1, , p. 1-11 (PMID 10368268, DOI 10.1016/S0969-2126(99)80004-5, lire en ligne)
- (en) Sangkee Rhee, Zong-jin Han, Keliang Liu, H. Todd Miles et David R. Davies, « Structure of a Triple Helical DNA with a Triplex−Duplex Junction », Biochemistry, vol. 38, no 51, , p. 16810-16815 (PMID 10606513, DOI 10.1021/bi991811m, lire en ligne)
- (en) Jean-Louis Mergny, Jian Sheng Sun, Michel Rougee, Thérèse Montenay-Garestier, Francisca Barcelo, Jacques Chomilier et Claude Hélène, « Sequence specificity in triple helix formation: experimental and theoretical studies of the effect of mismatches on triplex stability », Biochemistry, vol. 30, no 40, , p. 9791-9798 (PMID 1911764, DOI 10.1021/bi00104a031, lire en ligne)
- (en) David W. Ussery et Richard R. Sinden, « Environmental influences on the in vivo level of intramolecular triplex DNA in Escherichia coli », Biochemistry, vol. 32, no 24, , p. 6206-6213 (PMID 8512930, DOI 10.1021/bi00075a013, lire en ligne)
- (en) A. Dayn, G. M. Samadashwily et S. M. Mirkin, « Intramolecular DNA triplexes: unusual sequence requirements and influence on DNA polymerization », Proceedings of the National Academy of Sciences of the United States of America, vol. 89, no 23, , p. 11406-11410 (PMID 1454828, PMCID 50559, DOI 10.1073/pnas.89.23.11406, Bibcode 1992PNAS...8911406D, lire en ligne)
- (en) V. I. Lyamichev, S. M. Mirkin et M. D. Frank-Kamenetskii, « Structures of Homopurine-homopyrimidine Tract in Superhelical DNA », Journal of Biomolecular Structure and Dynamics, vol. 3, no 4, , p. 667-669 (PMID 3271043, DOI 10.1080/07391102.1986.10508454, lire en ligne)
- (en) A. B. Firulli, D. C. Maibenco et A. J. Kinniburgh, « Triplex Forming Ability of a c-myc Promoter Element Predicts Promoter Strength », Archives of Biochemistry and Biophysics, vol. 310, no 1, , p. 236-242 (PMID 8161210, DOI 10.1006/abbi.1994.1162, lire en ligne)