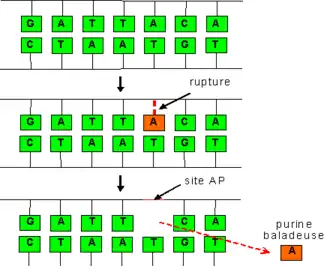
Dépurination et dépyrimidination
La dépurination est une altération de l'ADN où se rompt le lien entre une purine (adénine ou guanine) et le désoxyribose auquel elle est attachée. La dépyrimidination, beaucoup plus rare, correspond au même phénomène, mais pour la rupture du lien entre un désoxyribose et une pyrimidine (thymine ou cytosine). Ces deux altérations sont donc très similaires (on les réunit parfois sous le terme de déglycosylation), et ont exactement les mêmes effets: dans les deux cas, la base libérée laisse derrière elle une anomalie de la séquence génétique, en l’occurrence un site vacant, dit site AP (site apurinique/apyrimidique) ou site abasique.
Origine
Dans l'ADN, chaque base est attachée à un désoxyribose par une liaison N-C, dite N-glycosidique. Ces liens sont lentement hydrolysés, y compris en conditions physiologiques, au rythme de 3,0 × 10−11 s−1 pour les purines et 1,5 × 10−12 s−1 pour les pyrimidines[1] - [2].
Ces rythmes d'hydrolyse spontanée sont certes faibles, mais ils ne sont pas négligeables si on prend en compte la taille des génomes (106 à 1011 nucléotides, suivant les espèces). Dans le cas du génome humain (environ 3,4 milliards de paires de bases), cela représente environ 9 000 à 10 000 dépurinations spontanées par jour et par cellule (les dépyrimidinations se produisant à un rythme 20 fois plus lent)[3]. Cela en fait la première cause endogène d'altération de l'ADN, loin devant la déamination (100-500/jour/cellule)[4].
Si les dépurinations et dépyrimidinations spontanées sont des altérations indésirables, leurs analogues provoqués font par contre partie de la vie normale des cellules : lorsque des bases nucléotidiques sont endommagées, elles sont autant que possible réparées grâce au système de réparation par excision de base (BER, base excision repair). La première étape de ce processus de réparation est l'extraction des bases endommagées (déglycosilation) par des enzymes spécialisées, les glycosylases, ce qui permet de les remplacer ensuite par des bases intactes[5].
Bibliographie
- Réplication et réparation de l'ADN, Cours du Master Recherche "Biologie structurale et bio-informatique", Université Louis Pasteur Strasbourg, Département Intégrité du Génome, UMR CNRS-7175 ;
- Maintenance et variations du matériel génétique, Pr. J. Lunardi, Cours de Biochimie et Biologie Moléculaire, Université Joseph Fourier de Grenoble ;
- Altération et réparation de l’ADN, Cours de Licence Sciences et Technologie, Module de Biologie moléculaire 2, Université Paris 12 Val-de-Marne
Notes et références
- Gates 2009, An Overview of Chemical Processes That Damage Cellular DNA: Spontaneous Hydrolysis, Alkylation, and Reactions with Radicals, Chemical Research in Toxicology 22:1747–1760
- EC Friedberg, "DNA Repair and Mutagenesis", John Wiley & Sons, 1995
- Nakamura 1999, Endogenous apurinic/apyrimidinic sites in genomic DNA of mammalian tissues, Cancer Research 59:2522-2526
- (en) Committee to Assess Health Risks from Exposure to Low Levels of Ionizing Radiation, National Research Council, Health Risks from Exposure to Low Levels of Ionizing Radiation : BEIR VII – Phase 2, Washington, DC, National Academy Press, (ISBN 0-309-53040-7, présentation en ligne, lire en ligne)
- Wyatt 1999, 3-Methyladenine DNA glycosylases: structure, function, and biological importance, BioEssays 21:668–676