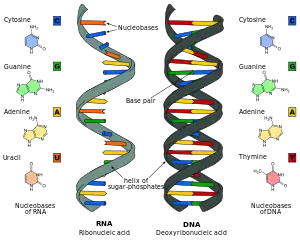
Analogue d'acide nucléique
Les analogues d'acides nucléiques sont des composés qui sont structurellement similaires aux ARN et ADN présents dans la nature. Ils ont des utilisations pour la recherche en médecine et en biologie moléculaire. Les nucléotides, qui constituent les acides nucléiques, sont composées de trois parties : un squelette phosphate, un sucre pentose (ribose ou désoxyribose) et l'une des quatre bases nucléiques. Dans un analogue d'acide nucléique, n'importe laquelle de ces parties peut être modifiée. Cela résulte typiquement en une modification de l'appariement ou de l'empilement des bases (il existe ainsi des bases universelles pouvant s'apparier avec n'importe quelle autre base). Les analogues permettent aussi des modifications de la structure de la chaîne, en remplaçant le squelette phosphate. Un squelette de PNA permet par exemple d'obtenir une structure en triple hélice[1].
Utilisations
En médecine, plusieurs analogues de nucléosides sont employés comme antiviraux ou anticancéreux. Ils sont généralement administrés sous forme de nucléosides et transformés en nucléotides à l'intérieur de la cellule, car les nucléotides qui sont chargés traversent difficilement les membranes cellulaires.
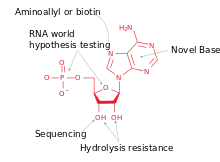
Les analogues d'acides nucléiques ont de nombreux emplois en biologie moléculaire :
- Détection de séquences particulières
- Fabrication d'ARN résistants à l'hydrolyse
- Séquençage de l'ADN grâce à des didésoxynucléotides
- Investigation des mécanismes enzymatiques, en se servant des analogues comme inhibiteurs
- Investigation des propriétés structurales des acides nucléiques
- Création de systèmes alternatif en biologie de synthèse
- Recherche sur les origines de la vie…
Analogues où le squelette est modifié
Analogues d'ARN résistants à l'hydrolyse
Les riboses de la molécule d'ARN sont très facilement hydrolysables, ce qui la rend difficile à synthétiser et utiliser de façon fiable. Des dérivés de ribose peuvent donc être utilisés. Les analogues les plus communs sont l'ARN substitué en 2'O-méthyl, l'acide nucléique verrouillé (Locked Nucleic Acid ou LNA), le morpholino[2] - [3] et les acides nucléiques peptidiques (Peptide nucleic acid ou PNA). Bien que ces oligonucléotides aient des squelettes différents, ils sont toujours capables de s'hybrider à l'ARN ou l'ADN, mais ils ne sont pas sensibles à l'hydrolyse par les nucléases. Il n'existe pas de méthode enzymatique pour les fabriquer, il est nécessaire de recourir à des méthodes de synthèse telles que la stratégie de la phosphoramidite ou (pour le PNA) la synthèse peptidique.
Rôle hypothétique dans l'apparition de la vie
Il est intéressant de noter que de tels analogues d'acides nucléiques pourraient avoir joué un rôle dans l'apparition de la vie, l'ARN connu actuellement étant trop complexe pour avoir été le premier acide nucléique. Des analogues plus simples comme le GNA, le TNA et le PNA pourraient être des candidats plus vraisemblables.
Analogues de bases
Bases présentes naturellement
On distingue les bases pyrimidines et purines. Elles sont capables de s'apparier grâce à deux ou trois liaisons hydrogène entre une fonction cétone) et une fonction amine ; elles peuvent aussi s'empiler par l'action de leurs cycles aromatiques (interaction de π-stacking).
Bases marquées par un fluorophore
Des fluorophores comme la rhodamine et la fluorescéine sont liés au cycle voisin du sucre (en para) par un bras flexible, de sorte qu'il soit positionné dans le sillon majeur de la double hélice. En général, on synthétise d'abord la séquence avec des nucléotides ayant seulement le bras, puis l'on couple ces bras à des fluorophores (marquage indirect). L'enzyme utilisée, la Taq polymérase, n'est en effet pas capable d'incorporer directement les fluorophores en raison de leur taille.
Bases constituant elles-mêmes un fluorophore
La plus fréquemment utilisée est la 2-aminopurine, qui présente la propriété intéressante d'être fortement fluorescente quand elle est libre en solution, mais considérablement moins lorsqu'elle est incorporée à un brin d'acide nucléique. La variation de l'intensité dépend de la séquence, mais elle est de l'ordre de cent fois[4].
Il existe d'autres bases du même type : 3-MI, 6-MI, 6-MAP[5], pyrrolo-dC[6] et ses dérivés[7], bases furaniques modifiées [8] et bien d'autres[9] - [10] - [11] - [12].
La fluorescence de ces bases est très dépendante de leur environnement direct. Ceci est utile pour observer les interactions entre protéines et ADN, et les transferts d'électron au sein de l'ADN. D'autres bases comme la 1,3-Diaza-2-oxophenothiazine, tC, ont un rendement de fluorescence presque indépendant des bases environnantes[13] - [14]. Par ailleurs, elles ont peu d'influence sur les propriétés physiques et chimiques de l'ADN, ce qui en fait des outils appréciables pour les analyses FRET. Le tCnitro, une base jouant le rôle d'accepteur de fluorescence, a été développé à cet effet[15]. Le couple tCO (donneur)/ tCnitro (accepteur) est ainsi la première paire pour FRET utilisant des bases nucléiques.
Modification de l'appariement des bases
| Paires de bases naturelles | |
|---|---|
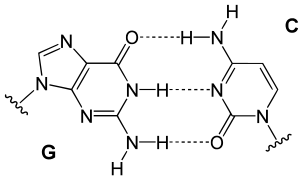 |
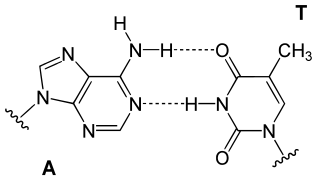 |
| Une paire GC, formant 3 liaisons hydrogène | Une paire AT, formant 2 liaisons hydrogène |
La diaminopurine (DAP) est présente naturellement chez le Cyanophage S-2L. Utilisée à la place de l'adénine, elle forme trois liaisons hydrogène avec la thymine, ce qui rend le brin plus stable et rend les interactions AT et GC aussi fortes l'une que l'autre[16]. Cela peut être utilisé pour analyser les interactions moléculaires mettant en jeu cette différence de force.
D'autres bases comme l'isoguanine et l'isocytosine (qui ont des groupements amine et cétone inversés par rapport à leurs homologues) peuvent être incorporées lors d'une amplification par PCR, même en présence des quatre bases canoniques[17].
| Paires de bases absentes dans la nature | ||
|---|---|---|
 |
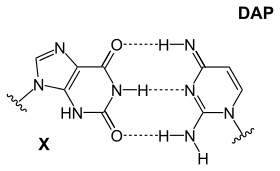 |
 |
| DAP-T | X-DAP | iG-iC |
Il est également possible de créer des bases qui s'apparient par interactions hydrophobes. On a par exemple le 2,4-difluorotoluène, un analogue de thymine, et le 4-méthylbenzimidazole, analogue d'adénine[18], ou encore l'isoquinoline et la pyrrolo[2,3-b]pyridine[19].
Bases universelles
Ces bases sont capables de s'apparier sans discrimination avec n'importe quelle autre base. Généralement, elles diminuent considérablement la température de fusion de la séquence. Parmi elles, on trouve les dérivés de la 2'-déoxyinosine (désoxynucléotide d'hypoxanthine), les analogues de nitroazole, et les bases aromatiques hydrophobes qui se lient sans liaison hydrogène. Elles n'ont cependant pas montré d'intérêt particulier dans la constitution d'amorces pour la polymérisation.
Bases étendues et xDNA
Il existe un jeu de bases nucléiques étendu, appelé xDNA, qui comporte en plus des quatre bases canoniques, quatre bases étendues qui sont en fait les bases canoniques auxquelles on a ajouté un cycle benzène. Une base classique s'apparie avec une base étendue, de sorte que la longueur totale de la paire de base soit constante. De cela, il résulte quatre combinaisons possibles : xA-T, xT-A, xC-G et xC-C. Ce système peut avoir une utilité pour des cas particuliers de recherche scientifique. Le yDNA est un système similaire, mais le benzène sert cette fois-ci à élargir les bases[20].
| Paires de bases artificielles ayant des propriétés particulières | |||
|---|---|---|---|
 |
 |
 |
|
| Base F-Z : Pas de liaison hydrogène | Base S-Pa: Trois liaisons hydrogène | Base xA-T : Liaison similaire à A-T |
Appariement avec un ion métallique
Les liaisons de type Watson-Crick peuvent être remplacées par un ion métallique central. Les nucléosides jouent alors le rôle de ligands. Différentes configurations bidentates permettent d'apparier deux nucléosides autour d'un atome métallique central : en tétraèdre, en dodécaèdre, et carrée plane. La formation d'une telle paire de base peut résulter de l'appariement non-canonique des bases habituelles en présence d'un ion métallique, ou du remplacement des atomes d'hydrogène impliqués dans les liaisons de Watson et Crick par des atomes métalliques[21].
L'introduction d'ions métalliques dans un duplex d'ADN peut lui conférer des propriétés de perméabilité magnétique[22] - [23] ainsi qu'une stabilité augmentée[24].
Un exemple bien documenté est la formation de la paire de bases T-Hg-T, qui met en jeu deux thymines reliées par un ion Hg<2+>[25] - [26]. Deux thymines ne peuvent pas s'apparier entre elles dans la nature, c'est donc bien un exemple d'appariement impossible stabilisé par l'action d'un métal. Il est également possible de former des paires A-Zn-T et G-Zn-C à pH élevé. Les ions Co²⁺ et Ni²⁺ peuvent aussi former ce type de complexes. Ici, le cation divalent est coordiné en complexe avec les bases, même si la configuration exacte n'est pas parfaitement connue[27].
De nombreuses nucléobases artificielles ont été développées pour optimiser la complexation avec des métaux. Par exemple, un nucléoside modifié avec un pyridine-2,6-dicarboxylate se complexe fortement et spécifiquement avec les ions Cu²⁺, sans se lier significativement avec les autres cations (cette affinité peut s'expliquer par la formation d'un complexe tridendate)[28]. Ce type de technologie pourrait constituer, dans le futur, un espoir pour la fabrication de câbles métalliques nanoscopiques capables de s'assembler eux-mêmes[29].
Système orthogonal
Selon certains chercheurs[30], il serait possible d'implémenter dans des cellules un système orthogonal complètement indépendant du matériel génétique déjà présent. Ceci permettrait d'assurer la sécurité d'utilisation des cellules modifiées[31].
Plusieurs approches sont envisagées :
- De nouveaux systèmes de bases et de squelettes comme ceux présentés dans cet article.
- La création d'une polymérase capable de synthétiser un (acide xénonucléique) (AXN). L'ARN polymérase T7 peut être utilisée comme modèle de départ[32].
- Des ribosomes modifiés, dont le site anti-séquence Shine-Dalgarno est altérée pour permettre la translation des ARNm orthogonaux seulement[33]
- De nouveaux ARNt correspondant à des acides aminés non-naturels.
Notes et références
- Petersson B et al. Crystal structure of a partly self-complementary peptide nucleic acid (PNA) oligomer showing a duplex-triplex network. J Am Chem Soc. 2005 Feb 9;127(5):1424–30.
- Summerton J and Weller D. Morpholino Antisense Oligomers: Design, Preparation and Properties. Antisense & Nucleic Acid Drug Development 1997; 7:187-195.
- Summerton J. Morpholino Antisense Oligomers: The Case for an RNase-H Independent Structural Type. Biochimica et Biophysica Acta 1999; 1489: 141-158.
- Ward et al. Fluorescence Studies of Nucleotides and Polynucleotides I. Formycin 2-Aminopurine Riboside 2,6-Diaminopurine Riboside and Their Derivatives. J. Biol. Chem. 1969; 244: 1228–37.
- Hawkins Fluorescent pteridine nucleoside analogs - A window on DNA interactions. Cell Biochem. Biophys. 2001; 34: 257–81.
- Berry et al. Pyrrolo-dC and pyrrolo-C: fluorescent analogs of cytidine and 2 '-deoxycytidine for the study of oligonucleotides. Tetrahedron Lett. 2004; 45: 2457–61.
- Wojciechowski et al. Fluorescence and hybridization properties of peptide nucleic acid containing a substituted phenylpyrrolo-cytosine designed to engage guanine with an additional H-bond. J. Am. Chem. Soc. 2008; 130: 12574-12575.
- Greco et al. Simple fluorescent pyrimidine analogues detect the presence of DNA abasic sites. J. Am. Chem. Soc. 2005; 127: 10784–85.
- Rist et al. Fluorescent nucleotide base analogs as probes of nucleic acid structure, dynamics and interactions. Curr. Org. Chem. 2002; 6: 775–93.
- Wilson et al. Fluorescent DNA base replacements: reporters and sensors for biological systems. Org, & Biomol. Chem. 2006; 4: 4265–74.
- Wilhelmsson Fluorescent nucleic acid base analogues. Q. Rev. Biophys. 2010; 43(2): 159-183.
- Sinkeldam et al. Fluorescent analogs of Biomolecular Building Blocks: Design, properties and applications. Chem. Rev. 2010; 110(5): 2579-2619.
- Wilhelmsson et al. A highly fluorescent DNA base analogue that forms Watson-Crick base pairs with guanine. J. Am. Chem. Soc. 2001; 123: 2434–35.
- Sandin et al. Fluorescent properties of DNA base analogue tC upon incorporation into DNA - negligible influence of neighbouring bases on fluorescence quantum yield. Nucleic Acids Res. 2005; 33: 5019–25.
- Börjesson et al. Nucleic acid base analog FRET-pair facilitating detailed structural measurements in nucleic acid containing systems. J. Am. Chem. Soc. 2009; 131: 4288–93.
- Kirnos MD, Khudyakov IY, Alexandrushkina NI, Vanyushin BF. 2-aminoadenine is an adenine substituting for a base in S-2L cyanophage DNA. Nature. 1977 Nov 24;270(5635):369–70.
- Johnson SC et al. A third base pair for the polymerase chain reaction: inserting isoC and isoG. Nucleic Acids Res. 2004 Mar 29;32(6):1937–41.
- Taniguchi Y, Kool ET. Nonpolar isosteres of damaged DNA bases: effective mimicry of mutagenic properties of 8-oxopurines. J Am Chem Soc. 2007 Jul 18;129(28):8836–44. Epub 2007 Jun 26.
- G. T. Hwang, F. E. Romesberg, J. Am. Chem. Soc. 2008, 130, 14872
- Liu H et al. (ET Kool Lab). A four-base paired genetic helix with expanded size. Science. 2003 Oct 31;302(5646):868–71
- S. D. Wettig, D. O. Wood.m J.S. Lee.J. Inorg. Biochem. 2003, 94, 94–99
- H. Zhang, A. Calzolari, R. Di Felice. J. Phys. Chem. B 2005, 109, 15345–15348.
- P. Aich, R. J. S. Skinner, S. D. Wettig, R. P. Steer, J. S. Lee.Biomol. Struct. Dyn. 2002, 20, 93–98.
- G. H. Clever, K. Polborn, T. Carell, Angew. Chem. Int. Ed.2005, 117, 7370–7374
- E. Buncel, C. Boone, H. Joly, R. Kumar, A. R. J. Norris, Inorg. Biochem.198525, 61–73
- A. Ono, H. Togashi, Angew. Chem.2004, 43, 4300–4302
- E. Meggers, P. L. Holland, W. B. Tolman, F. E. Romesberg, P. G. Schultz. J. Am. Chem. Soc.2000122, 10714–10715
- J. S. Lee, R. J. S. Skinner, L. J. P. Latimer, R. S. Reid. Biochem. Cell Biol.199371, 162–168
- K. Tanaka, A. Tengeiji, T. Kato, N. Toyama, M. Shionoya. Science2003, 299, 1212–1213
- Schmidt M. Xenobiology: a new form of life as the ultimate biosafety tool Bioessays Vol 32(4):322-331
- Herdewijn P, Marlière P.Toward safe genetically modified organisms through the chemical diversification of nucleic acids. Chem Biodivers. 2009 Jun;6(6):791–808.
- A. Shinkai, P. H. Patel, L. A. Loeb, J. Biol. Chem. 2001, 276, 18836
- Rackham O, Chin JW. A network of orthogonal ribosome x mRNA pairs.Nat Chem Biol. 2005 Aug;1(3):159-66. Epub 2005 Jul 17.
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Nucleic acid analogue » (voir la liste des auteurs).