Topoisomère
Des topoisomères ou isomères topologiques sont des formes distinctes de molécules de structures chimiques identiques mais qui diffèrent par leur forme topologique. Ce terme est principalement utilisé dans le cas de l'ADN, pour distinguer des ADN circulaires de séquence identique, mais qui diffèrent par le nombre d'enlacements, c'est-à-dire le nombre de tours qu'un des deux brins de la double hélice fait autour de l'autre[1].
Nombre d'enlacements
La conformation la plus fréquente de l'ADN, le duplex en forme B, comporte environ 10,5 paires de bases par tour d'hélice. Lorsque le nombre d'enlacements pour un ADN circulaire (plasmide, chromosome bactérien) correspond à cette valeur, il est dit relâché. Par exemple, pour un plasmide long de 2100 paires de bases, cela correspond à 200 tours d'hélices (2100 / 10,5), soit un nombre d'enlacement de 200.
Ce nombre d'enlacements est une constante topologique, liée au fait que chacun des deux brins d'ADN forme un cercle complet, fermé de manière covalente sur lui-même.
Différents états de topoisomères
Les topoisomères sont des formes d'ADN circulaires de séquence identique, qui diffèrent uniquement par le nombre d'enlacements.
État relâché
L'état relâché est celui ou l'ADN est dans sa configuration canonique avec ~10,5 paires de bases par tour d'hélice. Dans cette conformation, la contrainte sur la double hélice est minimale, c'est la configuration la plus stable. Cette valeur peut varier en fonction de la présence de différentes protéines associées à l'ADN ou de la présence d'agents intercalants.
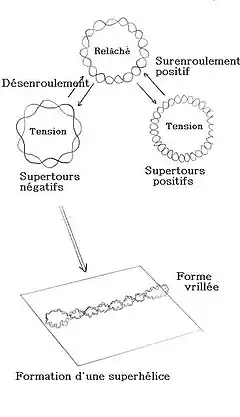
États surenroulés
Lorsque le nombre d'enlacement est supérieur ou inférieur à la valeur de l'état relâché, on obtient deux types de topoisomères :
- Si le nombre de tours d'hélice est plus grand que la valeur standard correspondant à l'état relâché, l'ADN est dit surenroulé positivement.
- À l'inverse, il peut aussi avoir un nombre de tour d'hélices plus faible, il est alors désenroulé ou surenroulé négativement.
Pour relâcher la contrainte liée au surenroulement et ramener localement le nombre de paires de bases par tour aux alentours de la valeur standard ~10,5, l'ADN adopte alors une forme vrillée, en formant une superhélice (hélice formée par le double-brin sur lui-même, en plus de l'hélice formée par un brin autour de l'autre). Lorsque le surenroulement est positif, il forme une superhélice gauche et lorsque le surenroulement est négatif, il forme une superhélice droite.

Le surenroulement négatif est le plus rencontré dans la nature car il permet une réduction de la taille globale de la molécule d'ADN. Le désenroulement le plus courant est de un supertour pour 200 paires de bases. En effet, le surenroulement négatif provoque une contrainte qui induit un enroulement de l'axe de l'hélice d'ADN, qui se vrille alors. Il présente alors deux avantages[1] :
- la formation de la superhélice rend plus compacte la molécule, notamment dans les bactéries ou les mitochondries où l'ADN est circulaire ;
- il permet d'absorber une partie de la contrainte générée par le déroulement local nécessaire à la réplication et à la transcription dans un ADN circulaire. En effet, lorsqu'une ADN polymérase ou une ARN polymérase utilise un ADN circulaire comme matrice, ces enzymes doivent créer une zone simple brin en déroulant la double-hélice.
Interconversion des topoisomères
Pour modifier la topologie de l'ADN et permettre l'interconversion des différents topoisomères, il est nécessaire de couper puis de suturer au moins un des deux brins. Les enzymes qui permettent de réaliser ces conversions sont des topoisomérases.
Les topoisomérases I relâchent le surenroulement de manière passive, en clivant puis en suturant un des deux brins
Le surenroulement négatif est créé de manière active chez les bactéries par une enzyme appelée ADN gyrase qui appartient à la famille des topoisomérases II.
Notes et références
- Xavier Coumoul, Frédéric Dardel et Étienne Blanc, Mémo visuel de biochimie : l'essentiel en fiches, Paris, Dunod, , 226 p. (ISBN 978-2-10-074226-4), p. 115-116