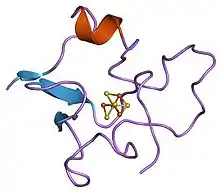
Protéine fer-soufre à haut potentiel
Les protéines fer-soufre à haut potentiel (ou HiPIP pour (en) High potential iron-sulfur proteins) sont une classe de ferrédoxines de formule 4Fe-4S à haut potentiel redox[2] qui interviennent dans le transport anaérobie d'électrons et qui sont produites par les bactéries photosynthétiques et Paracoccus denitrificans. Ces petites protéines présentent des variations importantes dans leurs séquences, leurs tailles (63 à 85 acides aminés) et dans leurs potentiels d'oxydoréduction. Comme illustré par le schéma suivant, le cluster fer-soufre est lié par quatre cystéines résiduelles conservées.
Amas [Fe4S4]
Les amas [Fe4S4] sont des cofacteurs abondants de métalloprotéines[3]. Ils participent aux séquences de transfert d'électrons. La structure centrale de l'amas [Fe4S4] est un cube avec des sommets Fe et S alternés. Ces clusters existent dans deux états d'oxydation avec un petit changement structurel. Deux familles d'amas de [Fe4S4] sont connues : la famille des ferrédoxines (Fd) et la famille des protéines fer-sulfure à haut potentiel. Le HiPIP et la ferrédoxine partagent le même état de repos : [Fe4S4]2+, qui ont les mêmes caractéristiques géométriques et spectroscopiques. Des différences apparaissent au niveau de leur état actif : le HiPIP se forme par oxydation en [Fe4S4]3+, et Fd se forme par réduction en [Fe4S4]+ .
[Fe4S4]3+(pour les HiPIP)Oxydation [Fe4S4]2+(repos)Réduction [Fe4S4]+(pour la ferrédoxine)
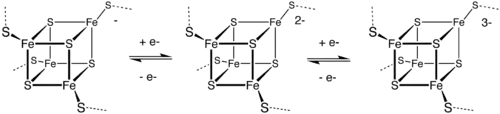
Les différents états d'oxydation s'expliquent par la combinaison des protéines avec le cluster [Fe4S4]. L'analyse des données cristallographiques suggère que le HiPIP peut conserver son état d'oxydation supérieur en formant moins de liaisons hydrogène avec l'eau. Le pli caractéristique des protéines enveloppe le cluster [Fe4S4] dans un noyau hydrophobe, ne pouvant former qu'environ cinq liaisons H conservées avec les ligands du cluster à partir du squelette. En revanche, la protéine associée aux ferrédoxines permet à ces clusters d'entrer en contact avec le solvant, ce qui entraîne des interactions de liaison H de huit protéines. La protéine se lie à la Fd via la structure conservée CysXXCysXXCys (X représente n'importe quel acide aminé)[4]. De plus, la structure protéique unique et les interactions dipolaires du peptide et de l'eau intermoléculaire contribuent à protéger le cluster [Fe4S4]3+ de l'attaque de donneurs d'électrons externes aléatoires, qui se protège de l'hydrolyse.
Analogues synthétiques
Les analogues HiPIP peuvent être synthétisés par des réactions d'échange de ligands de [Fe4S4{N(SiMe3)2}4]− avec quatre équivalents de thiols (RSH) comme suit :
- [Fe4S4{N(SiMe3)2}4]− + 4RSH → [Fe4S4 (SR)4]− + 4HN(SiMe3)2
L'amas précurseur [Fe4S4{N(SiMe3)2}4]− peut être synthétisé par une réaction en un seul pot de FeCl3, NaN(SiMe3)2 et NaSH. La synthèse d'analogues HiPIP peut aider les gens à comprendre les facteurs qui causent la variété redox de HiPIP[5].
Réactions biochimiques
Les HiPIP participent à de nombreuses réactions oxydantes chez les organismes biologiques, et sont particulièrement observés avec les bactéries anaérobies photosynthétiques, telles que Chromatium et Ectothiorhodospira. Les HiPIP sont des protéines périplasmiques des bactéries photosynthétiques. Ils jouent un rôle de navettes électroniques dans le flux électronique cyclique entre le centre de réaction photosynthétique et la coenzyme Q-cytochrome c réductase. Les autres réactions d'oxydation impliquées par HiPIP incluent la catalyse de l'oxydation du Fe (II), étant un donneur d'électrons pour la réductase et un accepteur d'électrons pour certaines enzymes oxydant le thiosulfate[6].
Références
- (en) M.M. Benning, T.E. Meyer, I. Rayment et H.M. Holden, « Molecular Structure of the Oxidized High-Potential Iron-Sulfur Protein Isolated from Ectothiorhodospira vacuolata », Biochemistry, vol. 33, no 9, , p. 2476–2483 (PMID 8117708, DOI 10.1021/bi00175a016).
- (en) « The molecular structure of the high potential iron-sulfur protein isolated from Ectothiorhodospira halophila determined at 2.5-A resolution », The Journal of Biological Chemistry, vol. 266, no 28, , p. 18660–18667 (PMID 1917989, DOI 10.2210/pdb2hip/pdb).
- (en) Perrin Jr et Ichive T., « Identifying sequence determinants of reduction potentials of metalloproteins », Biological Inorganic Chemistry, vol. 18, no 6, , p. 599–608 (PMID 23690205, PMCID 3723707, DOI 10.1007/s00775-013-1004-6)
- (en) Abhishek Dey, Francis Jr, Michael Adams et Elena Babini, « Solvent Tuning of Electronchemical Potentials in the Active Sites of HiPIP Versus Ferredoxin », Science, vol. 318, no 5855, , p. 1464–1468 (PMID 18048692, DOI 10.1126/science.1147753, Bibcode 2007Sci...318.1464D)
- (en) Yasuhiro Ohki, Kazuki Tanifuji, Norihiro Yamada et Motosuke Imada, « Synthetic analogues of [Fe4S4(Cys)3(His)] in hydrogenases and [Fe4S4(Cys)4] in HiPIP derived from all-ferric [Fe4S4{N(SiMe3)2}4] », Proceedings of the National Academy of Sciences of the United States of America, vol. 108, no 31, , p. 12635–12640 (PMID 21768339, PMCID 3150945, DOI 10.1073/pnas.1106472108)
- (en) Joan Valentine, Ivano Bertini, Harry Gray et Edward Stiefel, Biological Inorganic Chemistry: Structure and Reactivity, first, (ISBN 978-1891389436)
Liens externes
- (en-US) « High potential iron-sulfur proteins family profile », sur PROSITE (consulté le ) - Protéine fer-soufre à haut potentiel sur PROSITE
Lectures complémentaires
- (en) Terukazu Nogi, Insan Fathir, Masayuki Kobayashi, Tsunenori Nozawa et Kunio Miki, « Crystal structures of photosynthetic reaction center and high-potential iron-sulfur protein from Thermochromatium tepidum: Thermostability and electron transfer », Proceedings of the National Academy of Sciences, vol. 97, no 25, , p. 13561–13566 (PMID 11095707, PMCID 17615, DOI 10.1073/pnas.240224997, Bibcode 2000PNAS...9713561N)