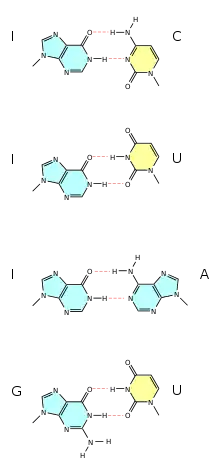
Wobble pairing
Le wobble pairing, littéralement « appariement bancal », est un mode d'appariement non canonique entre bases nucléiques que l'on observe principalement dans l'ARN. On trouve en particulier des paires de bases G–U, I–U, I–A et I–C, qui peuvent jouer un rôle important dans la structure secondaire des ARN. Elles diffèrent des paires Watson-Crick par la nature des bases et des liaisons hydrogène impliquées.
Les appariements wobble jouent un rôle très important dans la traduction du code génétique[1]. Ils permettent en effet de pallier en partie la disparité entre le nombre de codons codant (61, hors codons stop) et le nombre d'acides aminés (20), en utilisant des appariements bancals, ou wobble, à la première position de l'anticodon de l'ARN de transfert, ce qui permet à un même ARNt de reconnaître plusieurs codons synonymes. Cette hypothèse a été formulée pour la première fois par Francis Crick en 1966[2].
L'inosine (symbole I), base modifiée que l'on trouve fréquemment en première position de l'anticodon des ARNt, est particulièrement importante, car elle permet des appariements avec l'uracile (U), l'adénine (A) et la cytosine (C). La paire G–U est également très fréquente dans de nombreuses structures d'ARN.
Notes et références
- (en) Paul F. Argis, « Decoding the genome: a modified view », Nucleic Acids Res., vol. 32, , p. 223-238 (PMID 14715921, DOI 10.1093/nar/gkh185)
- (en) F.H. Crick, « Codon--anticodon pairing: the wobble hypothesis. », J. Mol. Biol, vol. 19, , p. 548-555 (PMID 5969078)