Cytométrie de masse
La cytométrie de masse (ou CyTOF, pour Cytometry by Time of Flight) est une méthode analogue à la cytométrie en flux pour l'analyse de populations cellulaires. Comme la cytométrie en flux, la cytométrie de masse repose sur le marquage spécifique d'antigènes cellulaires par des anticorps monoclonaux. Toutefois, contrairement à la cytométrie en flux, ces anticorps sont couplés à des isotopes non-radioactifs de métaux lourds (au lieu de fluorochromes) dont la détection se fait par spectrométrie de masse (au lieu de tubes photomultiplicateurs). L'utilisation d'isotopes permet ainsi de s'affranchir des limitations liées à l'autofluorescence ou au chevauchement spectral et donc de détecter un nombre d'antigènes plus important qu'en cytométrie en flux.
Principe

Marquage des cellules
Un cytomètre de masse peut théoriquement détecter 135 isotopes différents de masses atomiques allant de 75 à 209 Da[1]. En pratique, le nombre d'isotopes utilisables en cytométrie de masse est limité à 50, d'une part parce qu'il est nécessaire d'utiliser des isotopes non-cellulaires exclusivement et d'autre part parce que le nombre d'isotopes suffisamment purs est restreint. La famille des lanthanides est particulièrement utilisée en cytométrie de masse, non seulement parce que plusieurs isotopes de cette famille sont disponibles avec une pureté supérieure à 95% et que ces métaux ne sont pas présents dans les cellules, mais également parce que les métaux de cette famille ont des propriétés chimiques très proches entre eux. Les isotopes couplés à des polymères peuvent être conjugués aux anticorps par réduction partielle de ponts disulfure, permettant le couplage d'un groupement maléimide sur les polymères à des groupements cystéine-SH sur les anticorps. Ces anticorps peuvent ensuite être utilisés pour marquer les cellules de la même façon qu'en cytométrie en flux. Par ailleurs, les cellules peuvent également être marquées par du cisplatine pour évaluer leur viabilité et par une molécule s'intercalant dans l'ADN pour détecter les cellules indépendamment de leur marquage par des anticorps.
Acquisition des cellules
Les cellules passent par un nébuliseur, ce qui forme un nuage de gouttelettes contenant chacune une seule cellule. Ces gouttelettes sont ensuite ionisées par un plasma, générant des nuages d'ions comprenant les isotopes associés aux anticorps mais également divers atomes provenant des cellules (carbone, azote et oxygène). Le passage des nuages d'ions à travers un quadrupôle permet d'éliminer la plus grande partie des éléments atomiques cellulaires et de ne conserver que les isotopes lourds (> 100 Da), qui sont alors séparés dans la chambre de vol et analysés en fonction de leur rapport masse atomique sur charge.
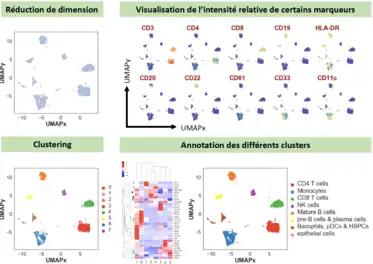
Analyse des données
Comme pour la cytométrie en flux, la cytométrie de masse permet d'identifier des populations cellulaires précises au sein d'un mélange hétérogène de cellules et d'associer une ou plusieurs populations cellulaires à un phénotype donné. Toutefois, la quantité de paramètres mesurés par cytométrie de masse ne permet pas une analyse manuelle fiable. En particulier, le nombre de paramètres mesurés correspond à autant de dimensions et l'analyse se heurte au problème de fléau de la dimension[2]. Pour contourner ce problème, les analyses classiques de cytométrie de masse impliquent l'utilisation de différents algorithmes. Dans un premier temps, la réduction de dimension projette les informations de chaque cellule sur deux dimensions (ou plus selon les algorithmes) en fonction de l'expression de chaque marqueur, fournissant une première visualisation simplifiée de l'hétérogénéité globale des populations cellulaires étudiées. Différents algorithmes permettent également de déterminer (avant ou après réduction de dimension) les groupes (clusters, ou grappes) de populations cellulaires distinctes.
Avantages et inconvénients
Avantages
L'une des limites de la cytométrie en flux réside notamment dans le fait que les anticorps sont couplés à des fluorochromes dont les spectres d'émission se chevauchent. Ce phénomène, appelé chevauchement spectral, nécessite à la fois de restreindre la détection des longueurs d'onde émises par les fluorochromes à l'aide de filtres optiques situés en amont des tubes photomultiplicateurs, et également d'effectuer une correction mathématique du chevauchement spectral résiduel (compensation). En cytométrie de masse les anticorps sont couplés à des isotopes de métaux lourds qui ne présentent presque pas de chevauchement spectral, ce qui permet d'utiliser un nombre d'anticorps (et donc de détecter un nombre d'antigènes différents) plus important. Ainsi, alors que le nombre d'anticorps utilisables simultanément en cytométrie en flux varie de 8 à 25, il est possible d'utiliser jusqu'à 40 anticorps différents en cytométrie de masse.
Inconvénients
Alors que la cytométrie en flux permet de déterminer la taille et la granulosité des cellules (paramètres FSC et SSC, respectivement), ce n'est pas directement possible en cytométrie de masse.
La cytométrie de masse implique la dégradation des cellules, et ne permet donc pas de les trier en fonction des paramètres mesurés. D'autre part, cette dégradation implique une perte de la population cellulaire de départ (près de 20 % de la population cellulaire de départ est analysée), compromettant ainsi la possibilité d'analyser des populations cellulaires très rares.
L'acquisition des cellules sur un cytomètre de masse est plus lente qu'avec un cytomètre en flux. Á titre de comparaison, un cytomètre de flux permet d'acquérir entre 2,000 et 100,000 cellules par seconde tandis qu'un cytométre de masse ne permet d'acquérir que 200 à 500 cellules par seconde[3]. La durée nécessaire pour acquérir un même nombre de cellules est donc plus importante en cytométrie de masse qu'en cytométrie de flux. Afin d'éviter une altération du signal due à la perte de sensibilité du cytomètre de masse au cours d'une acquisition, des billes de polystyrène sont ajoutées au mélange de cellules avant acquisition pour normaliser le signal[4].
Le couplage des anticorps aux isotopes s’effectue par un polymère ne permettant d’associer un anticorps qu’à 100 isotopes au maximum. L’intensité du signal correspondant aux anticorps couplés à des lanthanides est ainsi plus faible que celle de la plupart des fluorochromes classiquement utilisés en cytométrie de flux et peut donc affecter la détection d’antigènes faiblement exprimés.
Applications
La cytométrie de masse, avec le séquençage de cellule unique, a été utilisée pour étudier l'hétérogénéité de populations de cellules immunitaires jusqu'alors considérées comme étant relativement homogènes. Cette approche a été notamment appliquée à l'étude des cellules myéloïdes, lymphocytes T ou cellules lymphoïdes innées [5] - [6] - [7] - [8] - [9].
Outre l'hétérogénéité cellulaire, la cytométrie de masse a été utilisée pour mieux caractériser l'ontogénèse des cellules dendritiques[10]. La cytométrie de masse a également permis d'identifier un sous-type de cellules dendritiques contribuant à la suppression de réponses immunitaires excessives chez le fœtus[11] ou à caractériser les cellules immunitaires présentes dans le système nerveux central de la souris et à déterminer les changements associés au vieillissement ou à l'autoimmunité (en utilisant le modèle murin expérimental de sclérose en plaques)[12].
Finalement, la cytométrie de masse a été utilisée pour identifier des populations cellulaires associées à des pathologies comme la maladie cœliaque et la maladie de Crohn[13], et dans le cancer du rein[14].
Récemment, c'est grâce à la cytométrie de masse qu'il a été permis de caractériser précisément la réponse immunitaire face au SARS-CoV-2 chez les patients en réanimation[15].
Notes et références
- (en) Guojun Han et al., « Metal-isotope-tagged monoclonal antibodies for high-dimensional mass cytometry », Nature Protocols, (PMID 30258176, DOI 10.1038/s41596-018-0016-7)
- (en) « Mass cytometry: blessed with the curse of dimensionality », Nature Immunology, (PMID 27434000, DOI 10.1038/ni.3485, lire en ligne)
- Compte-tenu du fait que le nuage d'ions a une durée de vie approximative de 300 µs au cours de laquelle il est mesuré de 20 à 30 fois par le spectromètre, augmenter la rapidité du flux se ferait au détriment de la résolution cellulaire.
- (en) Rachel Finck et al., « Normalization of mass cytometry data with bead standards », Cytometry part A, (PMID 23512433, DOI 10.1002/cyto.a.22271)
- (en) Burkhard Becher et al., « High-dimensional analysis of the murine myeloid cell system », Nature Immunology, (PMID 25306126, DOI 10.1038/ni.3006)
- (en) Martin Guilliams et al., « Unsupervised High-Dimensional Analysis Aligns Dendritic Cells across Tissues and Species », Immunity, (PMID 27637149, DOI 10.1016/j.immuni.2016.08.015)
- Michael Thomas Wong et al., « A High-Dimensional Atlas of Human T Cell Diversity Reveals Tissue-Specific Trafficking and Cytokine Signatures », Immunity, (PMID 27521270, DOI 10.1016/j.immuni.2016.07.007)
- (en) Yannick Simoni et al., « Human Innate Lymphoid Cell Subsets Possess Tissue-Type Based Heterogeneity in Phenotype and Frequency », Immunity, (PMID 27986455, DOI 10.1016/j.immuni.2016.11.005)
- (en) Evan Newell et al., « Cytometry by time-of-flight shows combinatorial cytokine expression and virus-specific cell niches within a continuum of CD8+ T cell phenotypes », Immunity, (PMID 22265676, DOI 10.1016/j.immuni.2012.01.002)
- (en) Peter See et al., « Mapping the human DC lineage through the integration of high-dimensional techniques », Science, (PMID 28473638, DOI 10.1126/science.aag3009)
- (en) Naomi McGovern, « Human fetal dendritic cells promote prenatal T-cell immune suppression through arginase-2 », Nature, (PMID 28614294, DOI 10.1038/nature22795)
- (en) Dunja Mrdjen et al., « High-Dimensional Single-Cell Mapping of Central Nervous System Immune Cells Reveals Distinct Myeloid Subsets in Health, Aging, and Disease », Immunity, (PMID 29426702, DOI 10.1016/j.immuni.2018.01.011)
- (en) Vincent van Unen et al., « Mass Cytometry of the Human Mucosal Immune System Identifies Tissue- and Disease-Associated Immune Subsets », Immunity, (PMID 27178470, DOI 10.1016/j.immuni.2016.04.014)
- (en) Stéphane Chevrier et al., « An Immune Atlas of Clear Cell Renal Cell Carcinoma », Cell, (PMID 28475899, DOI 10.1016/j.cell.2017.04.016)
- Jérôme Hadjadj, Nader Yatim, Laura Barnabei et Aurélien Corneau, « Impaired type I interferon activity and inflammatory responses in severe COVID-19 patients », Science (New York, N.y.), vol. 369, no 6504, , p. 718–724 (ISSN 0036-8075, PMID 32661059, PMCID 7402632, DOI 10.1126/science.abc6027, lire en ligne, consulté le )