Séquençage de cellule unique
Le séquençage de cellule unique est un ensemble de techniques de biologie moléculaire qui permet l'analyse de l'information génétique (ADN, ARN, épigénome, etc.) à l'échelle d'une seule cellule, grâce à l'optimisation des technologies du séquençage nouvelle génération. En comparaison des techniques de séquençage traditionnel, cette technologie permet d'étudier les différences cellulaires avec une résolution optimale et ainsi de comprendre la particularité d'une cellule au sein de son micro-environnement[1] - [2].
Une nouvelle résolution
Une cellule humaine contient environ 6 milliards de paires de base d'ADN et 600 millions de base d'ARNm. Face à une telle quantité d'information, il serait difficile d'obtenir ces séquences par séquençage traditionnel (séquençage de Sanger par exemple). Appliquer les techniques de séquençage profond pour lire l'ADN et l'ARN d'une seule cellule permet d'analyser extensivement ces fonctions cellulaires[1]. À l'instar des expériences de séquençage nouvelle génération, le protocole de séquençage de cellule unique suit des étapes similaires :
- isolation des cellules,
- extraction des acides nucléiques,
- amplification,
- préparation de la library (répertoire des séquences),
- séquençage, et
- analyse bioinformatique.
Il est cependant plus difficile de séquencer sur cellules uniques que sur un groupe de cellules homogénéisées (extraits de tissu par exemple). La quantité de matériel génétique de départ est extrêmement faible et rend la qualité des résultats de l'expérience beaucoup plus sensible à la dégradation, la perte ou la contamination. De plus, travailler avec cette quantité d'acide nucléique - de l'ordre du picogramme - nécessité une étape d'amplification intensive qui génère des biais dans la quantification et la représentativité inégale des séquences générées[3].
Les améliorations techniques récentes font du séquençage de cellule unique un outil prometteur dans l'étude de problématiques auparavant inaccessibles. L'échelle de la cellule unique permet, entre autres, d'analyser : les tissus hétérogènes, les types cellulaires rares, le lignage cellulaire précis, le mosaïcisme génétique somatique, les microbes impossibles à cultiver ou encore l'évolution des maladies ou des tumeurs[4]. Ces avancées désormais à portée de mains font du séquençage de cellule unique une des révolutions techniques de ces dernières années[5].
Séquençage de l'ADN
Le séquençage de l'ADN d'une cellule unique comporte une étape d'isolation des cellules, une amplification du génome entier, la construction de la library pour enfin séquencer l'ADN grâce à un séquenceur de dernière génération (ex. Ion Torrent, Illumina). Cette méthode peut notamment être employée lors d'étude métagénomique ou pour le séquençage de nouvelles espèces. Une des méthodes populaires pour l'étape d'amplification est l'amplification à déplacement multiple (multiple displacement amplification). Cette technique a des applications en génétique microbienne, en écologie ou encore pour l'étude des maladies infectieuses. De plus, les données récoltées ainsi sur des micro-organismes pourraient permettre d'établir des procédés de culture[6]. Parmi les outils utilisés pour le séquençage de génome sur cellule unique on retrouve : SPAdes, IDBA-UD, Cortex ou HyDA[7].
Méthode
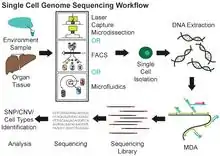
La MDA (Multiple displacement amplification) est une technique couramment employée qui permet d'amplifier des femtogrammes d'ADN bactérien pour obtenir des microgrammes de matériel utilisables pour le séquençage. Cette opération nécessite des primers aléatoires et l'ADN polymérase du bactériophage phi29. Lors de la réaction isothermale à 30 °C, l'ADN est amplifié avec ces réactifs. La MDA produit des brins d'une longueur de 12 à 100 kb utilisables pour le séquençage[6]. D'autres méthodes existent, comme le MALBAC[8].
Séquençage de l'ARN
Le séquençage de l'ARN en cellule unique permet de mesurer l'expression des gènes dans chaque cellule de l'échantillon. Le niveau d'expression de chaque gène (transcriptome) varie en fonction du type cellulaire et/ou de l'état de la cellule ce qui confère un phénotype particulier à la cellule. Par conséquent cette technologie permet d'estimer le phénotype de chaque cellule d'un échantillon. Elle est donc utilisée notamment pour cartographier un tissu au niveau cellulaire, et pour identifier des nouvelles sous-populations de cellules. Elle permet également d'estimer le niveau de différentiation de chaque cellule au sein d'un tissu, et par conséquent d'estimer la dynamique du processus de différenciation cellulaire.
Considération technique
Références
- (en) Eberwine J, Sul JY, Bartfai T, Kim J, « The promise of single-cell sequencing », Nat. Methods, vol. 11, no 1, , p. 25–27 (PMID 24524134, DOI 10.1038/nmeth.2769)
- https://www.france-science.org/Le-sequencage-a-l-echelle-de-la.html
- (en) Shintaku H, Nishikii H, Marshall LA, Kotera H, Santiago JG, « On-Chip Separation and Analysis of RNA and DNA from Single Cells », Anal. Chem., vol. 86, no 4, , p. 1953–7 (PMID 24499009, DOI 10.1021/ac4040218)
- (en) T. Nawy, « Single-cell sequencing », Nat Methods, vol. 11, no 1, , p. 18 (PMID 24524131, DOI 10.1038/nmeth.2771)
- (en) « Method of the Year 2013 », Nat Methods., vol. 11, no 1, (DOI 10.1038/nmeth.2801)
- "(en) RS. Lasken, « Single-cell Genomic Sequencing using Multiple Displacement Amplification », Curr Opin Microbiol., vol. 10, no 5, , p. 510–16 (PMID 17923430, DOI 10.1016/j.mib.2007.08.005)"
- (en) Z. Taghavi, Movahedi NS, Draghici S et Chitsaz H., « Distilled single-cell genome sequencing and de novo assembly for sparse microbial communities. », Bioinformatics., vol. 29, no 19, , p. 2395–2401 (DOI 10.1093/bioinformatics/btt420)
- "(en) L. Ning, Liu G, Li G, Hou Y, Tong Y et He J., « Current Challenges in the Bioinformatics of Single Cell Genomics. », Front Oncol., vol. 4, no 7, (PMID 24478987, PMCID 3902584, DOI 10.3389/fonc.2014.00007)"