Réparation SOS
La réparation SOS est un système de survie des bactéries en réponse à des lésions importantes de leur ADN.
Quelques dates importantes
- 1974 : Miroslav Radman, directeur de recherche au CNRS, est le premier à formuler l’existence d’une réparation SOS chez la bactérie. Il propose que les mutations soient liées à une réponse globale de la bactérie aux chocs génotoxiques et nomme ce phénomène la réponse SOS. Il postule l’existence de « polymérases SOS » inductibles par le stress cellulaire, capable de copier les nucléotides chimiquement altérés, rendant possible la survie cellulaire.
- 1999 : la mise en évidence des « polymérases SOS » vient confirmer l’hypothèse de Radman quant à l’existence de la réparation SOS de l’ADN. On démontre également que ce système est parfois accompagné de mutations capables d’entraîner une évolution adaptative des espèces.
Mécanisme d’action chez Escherichia coli
Le nombre de mécanismes d’action de la réparation SOS est variable en fonction des espèces bactériennes. En effet, chez Bacillus subtilis on dénombre quatre systèmes différents contre un seul chez Escherichia coli. La différence entre ces mécanismes est la nature des protéines impliquées qui peuvent muter. Le mécanisme le plus souvent rencontré est celui mis en évidence par E. coli.
Les différents effecteurs
- RecA : est une protéine essentielle dans le mécanisme de réparation SOS car elle permet l’expression des gènes codant les protéines de réparation de l’ADN.
- LexA : est une protéine qui joue le rôle de répresseur des gènes codant des protéines de réparation de l’ADN.
- SulA : est une protéine permettant l’inactivation de la réplication de l'ADN et de la division de la bactérie.
- Uvr A,B,C : sont des endonucléases du système d’excision. Ces protéines sont traduites en temps normal mais leur concentration s’accroît lors de la réponse SOS.
- UmuD ‘et UmuC : UmuD’ est une protéine modifiée provenant du clivage de la protéine UmuD, se complexant avec la protéine UmuC pour former un complexe UmuD’2C.
Mécanisme
- La réparation SOS va être déclenchée lorsque la bactérie va subir un stress important ce qui va entraîner des lésions de l’ADN mettant sa survie en péril.
- La bactérie ne peut pas stopper sa réplication (à l'inverse des eucaryotes) et devra agir plus vite pour réparer l'ADN endommagé. [Néanmoins il est possible de stopper sa réplication artificiellement en insérant la protéine SulA qui est une molécule utilisée comme un antibactérien car elle induit un blocage de la réplication car elle inhibe la gyrase, une topoisomérase impliquée dans la réplication de l'ADN.]
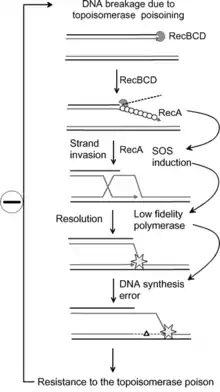
- La protéine RecA va se lier au niveau des brèches de l’ADN et amorcer les échanges de brins. Elle a également une activité protéolytique qui va permettre de cliver la protéine LexA.
- La protéine LexA est un répresseur du système SOS, lorsque celle-ci est clivée, les gènes de la réparation SOS vont alors être traduits en grande quantité.
- Sur l’ADN endommagé, le gène umuDC va être traduit en protéines. Ce dernier donne le pouvoir mutagène à la bactérie et lui permet de survivre. D’autres gènes de la réponse SOS nommés uvr vont enlever les bases endommagées. Ils codent l’endonucléase du système d’excision généralisé afin de réparer l’ADN lésé. Les protéines UmuC et UmuD néosynthétisées aident la polymérase III à franchir les obstacles dus aux mutations.
- La protéine UmuD va se cliver automatiquement en une protéine UmuD’ modifiée qui va se complexer à UmuC. Ceci forme un complexe UmuD’2C qui va permettre de réparer le brin d’ADN lésé en permettant à la polymérase III de passer au-dessus des obstacles dus aux lésions.
Implication du mécanisme SOS sur l’évolution des bactéries
Résistance bactérienne aux antibiotiques
Tout d’abord, il est à préciser que ce phénomène fonctionne seulement pour les antibiotiques attaquant la bactérie au niveau de l’ADN. Ainsi, des scientifiques Français ont pu démontrer que la résistance aux antibiotiques est finement liée au mécanisme de réparation SOS de l’ADN (Guérin et al.). Chez les bactéries, des éléments génétiques appelés intégrons sont connus pour être les principaux acteurs dans l'acquisition et la dissémination des multi-résistances aux antibiotiques. Les intégrons sont des plateformes au niveau desquelles sont insérées et exprimées des cassettes de gène pouvant contenir entre autres des gènes de résistances à des antibiotiques. L'intégration de ces cassettes est permise par l'activité d'une intégrase particulière, IntI, codée par le gène intI retrouvé chez tous les intégrons. Or, chez certaines des intégrases d'intégron, un motif homologue au site de fixation de LexA a pu être identifié. Il a ainsi été montré que la réponse SOS régule l'expression de l'intégrase de l'intégron de classe 1 de Escherichia coli et de l'intégon VchIntIA de Vibrio cholerae. Ainsi, lorsque la réponse SOS est déclenchée, l'expression de l'intégrase est activée ce qui va permettre l'insertion et le réarragement des cassettes de gène, ce qui constitue un avantage adaptatif notamment chez les bactéries pathogènes Gram -.
Réparation SOS à l’origine de la distinction des bactéries à paroi Gram négatif / Gram positif
Il est probable que l’ancêtre commun à toutes les bactéries à paroi Gram positif ait été une bactérie à paroi Gram négatif ayant perdu sa membrane externe et dont la paroi s’est épaissie en réponse aux contraintes de cette nouvelle structure. Cette séparation aurait eu lieu il y a 1,4 milliard d’années. Ainsi, une étude a récemment montré que les mutations induites par la réparation SOS seraient à l’origine de la divergence entre les bactéries à paroi Gram positif et Gram négatif. Ces différentes divergences entre espèces sont utilisées par les scientifiques pour estimer la date à laquelle vivaient d’autres organismes.
Voir aussi
Sources
- Lederberg J, Encyclopedia of Microbiology, « SOS Response », second édition, vol.4, p. 336-343, 2000
- Taddei F, Matic I, Radman M, Pour la science, « SOS génome : réparation et évolution », no 269, , p66-73
- Little JW, Mount DW, The SOS regulatory system of Escherichia Coli, Cell, 1982
- Prescott, Harley, Klein, Microbiologie, 2° édition, 2003, p. 255-256
- Guérin E, Cambray G, Sanchez-Alberola N, Campoy S, Erill I, Da Re S, Gonzalez-Zorn B, Barbé J, Ploy M-C, Mazel D, "The SOS Response Controls Integron Recombination", Science, May 2009, vol324, p1034
- http://www.academie-sciences.fr/membres/R/Radman_Miroslav.htm, consulté le 06/05/10
- http://bacterioblog.over-blog.com/100-index.html
- http://www.bulletins-electroniques.com/actualites/59743.htm
- Autre : support cours Biologie moléculaire de Mme Michaud, 2009-2010, IUT Clermont Ferrand, ABB