Hélentron
Un hélentron est un type d'élément transposable de classe II, fonctionnant uniquement par un intermédiaire ADN.
Tout comme les hélitrons (en), les hélentrons ne produisent pas de duplication dans les chaînes latérales de l'ADN envahi. Initialement reconnus comme des hélitrons, les hélentrons sont distingués pour la première fois en 2003[1] par l'analyse des séquences ADN du poisson-zèbre (Danio Rerio) et de Sphoeroides nephelus.
Structure
Les hélentrons présentent plusieurs séquences caractéristiques, situées entre leurs deux extrémités terminales[2]:
- deux répétitions inversées (Terminal Inverted Repeats) au niveau des extrémités du transposon, le plus souvent palindromiques et mesurant entre 12 et 15 paires de bases (bp),
- une séquence palindromique supplémentaire située avant l'extrémité 3' du transposon, impliquée dans la reconnaissance du site de terminaison de l'hélentron,
- une seule phase de lecture ouverte (Open Reading Frame) codant une longue protéine Rep-Hel composée de 3 domaines protéiques différents: 1) un motif N-terminal en doigt de zinc permettant la reconnaissance de l'hélentron par la protéine codée ainsi qu'une endonucléase Rep différente de celle des hélitrons par une variation sur trois acides aminés[3]; 2) une hélicase réalisant le parcours des chaînes ADN de l'extrémité 5' à 3' ; 3) une AP-endonucléase C-terminale (que ne possèdent pas les hélitrons).
Mécanisme de duplication et capture de gènes
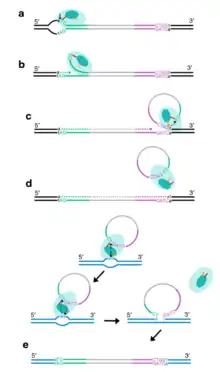
Le mécanisme de duplication des hélentrons est encore mal compris mais, tout comme celui des hélitrons, il se fait par réplication circulaire: après avoir clivé un seul brin de l'hélentron en son extrémité 5', Rep-Hel s'avance le long de la séquence ADN par activité de l'hélicase en arrachant progressivement ce brin ADN et enroulant celui-ci pour former un cercle liant les deux extrémités du transposon entre elles. Par activité de l'endonucléase, l'hélentron est ensuite intégré à la séquence ADN-cible de l'hôte dans une zone riche en thymine, entre une paire TT[2].
De nombreux hélentrons comprennent également des séquences ADN appartenant à l'hôte[3]. Celles-ci sont issues de l'intégration des séquences au voisinage de l'hélentron dans le transposon lui-même. En effet, il peut arriver que l'enroulement de la séquence de l'hélentron se poursuive au-delà de la terminaison 3' du transposon et englobe une partie du génome avoisinant qui sera alors déplacé dans une autre région. C'est notamment le cas chez le moustique Culex quinquefasciatus où une transposition d'hélentron a donné naissance à huit copies d'un protogène codant une nouvelle histone[2].
La capacité à créer de tels gènes de novo peut s'expliquer de plusieurs façons: si l'hélentron capture une séquence de l'hôte correspondant à un promoteur, en s'insérant ailleurs dans le génome, l'hélentron peut activer une portion d'ADN précédemment non transcrite grâce à l'introduction de ce promoteur. Si ce gène apporte une amélioration à l'hôte, il peut être amené à s'amplifier et se distinguer par sélection positive. Par ailleurs, l'hélentron peut également transposer un exon et l'insérer dans une séquence codante ailleurs dans le génome, participant ainsi au brassage d'exons[5].
Distribution
La distribution des hélentrons au sein des eukaryotes est moins grande que celle des hélitrons. En particulier, alors que ceux-ci sont en quantité très importante chez de nombreuses espèces de plantes (notamment le maïs où les hélitrons auraient participer à la formation de 24 000 protogènes[6]), aucun hélentron n'a encore été détecté au sein de ces organismes.
Les hélentrons sont ont été trouvés chez les poissons, les oomycètes, et les insectes (en particuier, chez la drosophile où l'élément transposable le plus abondant, DINE-1, est la version non autonome d'un hélentron[3]). Aucun hélentron n'a été trouvé chez les mammifères à ce jour.
Lien avec les hélitrons
Lors de l'analyse du génome du mildiou de la pomme de terre (Phytophthora infestans), une séquence comportant les domaines Rep et Hel identiques à ceux conservés chez tous les hélentrons a été identifiée, avec cependant l'absence notable du domaine lié à l'AP-endonucléase[3]. Ce proto-hélentron pourrait faire le lien entre les deux familles de transposons à réplication circulaire présents chez les eukaryotes que sont les hélitrons (en) et les hélentrons: il fait apparaître l'hypothèse d'un ancêtre commun aux deux transposons présentant les spécificités nucléotidiques des hélentrons avant l'acquisition du domaine de l'AP-endonucléase.
Notes et références
- Russell T. M. Poulter, Timothy J. D. Goodwin et Margaret I. Butler, « Vertebrate helentrons and other novel Helitrons », Gene, vol. 313, , p. 201–212 (ISSN 0378-1119, DOI 10.1016/S0378-1119(03)00679-6, lire en ligne, consulté le )
- Jainy Thomas et Ellen J. Pritham, « Helitrons, the Eukaryotic Rolling-circle Transposable Elements », Microbiology Spectrum, vol. 3, no 4, (ISSN 2165-0497, PMID 26350323, DOI 10.1128/microbiolspec.MDNA3-0049-2014, lire en ligne, consulté le )
- Jainy Thomas, Komal Vadnagara et Ellen J Pritham, « DINE-1, the highest copy number repeats in Drosophila melanogaster are non-autonomous endonuclease-encoding rolling-circle transposable elements (Helentrons) », Mobile DNA, vol. 5, , p. 18 (ISSN 1759-8753, PMID 24959209, PMCID 4067079, DOI 10.1186/1759-8753-5-18, lire en ligne, consulté le )
- Ivana Grabundzija, Simon A. Messing, Jainy Thomas et Rachel L. Cosby, « A Helitron transposon reconstructed from bats reveals a novel mechanism of genome shuffling in eukaryotes », Nature Communications, vol. 7, (ISSN 2041-1723, PMID 26931494, PMCID 4778049, DOI 10.1038/ncomms10716, lire en ligne, consulté le )
- Jainy Thomas, Caleb D. Phillips, Robert J. Baker et Ellen J. Pritham, « Rolling-Circle Transposons Catalyze Genomic Innovation in a Mammalian Lineage », Genome Biology and Evolution, vol. 6, no 10, , p. 2595–2610 (ISSN 1759-6653, PMID 25223768, PMCID 4224331, DOI 10.1093/gbe/evu204, lire en ligne, consulté le )
- Lixing Yang et Jeffrey L. Bennetzen, « Distribution, diversity, evolution, and survival of Helitrons in the maize genome », Proceedings of the National Academy of Sciences of the United States of America, vol. 106, no 47, , p. 19922–19927 (ISSN 0027-8424, PMID 19926865, PMCID 2785268, DOI 10.1073/pnas.0908008106, lire en ligne, consulté le )