Fluorescence-Activating and absorption-Shifting Tag
FAST (Fluorescence-Activating and absorption-Shifting Tag) est une petite étiquette protéique qui permet le marquage fluorescent de protéines d'intérêt auxquelles elle est génétiquement fusionnée. Non fluorescente, contrairement aux protéines naturellement fluorescentes comme la GFP, elle peut s'associer sélectivement à un chromophore fluorogénique dérivé du 4-hydroxybenzylidène rhodanine (HBR), lui-même non fluorescent sous sa forme libre. C'est la complexation entre FAST et le fluorogène qui déclenche la fluorescence du système sous l'effet d'un double changement spectroscopique : augmentation du rendement quantique de fluorescence et déplacement vers le rouge de la bande d'absorption du fluorogène. FAST, petite protéine de 14 kDa, a été générée par évolution dirigée à partir de la protéine photoactive jaune (en) (PYP). Le système FAST-fluorogène peut être utilisé en microscopie à fluorescence, en cytométrie en flux ou toute autre méthode fluorométrique pour explorer le vivant : biosenseurs, trafic de protéines, criblage à haut contenu (en), etc.
L'invention de FAST a été publiée pour la première fois en 2016 par des chercheurs de l'École normale supérieure de Paris[1].
Mécanisme
FAST relève d'une approche hybride de marquage sélectif des protéines : un domaine peptidique, appelé "tag", est fusionné génétiquement à une protéine d'intérêt (par combinaison de leurs gènes respectifs par transfection ou infection) et joue le rôle de point d'ancrage pour une sonde fluorescente synthétique.
Cette approche chémogénétique est déjà mise en œuvre dans plusieurs systèmes :
- depuis 2003, SNAP-tag (en), ultérieurement évolué en CLIP-tag, un système bicomposant constitué d'un peptide de 19 kDa dérivé d'une enzyme humaine, l'O6-méthylguanine-ADN méthyltransférase, évoluée pour réagir avec des dérivés O6-benzylguanine-fluorophore ;
- depuis 2008, HaloTag (en), un système bicomposant constitué d'un peptide de 33 kDa dérivé d'une enzyme bactérienne, une haloalkane deshalogénase, qui se lie spécifiquement à un ligand synthétique halogéné fonctionnel, le plus souvent fluorescent pour l'imagerie (par ex. Coumarine, Oregon Green, Alexa Fluor 488, diAcFAM et TMR).
FAST a été obtenu à partir de la protéine photoactive jaune (PYP), un photorécepteur naturel présent chez Halorhodospira halophila (en), une bactérie extrémophile vivant dans des milieux hypersalins. Ce photorécepteur réagit à la lumière bleue grâce à la présence d'une molécule d'acide para-hydroxycinnamique attachée à un résidu cystéine. L'acide para-hydroxycinnamique a une structure proche de 4-hydroxybenzylidène rhodanine (HBR) et de ses dérivés qui sont des fluorogènes, c'est-à-dire qu'ils ne sont pas fluorescents en solution, mais peuvent le devenir lorsque l'on restreint leurs degrés de liberté, par exemple par complexation. La PYP a donc été mutée par des techniques d'évolution dirigée pour exacerber sa reconnaissance d'analogues d'HBR et l'activation de leur fluorescence. Finalement FAST a été généré par introduction de neuf mutations.
Deux versions de FAST sont décrites, différant par une mutation :
- FAST1 : MEHVAFGSEDIENTLAKMDDGQLDGLAFGAIQLDGDGNILQYNAAEGDITGRDPKQVIGKNFFKDVAPGTDSPEFYGKFKEGVAS
GNLNTMFEWMIPTSRGPTKVKVHMKKALSGDSYWVFVKRV
- FAST2 (a.k.a. iFAST[2]): MEHVAFGSEDIENTLAKMDDGQLDGLAFGAIQLDGDGNILQYNAAEGDITGRDPKQVIGKNFFKDVAPGTDSPEFYGKFKEGVAS
GNLNTMFEWMIPTSRGPTKVKIHMKKALSGDSYWVFVKRV
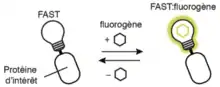
Contrairement aux systèmes décrits plus hauts, FAST présente donc en particulier la double caractéristique suivante :
- le ligand ne devient fluorescent qu'une fois lié avec le tag protéique ; sa forme libre ne génère pas de bruit de fond ;
- la liaison entre le tag protéique et le fluorogène est non covalente ; c'est un complexe, réversible donc, on peut "éteindre" la fluorescence par un simple lavage du fluorogène.
Applications
De nombreux fluorogènes ont été développés offrant toute une gamme de couleurs différentes et d'affinités différentes pour le tag FAST et ses dérivés, e.g., tdFAST[3]. Certains d'entre eux, non perméants, c'est-à-dire qu'ils ne peuvent pas traverser la membrane cellulaire, marquent spécifiquement les protéines membranaires ou extracellulaires permettant ainsi de suivre leur trafic à partir de leur traduction jusqu'à leur excrétion[4].
FAST est utilisé pour l'imagerie de biofilms anaérobies[5] grâce à sa faculté de fluorescer même dans des milieux pauvres en oxygène contrairement à la GFP ou ses dérivés. Pour la même raison, il permet d'imager Clostridium[6], une bactérie anaérobie utilisée pour la fermentation de la biomasse, plus particulièrement la fermentation acétone-butanol-éthanol.
FAST peut aussi être appliqué pour la microscopie à super-résolution de cellules vivantes[7].
Références
- Plamont, M. A., Billon-Denis, E., Maurin, S., Gauron, C., Pimenta, F. M., Specht, C. G., ... & Moncoq, K. (2016). Small fluorescence-activating and absorption-shifting tag for tunable protein imaging in vivo. Proceedings of the National Academy of Sciences, 113(3), 497-502.
- Tebo, A. G., Pimenta, F. M., Zhang, Y., & Gautier, A. (2018). Improved chemical-genetic fluorescent markers for live cell microscopy. Biochemistry, 57(39), 5648-5653.
- « The Twinkle Factory »
- Li, C., Mourton, A., Plamont, M. A., Rodrigues, V., Aujard, I., Volovitch, M., ... & Joliot, A. (2018). Fluorogenic probing of membrane protein trafficking. Bioconjugate chemistry, 29(6), 1823-1828.
- Monmeyran, A., Thomen, P., Jonquière, H., Sureau, F., Li, C., Plamont, M. A., ... & Henry, N. (2018). The inducible chemical-genetic fluorescent marker FAST outperforms classical fluorescent proteins in the quantitative reporting of bacterial biofilm dynamics. Scientific reports, 8(1), 10336.
- Charubin, K., Bennett, R. K., Fast, A. G., & Papoutsakis, E. T. (2018). Engineering Clostridium organisms as microbial cell-factories: challenges & opportunities. Metabolic engineering, 50, 173-191.
- Venkatachalapathy, M., Belapurkar, V., Jose, M., Gautier, A., & Nair, D. (2019). Live cell super resolution imaging by radial fluctuations using fluorogen binding tags. Nanoscale, 11(8), 3626-3632.