Diversité phylogénétique
La diversité phylogénétique est une mesure de la biodiversité considérant les relations de parenté entre les espèces[1]. Cette mesure permet d’incorporer l’histoire évolutive dans les indices de biodiversité d’une communauté. Pour ce faire, il faut utiliser les longueurs de branches d'un arbre phylogénétique séparant les différentes unités taxonomiques (p.ex. les espèces) d'un ensemble. Ainsi, contrairement à la richesse spécifique, qui ne s’intéresse qu'au nombre d’espèces présentes à un site, chaque taxon n'a pas le même poids dans la mesure de la diversité phylogénétique. Une espèce ayant divergé plus tôt que les autres aura un poids plus élevé, puisqu’elle a une plus longue histoire évolutive indépendante aux autres espèces. La diversité phylogénétique est associée à la diversité phénotypique et fonctionnelle, et donc, à la stabilité d'un écosystème et sa productivité. Cette mesure a été développée dans le but d'aider à identifier les priorités de conservation.

Histoire
Traditionnellement, la biodiversité est mesurée par le nombre d’espèces présentes à un site (richesse spécifique). Pour cette mesure, chaque espèce contribue également à la mesure de biodiversité. En 1972, Robert Whittaker suggère que cette simple mesure n’est pas suffisante pour quantifier toute la complexité de la biodiversité[2]. Il propose donc trois mesures permettant de mieux définir la diversité spécifique :
- Diversité alpha : Le nombre d’espèces coexistant dans un même site.
- Diversité bêta : Le taux de remplacement des espèces dans un gradient environnemental. Elle consiste à comparer les espèces uniques aux différents sites.
- Diversité gamma : La diversité totale des espèces dans un paysage. La diversité gamma est donc le produit de la diversité bêta et alpha.

Puis, dans un article publié dans Nature en 1990, Robert May discute de la façon dont chaque espèce devrait être valorisée lors de l'établissement d'un plan de conservation[3]. Selon lui, chaque espèce ne devrait pas être considérée égale lorsque l'on mesure la biodiversité. Il prend en exemple le tuatara (sphénodon), un genre ne comptant que quelques espèces et le seul encore existant de l’ordre des Rhynchocephalia. Cet ordre étant le seul groupe frère des squamates (lézards et serpents), il est un taxon très important dans la compréhension de l’évolution des reptiles. Selon Robert May, il faudrait valoriser davantage la conservation d’un tel organisme que d’autres ayant plusieurs espèces proches, comme les couleuvres. En effet, il considère que le tuatara, étant un taxon particulièrement distinct parmi les reptiles, devrait présenter un intérêt particulier en conservation. Il propose donc de modifier la façon dont l’on mesure la biodiversité afin de prendre en compte non seulement l'abondance de chaque espèce, mais aussi leur histoire évolutive.
À la suite de cet article, Van-Wright, Humphries et Williams ont introduit un premier indice prenant en compte l’histoire évolutive des espèces[4]. En 1991, ils présentent la diversité taxique. Cette mesure utilise les informations présentes dans un cladogramme afin de prendre en compte le caractère distinctif de chaque espèce. En d'autres mots, ils proposent de regarder les relations de parenté des espèces présentes à un site pour en déterminer leur unicité. Moins une espèce a d’ancêtres communs avec les autres, plus elle est considérée comme distincte. Pour calculer la diversité taxique d’un ensemble d’espèces, les auteurs proposent la démarche suivante[4] :
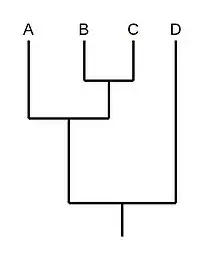
- Pour chaque espèce, calculer le nombre d’ancêtres communs au reste des espèces (le nombre de nœuds du cladogramme de la racine à chaque feuille). Cela représente notamment le nombre de sous-groupes que l'on peut créer pour lesquels on peut inclure chaque espèce. Dans l’exemple de cladogramme, l’espèce A fait partie de deux sous-groupes : ABC et ABCD.
- Faire la somme des nombres d’ancêtres communs calculés précédemment. Dans l’exemple, cette somme est 9 (2 pour l'espèce A, 3 pour l'espèce B, 3 pour l'espèce C et 1 pour l'espèce D).
- Pour chaque espèce, diviser la somme calculée à l’étape 2 par le nombre de groupes pour lesquels l’espèce est membre (étape 1).
- Afin de standardiser les poids de diversité taxique, il faut diviser chaque valeur calculée à l’étape 3 par la valeur minimale. Ainsi, dans l'exemple de cladogramme, les poids de diversité taxique des espèces A, B, C et D sont respectivement 1.5, 1, 1 et 3. Plus ce chiffre est élevé, plus l'espèce est considérée comme distincte par rapport aux autres.
Cette mesure de biodiversité, bien que proche, n'est pas exactement égale à la diversité phylogénétique. C’est en 1992 que celle-ci a été introduite par Daniel P. Faith[1]. Ce dernier définit la diversité phylogénétique d’un assemblage d’espèces comme la somme de la longueur des branches d’un arbre phylogénétique reliant l’ensemble des membres de cet assemblage. Ainsi, en plus de considérer la position des espèces dans le cladogramme, comme proposé par Richard Vane-Wright et ses collaborateurs[4], cet indice considère la longueur des branches, soit le temps de divergence des différentes espèces. Les arbres phylogénétiques étant traditionnellement construits à partir des traits des espèces, une grande diversité phylogénétique devrait être synonyme d’une grande diversité de traits (comportementales, morphologiques, etc.). De plus, il propose que cette mesure ne devrait pas être réservée pour comparer uniquement des espèces, mais qu'elle peut aussi être utilisée pour d’autres niveaux taxonomiques comme les populations d'une même espèce ou les genres.
Cette mesure peut être utilisée pour comparer la biodiversité de différents sites d’un écosystème. Pour ce faire, Daniel P. Faith propose la méthodologie suivante[1] :
- Construire l’arbre phylogénétique de l’ensemble des espèces présentes dans l’écosystème.
- Additionner les longueurs des branches de l’arbre bâti à l’étape 1 permettant de connecter le sous-ensemble d’espèces présentes à un site.
- Répéter l’étape 2 pour chaque site.
Ainsi, en comparant le score des différents sites, il est possible de déterminer ceux qui contribuent le plus à la diversité phylogénétique de l’écosystème. Ceux-ci devraient donc faire l’objet d’un effort de conservation particulier.
Il est aussi possible de trouver la contribution de chaque espèce à la diversité phylogénétique. Une des méthodes suggérées est de simuler l’extinction de chacune des espèces et d’en comparer l’effet sur l'indice de diversité phylogénétique. Les espèces pour lesquelles l’extinction résulte à une grande perte de diversité phylogénétique devraient être particulièrement importantes dans la conservation de l'histoire évolutive de la communauté.
Depuis l'introduction du concept de diversité phylogénétique, plusieurs travaux ont porté sur l'utilisation de nouveaux indices. De plus, le domaine de l'écologie des communautés s'est penché sur les relations entre la diversité phylogénétique et la composition, la structure et les fonctions d'une communauté.
Mesures
De très nombreux indices de diversité phylogénétique ont été développés avec les années. Il existe plus de 70 mesures de la diversité phylogénétique, dont plusieurs sont fortement corrélées[5]. Certains indices ont incorporé l’abondance des différentes unités taxonomiques dans le calcul. Ainsi, pour ces indices, les espèces abondantes ont un poids supérieur ou inférieur comparativement aux espèces rares. D’autres comparent différents ensembles de taxa, un peu comme le fait la diversité bêta. Par exemple, certains indices calculent la longueur des branches des espèces uniques à chaque site. Finalement, certains indices combinent la diversité phylogénétique et le nombre d'espèces. Tous ces indices peuvent être divisés en trois catégories selon ce qu’ils cherchent à mesurer : la richesse, la divergence et la régularité.
Richesse
Les mesures de richesse phylogénétique servent à quantifier la différence phylogénétique d’un ensemble de taxa[5]. Ainsi, ces indices permettent de mesurer l’histoire évolutive accumulée par un ensemble de taxa. L'indice le plus commun est la diversité phylogénétique proposée par Faith[1]. Celle-ci fait la somme des longueurs de branches de l’arbre incluant tous les membres de l’ensemble. Au lieu d'utiliser la longueur des branches, d'autres indices font la somme de la distance entre toutes les paires d'espèces d'un ensemble.
Divergence
Les mesures de divergence cherchent à quantifier la distribution des taxa d’un ensemble[5]. Par exemple, il est possible de calculer la divergence phylogénétique en divisant la richesse phylogénétique par la richesse spécifique. Ainsi, on obtient la longueur de branches moyennes reliant chaque unité taxonomique. Une autre mesure courante de la divergence est de calculer la moyenne des longueurs de branches entre chaque paire de taxa. Ainsi, on mesure la distance phylogénétique moyenne entre les taxa. Ces indices servent donc à quantifier la différence entre les unités taxonomiques d’un ensemble d’intérêt.
Régularité
La régularité mesure à quel point les taxa d’un ensemble sont également distancés phylogénétiquement[5]. En d’autres mots, on compare la distribution des taxa d’une phylogénie à un arbre phylogénétique dont tous les taxa ont une distance évolutive égale. La régularité est maximale lorsque toutes les paires d’espèces sont également distancées sur l’arbre phylogénétique. Au contraire, si certaines espèces sont très proches et d'autres éloignées, la régularité sera inférieure. Une mesure permettant de quantifier cette régularité phylogénétique est la variance des distances entre chaque paire d’un ensemble.
Intérêts en conservation
Le concept de diversité phylogénétique a d’abord été développé pour définir les priorités en conservation[1]. En effet, les programmes de conservation, ayant des ressources souvent limitées, doivent choisir quelles espèces ou quels sites pour lesquels les efforts de conservation devraient être concentrés. La diversité évolutive a notamment été identifiée comme l’une des priorités en conservation[3]. Celle-ci devrait être étroitement liée à la diversité phénotypique (diversité des traits comportementaux, morphologiques, etc.). Il est aussi suggéré que la diversité phylogénétique est liée aux services écosystémiques et la résilience face aux changements environnementaux. Ainsi, conserver la diversité évolutive a été avancée comme une approche plus efficace pour conserver les services écosystémiques que la conservation de la richesse spécifique, par exemple.
La première approche de conservation est celle visant les espèces. Elle consiste à déterminer quelles espèces devraient être une priorité de conservation. Avec le calcul de la diversité phylogénétique, il est possible de déterminer la contribution de chaque espèce, ou groupe taxonomique, à la biodiversité selon l’histoire évolutive d’un ensemble d’organismes[1]. Il est aussi possible de déterminer un sous-ensemble d’unités taxonomiques maximisant la diversité phylogénétique. L’une des stratégies est de former différentes combinaisons d’espèces en sous-ensembles et d’en comparer leur diversité. Cela permet d’identifier les combinaisons d'espèces (ou autre unité taxonomique) permettant de maximiser la conservation de la diversité évolutive.
Une seconde approche consiste à protéger les sites ayant une grande diversité phylogénétique. Pour ce faire, il faut déterminer et comparer la diversité phylogénétique de différents endroits. Les sites ayant la plus grande diversité phylogénétique devraient être ceux à prioriser afin de conserver au maximum l’histoire évolutive d’un paysage.
Même si la diversité phylogénétique est un concept qui, en théorie, devrait influencer les priorités de conservation, très peu de programmes utilisent cette mesure de biodiversité[6]. Une des raisons qui a été avancée pouvant expliquer cette hésitation à inclure cette mesure est le nombre d'indices qui ont été proposés. En effet, le nombre d'indices ayant rapidement augmenté, il n'était pas clair, jusqu'à récemment, quelle mesure de diversité phylogénétique devait être maximisée. Une autre raison qui a été suggérée est le manque d'études montrant le lien entre la diversité phylogénétique, la résilience et les services d'un écosystème.
Utilisation en écologie des communautés et des écosystèmes
Depuis l’introduction du concept de diversité phylogénétique, plusieurs études se sont intéressées aux liens entre celui-ci et la composition et structure d’une communauté, ainsi que les services écosystémiques[7]. Par exemple, une étude s’est penchée sur la relation entre la productivité d’une communauté de végétaux et différentes mesures de diversité. Ils ont montré que la productivité pouvait être mieux prédite par la diversité phylogénétique que par la diversité spécifique ou fonctionnelle[8].
Une autre étude s’est intéressée au lien entre la stabilité d’un écosystème et la diversité phylogénétique [9]. Pour faire cela, ils ont regardé la réponse de la biomasse de différentes communautés végétales face à des variations environnementales. Ils ont trouvé que cette stabilité était supérieure dans les communautés qui comportaient des espèces plus distancées phylogénétiquement, que lorsque les espèces étaient tous proches.
Un dernier exemple d’étude a regardé la structure phylogénétique de communautés d’arbres dans une forêt tropicale du Panama[10]. Cette étude a trouvé que pour les jeunes communautés et celles sur les plateaux, les arbres avaient tendance à être regroupés avec des espèces phylogénétiquement similaires. Au contraire, dans les habitats en pente ou dans les marais, les espèces d’arbres avaient tendance à être regroupées parmi des espèces plus phylogénétiquement distinctes.
Critiques
Quelques critiques ont été faites à l'utilisation de la diversité phylogénétique. Celles-ci pourraient, entre autres, expliquer pourquoi cette mesure n'est pas encore couramment utilisée dans l'identification des priorités de conservation.
Une première critique de la diversité phylogénétique est sa forte corrélation avec la diversité spécifique. En d’autres mots, maximiser le nombre d’espèces devrait, dans bien des cas, maximiser aussi l’histoire évolutive d’un ensemble d’espèces. L’argument est donc que de s’attarder à la diversité phylogénétique n’est pas d’un réel intérêt, car cela ajoute une complexité et une incertitude inutiles dans la conception d'un plan de conservation. Il a cependant été démontré que le lien entre richesse spécifique et diversité phylogénétique n’était pas aussi fort que certains auteurs ont présumé, et que l’utilisation de la diversité phylogénétique pouvait avoir des conséquences non négligeables sur les politiques de conservation. Par exemple, une étude sur la région floristique du Cap a montré que la détermination des priorités de conservation basées sur la mesure de diversité phylogénétique modifiait les sites à protéger comparativement à ceux déterminer avec la richesse spécifique [11]. L’étude a aussi montré que ces différences permettaient de maximiser davantage la diversité phénotypique (diversité des traits morphologiques, comportementaux, etc.).
Une seconde critique vise la relation entre diversité phylogénétique et la diversité fonctionnelle. Un des arguments importants pour l’utilisation de la diversité phylogénétique est qu’elle devrait permettre de maximiser aussi la diversité fonctionnelle. Celle-ci est extrêmement importante, notamment car elle est associée au fonctionnement des écosystèmes. Cependant, sa mesure est souvent très incertaine et nécessite beaucoup de ressources. Ainsi, la diversité phylogénétique, étant plus simple à mesurer, a été suggérée comme indice permettant de maximiser la diversité fonctionnelle. Or, récemment, des études ont montré que dans bien des cas, maximiser la diversité phylogénétique ne permettait pas de conserver davantage la diversité fonctionnelle[12] - [13]. Les auteurs de ces études ont notamment montré que dans certains cas, le fait de maximiser la diversité phylogénétique pouvait, au contraire, diminuer la diversité fonctionnelle.
Notes et références
- D.P. Faith, Conservation evaluation and phylogenetic diversity, Biological conservation, 61(1), 1992, p. 1-10.
- R.H. Whittaker, Evolution and measurement of species diversity, Taxon, 1972, p. 213-251.
- R.M. May, Taxonomy as destiny, Nature, 347(6289), 1990, p. 129-130.
- R.I. Vane-Wright, C.J. Humphries, P.H. Williams, What to protect?—Systematics and the agony of choice, Biological conservation, 55(3), 1991, p. 235-254.
- C.M. Tucker, M.W. Cadotte, S.B. Carvalho, T.J. Davies, S. Ferrier, S.A. Fritz, R. Grenyer, M.R. Helmus, L.S. Jin, A.O. Mooers, S. Pavoine, A guide to phylogenetic metrics for conservation, community ecology and macroecology Biological Reviews, 92(2), 2017, p. 698-715.
- M. Winter, V. Devictor, O. Schweiger, Phylogenetic diversity and nature conservation: where are we?, Trends in Ecology & Evolution, 28(4), 2013, p. 199-204.
- J. Cavender‐Bares, K.H. Kozak, P.V. Fine, S.W. Kembel, The merging of community ecology and phylogenetic biology, Ecology letters, 12(7), 2009, p. 693-715.
- M.W. Cadotte, J. Cavender-Bares, D. Tilman, T.H. Oakley, Using phylogenetic, functional and trait diversity to understand patterns of plant community productivity, PloS one, 4(5), 2009 : e5695.
- M.W. Cadotte, R. Dinnage, D. Tilman, Phylogenetic diversity promotes ecosystem stability, Ecology, 93(sp8), 2012 : S223-S233.
- S.W Kembel, S.P. Hubbell, The phylogenetic structure of a neotropical forest tree community, Ecology, 87(sp7), 2006 : S86-S99.
- F. Forest, R. Grenyer, M. Rouget, T.J. Davies, R.M. Cowling, D.P. Faith, A. Balmford, J.C. Manning, Ş. Procheş, M. van der Bank, G. Reeves, Preserving the evolutionary potential of floras in biodiversity hotspots, Nature, 445(7129), 2007, p. 757
- F. Mazel, A.O. Mooers, G.V.D. Riva, M.W. Pennell, Conserving phylogenetic diversity can be a poor strategy for conserving functional diversity, Systematic biology, 66(6), 2017, p. 1019-1027.
- F. Mazel, M.W. Pennell, M.W. Cadotte, S. Diaz, G.V. Dalla Riva, R. Grenyer, F. Leprieur, A.O. Mooers, D. Mouillot, C.M. Tucker, W.D. Pearse, Prioritizing phylogenetic diversity captures functional diversity unreliably, Nature communications, 9(1), 2018, p. 2888