Dégradation d'Edman
La dégradation d'Edman, développée par Pehr Edman, est une méthode de séquençage des acides aminés dans un peptide. Dans ce procédé, le groupement amino-terminal est marqué et clivé du peptide sans perturber les liaisons peptidiques entre d'autres groupements d'acides aminés.
Mécanisme
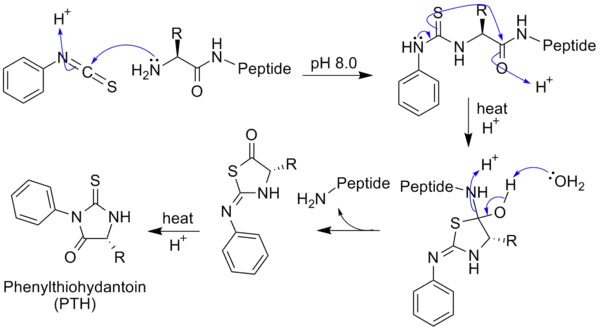
L'isothiocyanate de phényle réagit avec un groupe amino N-terminal non chargé, dans des conditions légèrement alcalines, pour former un dérivé cyclique de phénylthiocarbamide. Ensuite, dans des conditions acides, ce dérivé de l'acide aminé terminal est clivé sous la forme d'un dérivé de thiazolinone. L'acide aminé thiazolinone est ensuite sélectivement extrait dans un solvant organique et traité avec de l'acide pour former le dérivé phénylthiohydantoïne (PTH) - acide aminé plus stable qui peut être identifié en utilisant une chromatographie ou une électrophorèse. Cette procédure peut alors être répétée à nouveau pour identifier l'acide aminé suivant. Un inconvénient majeur de cette technique est que les peptides étant séquencés de cette manière ne peuvent pas avoir plus de 50 à 60 résidus (et en pratique, moins de 30). La longueur du peptide est limitée en raison de la dérivation cyclique n'étant pas toujours achevée. Le problème de dérivation peut être résolu par clivage de peptides de grande taille en peptides plus petits avant de procéder à la réaction. Il est possible de précisément séquencer jusqu'à 30 acides aminés avec des machines modernes capables de plus de 99 % d'efficacité par acide aminé. Un avantage de la dégradation d'Edman est qu'elle n'utilise que 10 - 100 picomoles de peptide pour le procédé de séquençage. La réaction de dégradation d'Edman a été automatisée en 1967 par Edman et Beggs pour accélérer le processus et 100 dispositifs automatisés étaient utilisés dans le monde entier en 1973[1].
Limitations
Comme la dégradation d'Edman se fait à partir de groupement N-terminal de la protéine, elle ne fonctionnera pas si le groupement N-terminal a été chimiquement modifié (par exemple par acétylation ou formation d'acide pyroglutamique). La dégradation d'Edman n'est généralement pas utile pour déterminer les positions des ponts disulfures. Il nécessite également des quantités peptidiques de 1 picomole ou plus pour des résultats discernables.
Analyse couplée
Après une SDS-PAGE, les protéines peuvent être transférées à une membrane "buvard" de polyfluorure de vinylidène (PVDF) pour une analyse ultérieure. La dégradation d'Edman peut être effectuée directement à partir d'une membrande PVDF. Le séquençage des groupements N-terminaux résultant de 5 ou 10 acides aminés peut être suffisant pour identifier une protéine d'intérêt.
Voir aussi
Références
- Niall HD, « Automated Edman degradation: the protein sequenator », Meth. Enzymol., vol. 27, , p. 942–1010 (ISBN 978-0-12-181890-6, PMID 4773306, DOI 10.1016/S0076-6879(73)27039-8)