Hybridation génomique comparative
L'hybridation génomique comparative (en anglais, Comparative Genomic Hybridization ou CGH) est une technique de cytogénétique moléculaire permettant d'analyser les variations du nombre de copies dans l'ADN.
Principe
Dans un organisme diploïde tel que l'humain, chaque segment d'ADN peut être trouvé en duplicat : une copie est présente sur chacun des deux chromosomes d'une paire. Dans certaines pathologies, le nombre de copies peut varier : il peut augmenter par exemple en cas de duplication et diminuer dans le cas de délétion. L'hybridation génomique comparative permet de diagnostiquer ces pathologies et d'identifier les régions chromosomiques impliquées.
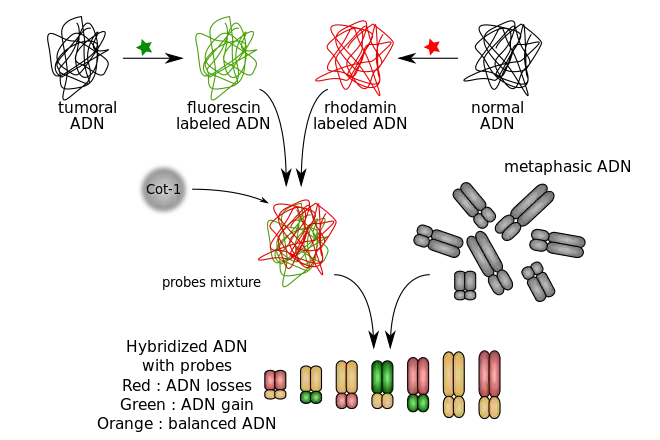
Méthode
- Marquage des sondes
L'ADN d'un patient est extrait de tissu normal (souvent des lymphocytes) et de tissu potentiellement tumoral puis marqué par des fluorochromes. En général, l'ADN tumoral est marqué en vert par la fluorescéine (sous forme de dUTP-FITC) et l'ADN de tissu sain en rouge par la rhodamine (sous forme de dUTP-TR).
Les sondes sont mélangées ensuite avec de l'ADN Cot-1, qui permet de supprimer les séquences répétitives d'ADN, et une polymérase, qui permet de produire des fragments de 0,6 à 3 kb (milliers de paires de bases).
- Hybridation
Le mélange d'ADN marqué est mélangé avec de l'ADN de contrôle en métaphase. L'ADN marqué va alors s'hybrider de façon compétitive avec l'ADN métaphasique :
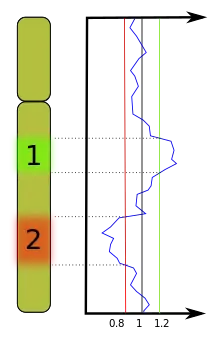
- si le nombre de copies est augmenté, la sonde tumorale s'hybridera préférentiellement et le segment de chromosome apparaitra en vert.
- si le nombre de copies est diminué, la sonde de tissu sain s'hybridera préférentiellement et le segment de chromosome apparaitra en rouge.
- si le nombre de copies n'est pas modifié, les deux sondes s'hybrideront en quantité équivalente et le segment de chromosome apparaitra en orangé.
- Détection
La fluorescence est ensuite détectée par un microscope à épifluorescence et quantifié par analyse d'image quantitative.
Cette technique est capable de détecter des pertes ou des gains du nombre de copie d'ADN au niveau des chromosomes. En 2006, les référentiels indiquaient pour la sensibilité qu'il fallait des régions de 5 à 10 Mb (millions de paires de bases) pour détecter la perte d'une seule copie. En revanche, la détection d'une amplification était possible à partir de 1 Mb.
Considérations techniques et limitations
- Cette technique détecte seulement les modifications chromosomiques non équilibrées. Des aberrations chromosomiques structurales telle que les translocations réciproques ou les inversions ne seront pas détectées.
- Lors de l'analyse quantitative, le ratio de fluorescence (sain / tumoral) le plus fréquent est utilisé comme référence. De ce fait, cette technique ne donne pas d'informations concernant la ploïdie : des tétraploïdes ne présentant pas de réarrangements déséquilibrés apparaitront normaux.
- Les puces d'hybridation génomique comparative utilisent des sondes fixées sur puce et permettent d'augmenter la sensibilité, la gamme dynamique (la longueur des segments analysables) et le débit. Elles permettent également de cartographier les régions chromosomiques impliquées dans les pathologies analysées.
Applications
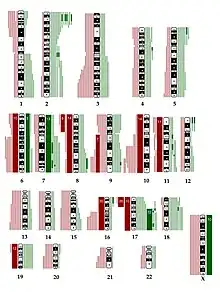
- En recherche et en clinique, pour l'étude et le diagnostic des cancers, de malformations congénitales, des retards mentaux, etc. Voir en particulier les articles sur les modifications chromosomiques pour des exemples de pathologies présentant des modifications dans la structure des chromosomes : Duplication (génétique), Cassure chromosomique.
Voir aussi
- Puce d'hybridation génomique comparative (CGH Array), une technique équivalente sur puce.
Liens externes
- L'hybridation génomique comparative sur microréseau d'ADN en pathologie chromosomique constitutionnelle (article de Pathologie Biologie, Volume 55, Issue 1, , Pages 13-18)
- Virtual Grand Rounds: "Differentiating Microarray Technologies and Related Clinical Implications" by Arthur Beaudet, MD
- Progenetix database : Collection de données de CGH provenant de publications (16000 cas, ).
- « CGH article tracker »(Archive.org • Wikiwix • Archive.is • Google • Que faire ?) : Liste de publications contenant des données originales de CGH.
- NCBI's Cancer Chromosomes : Base de données du NCBI regroupant des données de génomique et de cytogénétique.
- Une bibliographie sur les variations du nombre de copies de l'ADN