Distance génétique (génétique formelle)
La distance génétique désigne la distance virtuelle entre deux locus situés sur un même chromosome. Elle s'exprime le plus souvent en centimorgans.
Historique
À partir de la découverte des gènes liés et de la recombinaison par enjambement, l'un des étudiants de Morgan, Alfred Sturtevant, a mis au point une méthode permettant d'établir une carte génétique, c'est-à-dire une liste ordonnée des locus tout le long d'un chromosome.
Alfred Sturtevant commença sa carrière en génétique en 1911 comme étudiant de deuxième cycle dans le laboratoire du grand généticien Thomas Hunt Morgan. Morgan confia à Sturtevant la tâche d'expliquer certaines données. Celles-ci concernaient différentes combinaisons alléliques qui apparaissaient dans des croisements de gènes présentant une transmission liée au chromosome X. En une soirée, Sturtevant exploita toutes les données et ce faisant, développa la méthode analytique permettant de cartographier les gènes sur les chromosomes, qui est toujours en vigueur aujourd'hui.
En se remémorant l'évènement plus tard, il dit : « À la fin de l'année 1911, en parlant avec Morgan, je réalisai soudain que les variations dans l'intensité de la liaison génétique (association entre les allèles de différents gènes) déjà attribuées par Morgan à la séparation spatiale des gènes, offraient la possibilité de déterminer des séquences linéaires des gènes sur un chromosome. Je rentrai chez moi et passai la plus grande partie de la nuit à élaborer la première carte chromosomique ».
L'analyse fut publiée en 1913. L'introduction disait : « Le parallèle entre le comportement des chromosomes en réduction, et celui des facteurs de Mendel lors de la ségrégation a été souligné pour la première fois par Walter Sutton (1902), bien que plus tôt dans la même année Theodor Boveri (1902) ait fait référence à une association possible ».
L'article se poursuivait par la cartographie de six gènes liés à l'X, faisant appel à la logique suivante : « La proportion de crossing-over pourrait être utilisée comme un indicateur de la distance séparant n'importe quelle paire de facteurs. Puis, en déterminant les distances entre A et B et entre B et C, on devait pouvoir prédire AC. Car, si les proportions de crossing-over représentent réellement la distance, AC doit être approximativement égale à AB plus BC ou à AB moins BC, et non pas à une valeur intermédiaire ».
La carte du chromosome X de Sturtevant (la première carte génétique) était la suivante. Le zéro correspondait arbitrairement à une extrémité déterminée de la carte :
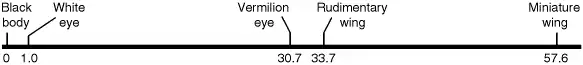
Remarquez que (par exemple) la distance génétique entre le gène de l'aile rudimentaire et celui de l'aile miniature est de 57.6 moins 33.7 soit 23.9 unités génétiques.
La conclusion de Sturtevant était la suivante : « les résultats apportent un nouvel argument en faveur de la conception chromosomique de l'hérédité, car ils indiquent fortement que les facteurs étudiés sont arrangés de façon linéaire, au moins du point de vue mathématique ».
Définition
La distance génétique est la distance qui sépare deux gènes sur un même chromosome. Sturtevant a exprimé la distance entre les gènes en unités cartographiques. De nos jours, on emploie plutôt le terme Centimorgan(cM) en l'honneur de Morgan.
Calcul de la distance génétique
La distance génétique se calcule à partir de la fréquence de recombinaison () , égale à :
avec = nombre de produits recombinés et = nombre total de produits
L'unité de distance génétique est le (cM). Elle correspond à un segment de chromatide sur lequel la probabilité qu'un crossing-over s'effectue est de 1 %. Le morgan est donc l'intervalle où se produit en moyenne un événement de recombinaison par crossing-over lors de la méiose.
La distance génétique augmente avec la taille de l'intervalle séparant deux marqueurs : lorsque la distance physique grandit, la probabilité qu'il se produise un crossing-over grandit également. Néanmoins, la relation entre la fréquence des remaniements consécutifs aux crossing-over et la fréquence de recombinaison n'est pas toujours linéaire. Elle dépend de la position relative des marqueurs en jeu.
On peut arbitrairement distinguer trois situations :
- Les deux marqueurs sont proches : sur le segment chromosomique compris entre ces deux marqueurs, il ne se produit jamais plus qu'un crossing-over par méiose; la fréquence de recombinaison reflète alors directement la fréquence des remaniements consécutifs aux crossing-over. Tous les produits de la méiose qui ont hérité d'une chromatide qui a été touchée par un crossing-over(et seulement ceux-là) sont recombinés. Donc dans ce cas, la distance génétique est égale au pourcentage de produits recombinés.
- Les deux marqueurs sont plus éloignés que dans le cas précédent : sur le segment chromosomique compris entre ces deux marqueurs, il peut y avoir soit aucun, soit un seul, soit plusieurs crossing-over par méiose. La fréquence de recombinaisons sous-estime alors la fréquence de remaniements consécutifs aux crossing-over multiples. En effet, lorsque ces derniers affectent les deux chromatides, ils restaurent des associations parentales et ne peuvent donc pas être décelés; quand ils touchent trois des quatre chromatides, seulement deux chromatides donnent des produits recombinés, comme si seulement un crossing-over s'était produit. Donc dans ce cas, il est nécessaire pour calculer correctement la distance génétique entre les deux marqueurs d'utiliser des méthodes permettant d'estimer la fréquence des crossing-over multiples.
- Les deux marqueurs sont situés à une distance telle qu'il se produit toujours au moins un crossing-over entre eux. À la méiose, les simples crossing-over produisent deux produits parentaux et deux produits recombinés. De même, les crossing-over multiples, affectant de manière aléatoire les quatre chromatides, produisent globalement autant de produits parentaux que de produits recombinés. En conséquence, la fréquence totale des produits recombinés est égale à 0.5. Donc dans ce cas, les deux marqueurs sont généralement indépendants. Il est impossible de calculer une distance génétique.
En résumé, les distances génétiques ne peuvent être calculées de manière fiable que pour les fréquences de recombinaison faibles, dans des intervalles assez petits pour que les crossing-over multiples soient absents. Si la fréquence de produits recombinés est supérieure à une certaine valeur(de l'ordre de 0,1), il faut alors rechercher des marqueurs intermédiaires. Si on ne dispose pas de tels marqueurs, il faut utiliser des méthodes de calcul de distances génétiques permettant d'estimer la fraction de crossing-over multiples.
Application
La distance génétique du génome (mesurée en centimorgans) consiste donc à localiser sur les chromosomes la région en cause dans une maladie génétique. Une telle distance génétique a permis de placer et organiser une multitude de marqueurs polymorphes sur les différents chromosomes.
Ces marqueurs polymorphes, utilisés dans les études génétiques sont :
- des micro ou mini-satellites;
- des SNP (single nucleotide polymorphism), qui, comme leur nom l'indique concernent un seul nucléotide en une position donnée du génome, dont la nature est variable entre les individus d'une même espèce, sans pourtant avoir la moindre conséquence sur l'expression de gènes.
Aujourd'hui, ces différentes distances sont unifiées pour plusieurs organismes et l'on peut voir dans les banques de données la localisation des gènes, marqueurs et maladies sur les différents chromosomes d'une espèce.
- Les Banques de données
Un site unifié du National Institute of Health (NIH) américain regroupe l'ensemble des informations et des logiciels permettant l'analyse des génomes des êtres vivants.
Le site ncbi (National Center of Biotechnology Information) public mis en place en 1988 est consultable par tous sur internet[1].
Notes et références
Bibliographie
- Griffiths, Gelbart, Miller, Lewontin, Analyse Génétique Moderne, 1re édition, chapitre 5, De Boeck Université, 2001
- Rossignol, Berger, Deutsch, Fellous, Lamour-Isnard, Ozier-Kalogeropoulos, Picard, de Vienne, Génétique, chapitre 5, Paris, Dunod, 2000
- Étienne, Clauser, Housset, Roingeard, Biochimie génétique. Biologie moléculaire, 9e édition, chapitre 8, , Masson, 2006
- Campbell et Reece, Biologie, 7e édition, chapitre 13, Pearson Education, 2007
- Alberts, Bray, Hopkin, Johnson, Lewis, Raff, Roberts, Walter, L'Essentiel de la Biologie Cellulaire, 2e édition, chapitre 20, Flammarion, 2005
- Watson, Baker, Bell, Gann, Levine, Losick, Biologie moléculaire du gène, 6e édition, partie 1.5, Pearson Education, 2005



